4.1 scRNA seq 概要
什么是单细胞 RNA 测序?¶
单细胞 RNA 测序是一种用于研究单个细胞内基因表达模式的方法。通过这种方法,可以分析细胞群体内每个单细胞的基因表达情况。它常用于细胞鉴定、细胞生命周期研究以及细胞发育等方面。
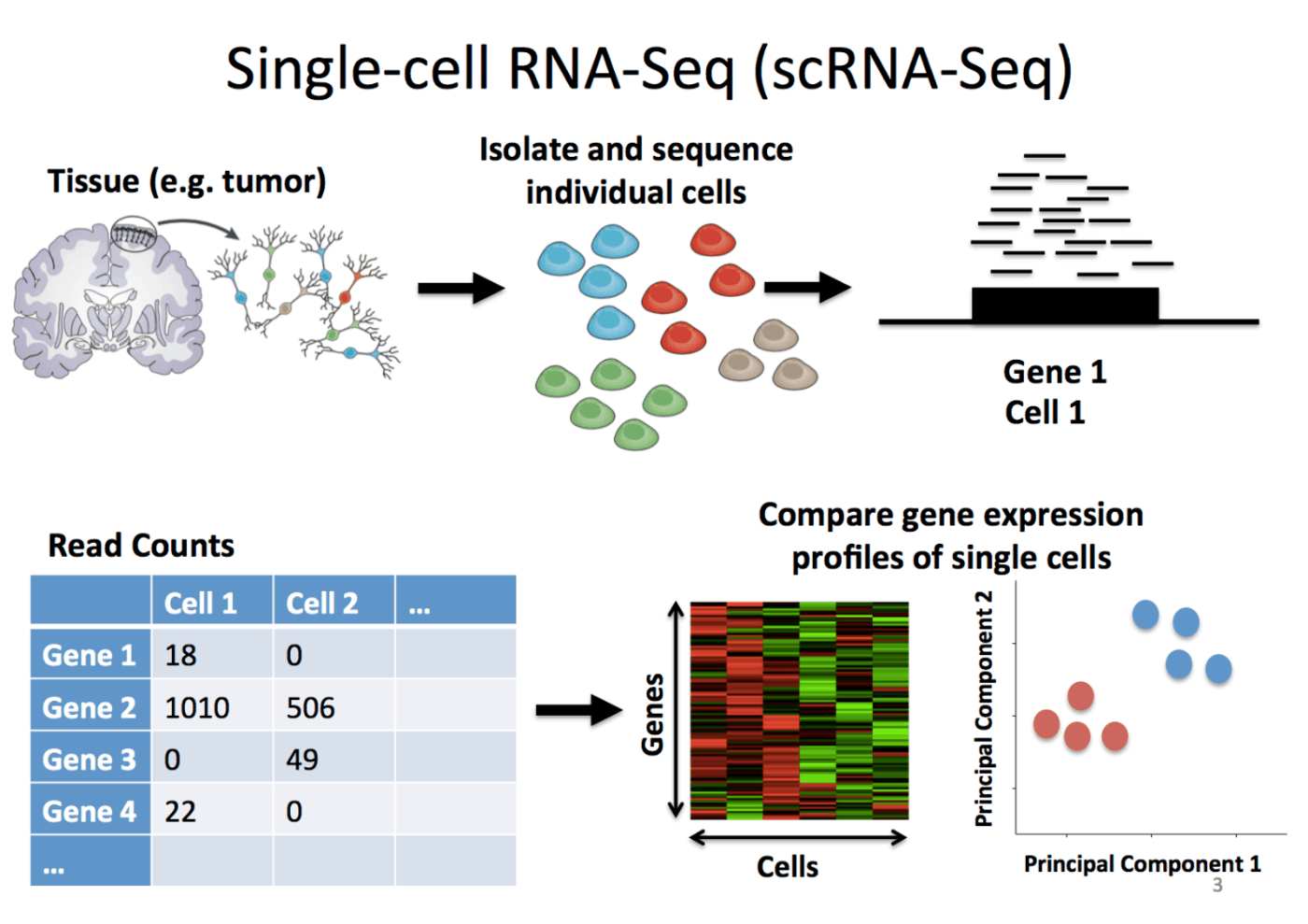
关于单细胞 RNA 测序 (scRNA-seq)
单细胞 RNA 测序(single-cell RNA sequencing)是一种用于研究单个细胞内基因表达模式的方法。通过这种方法,可以分析细胞群体内每个单细胞的基因表达情况。它常用于细胞鉴定、细胞生命周期研究以及细胞发育等方面。
scRNA-seq 数据库¶
scRNA-seq 数据已被注册到多个公共数据库中。访问这些数据库可以找到 scRNA-seq 数据。
- NCBI GEO: 由 NCBI 运营,提供 bulk RNA-seq 和 scRNA-seq 数据。
- Single Cell Expression Atlas (SCEA): 由 EMBL-EBI 运营的 scRNA-seq 数据库。
- Single Cell Portal (SCP): 由 MIT 和哈佛大学的 Broad Institute 运营的 scRNA-seq 数据库,截至 2023 年 1 月底,包含 507 项研究和 29,614,655 个细胞的表达数据。
- The Human Cell Atlas (HCA): 收录人类来源细胞的 scRNA-seq 数据。
- The Mouse Cell Atlas (MCA): 收录小鼠来源细胞的 scRNA-seq 数据。
- SCPortalen: 由理研运营,主要收录人类和小鼠的 scRNA-seq 数据。
10x Genomics 格式¶
10x Genomics 是一家提供单细胞 RNA 测序技术的公司,可以低成本分析大量细胞,因而被广泛应用。10x Genomics 的数据使用独特的格式,为了解析这种格式的数据,10x Genomics 提供了 cellranger 这个特殊的库,但也可以用 Seurat 进行解析。
10x Genomics 的独特格式包含以下文件:
$ tree filtered_feature_bc_matrix
filtered_feature_bc_matrix
├── barcodes.tsv.gz
├── features.tsv.gz (或genes.tsv.gz)
└── matrix.mtx.gz
选择用于 scRNA-seq 解析的语言¶
单细胞 RNA-seq 解析有 Python 和 R 两个包可用。选择哪一个取决于各自的优缺点,以及用户的需求和经验。
Python:Scanpy¶
- 速度: Scanpy 利用了用于快速计算的库(如 NumPy 和 SciPy)。
- 灵活性: 易于与其他 Python 库和工具集成,便于自定义分析和机器学习实现。
- 注释: Scanpy 具有自动为细胞簇注释的工具。
主要特点: - 支持从常规 scRNA-seq 预处理到降维、聚类和数据可视化的完整工作流程。 - 易于与 Python 的可视化工具(如 Matplotlib 和 Plotly)集成。 - 与其他基于 Python 的 scRNA-seq 工具兼容。
R:Seurat¶
- 全面性: Seurat 为 scRNA-seq 数据解析提供了全面的工具集。
- 社区支持: R 的生物信息学社区非常活跃,有许多额外的资源和教程可用。
- 集成: 易于与其他 R 的生物信息学工具(特别是 Bioconductor 项目)集成。
主要特点: - 提供丰富的功能用于聚类、降维和细胞注释等 scRNA-seq 解析。 - 利用 R 的 ggplot2 进行强大的数据可视化。 - 提供多种数据整合和比较分析工具集。
总的来说,选择 Python 还是 R 取决于用户的习惯和需求。如果日常使用 Python 或需要实现自定义编码和机器学习,可以选择 Scanpy。如果已经熟悉 R 的生物信息学生态系统,或者需要丰富的社区支持和教程,可以选择 Seurat。两者都是非常优秀且广泛使用的工具。