4.4 使用 Seurat 进行数据分析的一般顺序
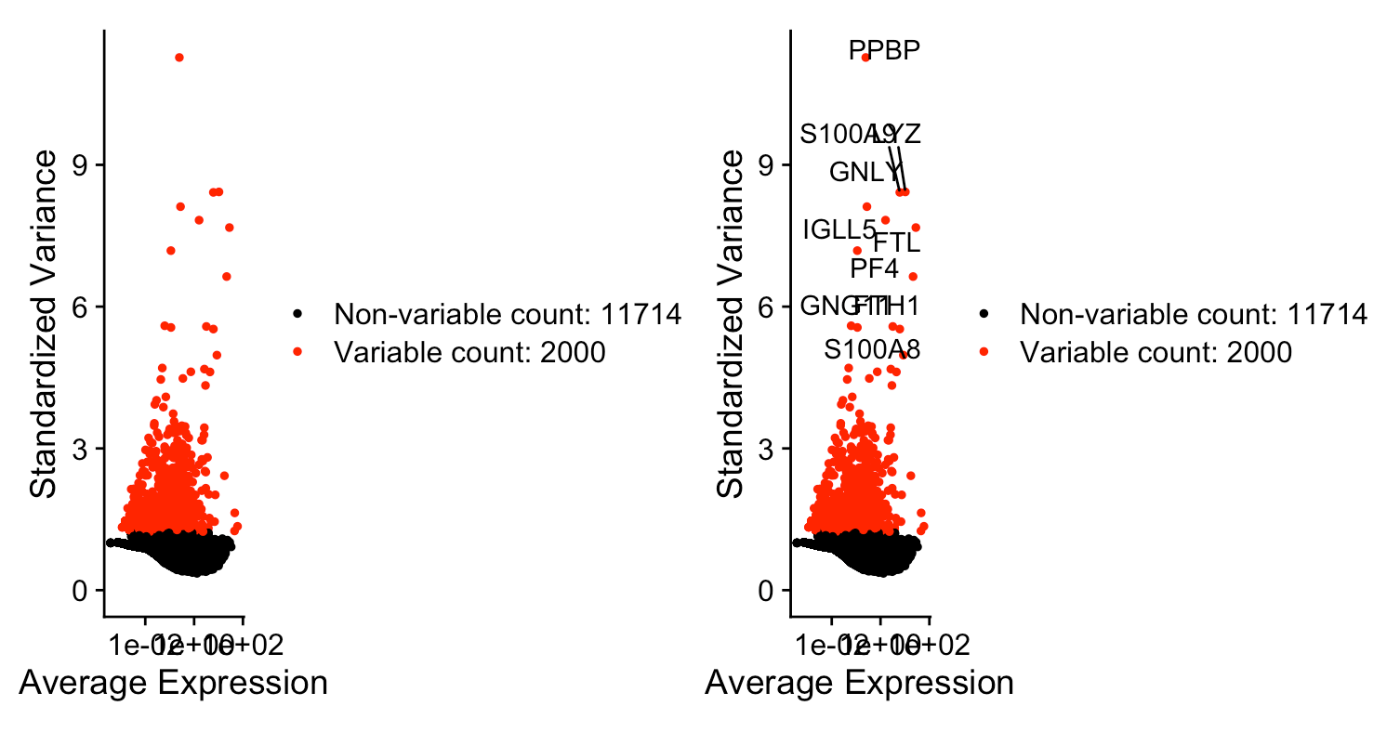
提取和可视化变异基因
计算在数据集中细胞间变异性较大的子集,其中某些细胞表现出高表达,而其他细胞表现出低表达。默认情况下,每个数据集返回 2000 个变异基因。
# 使用FindVariableFeatures函数准备数据
pbmc <- FindVariableFeatures(pbmc, selection.method = "vst", nfeatures = 2000)
# 获取前10个变异基因的名称
top10 <- head(VariableFeatures(pbmc), 10)
# 绘制图形
plot1 <- VariableFeaturePlot(pbmc)
plot2 <- LabelPoints(plot = plot1, points = top10, repel = TRUE)
plot1 + plot2
降维和细胞聚类
主成分分析 (PCA)¶
在进行 PCA 等降维方法之前,需要通过线性变换进行预处理。请执行以下命令:
all.genes <- rownames(pbmc)
pbmc <- ScaleData(pbmc, features = all.genes)
对缩放后的数据执行 PCA 的命令如下:
pbmc <- RunPCA(pbmc, features = VariableFeatures(object = pbmc))
成功后,你会看到类似如下的输出:
PC_ 1
Positive:
CST3, TYROBP, LST1, AIF1, FTL, FTH1, LYZ, FCN1, S100A9, TYMP
FCER1G, CFD, LGALS1, S100A8, CTSS, LGALS2, SERPINA1, IFITM3, SPI1, CFP
PSAP, IFI30, SAT1, COTL1, S100A11, NPC2, GRN, LGALS3, GSTP1, PYCARD
Negative:
MALAT1, LTB, IL32, IL7R, CD2, B2M, ACAP1, CD27, STK17A, CTSW
CD247, GIMAP5, AQP3, CCL5, SELL, TRAF3IP3, GZMA, MAL, CST7, ITM2A
MYC, GIMAP7, HOPX, BEX2, LDLRAP1, GZMK, ETS1, ZAP70, TNFAIP8, RIC3
PC_ 2
Positive:
CD79A, MS4A1, TCL1A, HLA-DQA1, HLA-DQB1, HLA-DRA, LINC00926, CD79B, HLA-DRB1, CD74
HLA-DMA, HLA-DPB1, HLA-DQA2, CD37, HLA-DRB5, HLA-DMB, HLA-DPA1, FCRLA, HVCN1, LTB
BLNK, P2RX5, IGLL5, IRF8, SWAP70, ARHGAP24, FCGR2B, SMIM14, PPP1R14A, C16orf74
Negative:
NKG7, PRF1, CST7, GZMB, GZMA, FGFBP2, CTSW, GNLY, B2M, SPON2
CCL4, GZMH, FCGR3A, CCL5, CD247, XCL2, CLIC3, AKR1C3, SRGN, HOPX
TTC38, APMAP, CTSC, S100A4, IGFBP7, ANXA1, ID2, IL32, XCL1, RHOC
PC_ 3
Positive:
(省略)
为了更清晰地查看,可以使用以下命令限制为查看前 5 个成分:
print(pbmc[["pca"]], dims = 1:5, nfeatures = 5)
如果成功,你会看到类似如下的输出,显示了 PC_1 到 PC_5:
> print(pbmc[["pca"]], dims = 1:5, nfeatures = 5)
PC_ 1
Positive: FTL, FTH1, COTL1, CST3, OAZ1
Negative: MALAT1, IL32, LTB, CCL5, CTSW
PC_ 2
Positive: FTL, TYROBP, S100A8, S100A9, FCN1
Negative: ACTG1, STMN1, TUBA1B, TYMS, ZWINT
PC_ 3
Positive: CD74, HLA-DRA, HLA-DPB1, HLA-DQB1, HLA-DQA1
Negative: PPBP, GNG11, SPARC, PF4, AP001189.4
PC_ 4
Positive: CD74, HLA-DQB1, HLA-DQA1, HLA-DRA, HLA-DQA2
Negative: NKG7, GZMA, GNLY, PRF1, FGFBP2
PC_ 5
Positive: ZWINT, KIAA0101, RRM2, HMGB2, AQP3
Negative: NKG7, CD74, HLA-DQA1, GNLY, SPON2
可视化 PCA 成分¶
VizDimLoadings¶
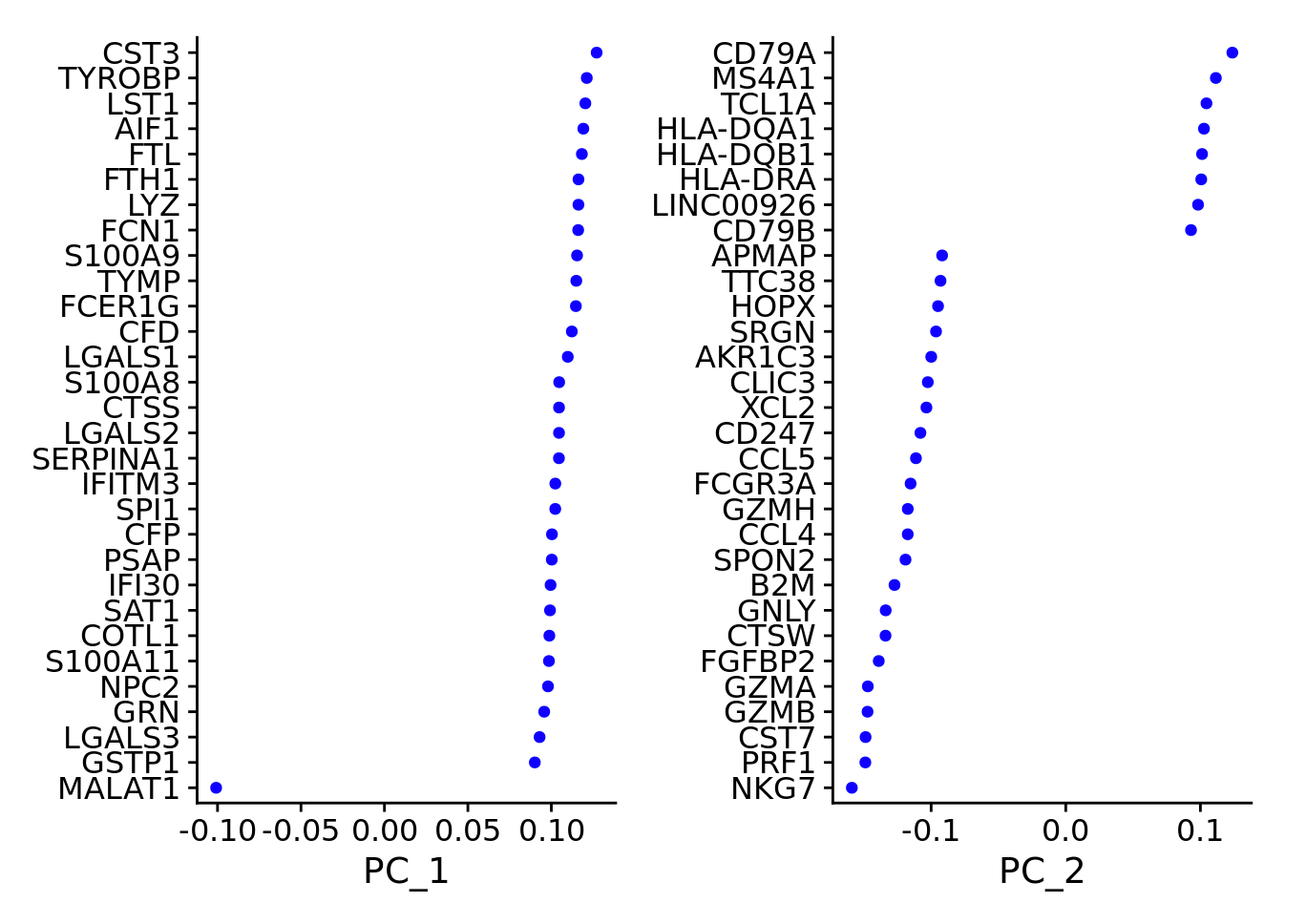
首先,查看哪些基因对各成分的贡献最大。请执行以下命令:
VizDimLoadings(pbmc, dims = 1:2, reduction = "pca")
这个命令将显示贡献率高的基因。
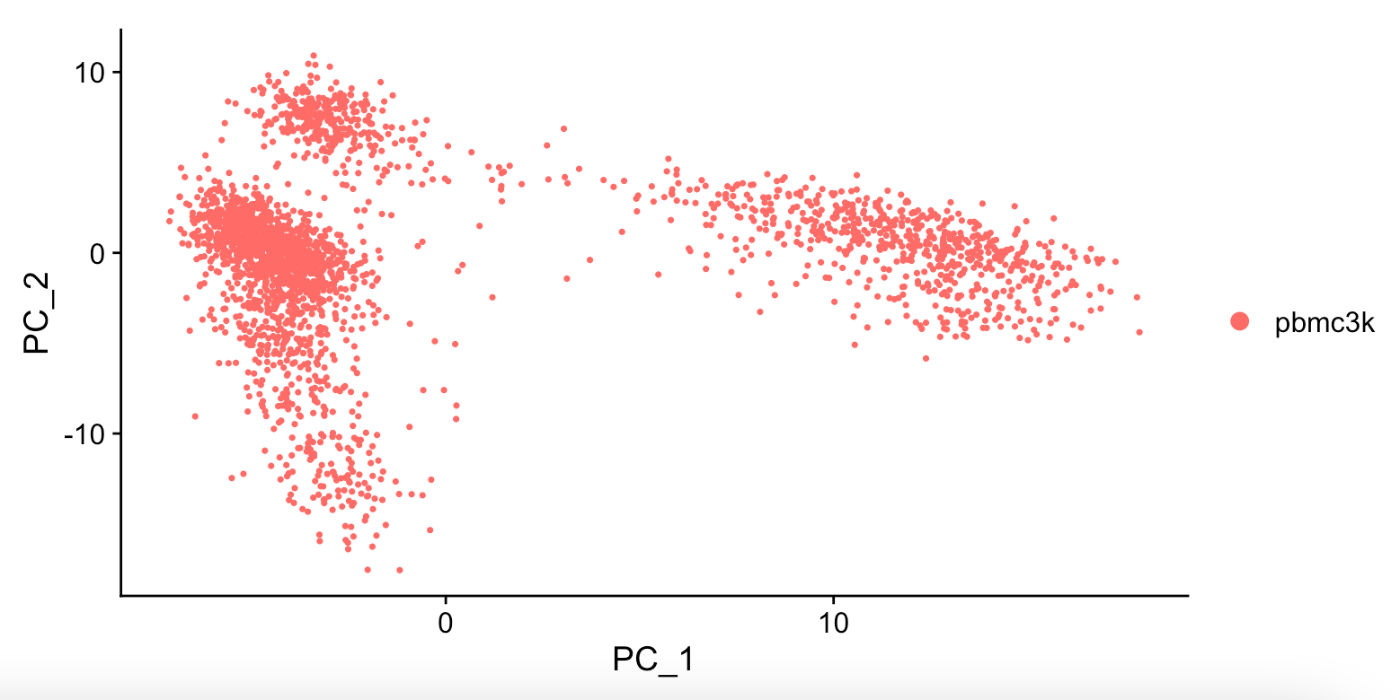
DimPlot¶
使用常规的 PC_1 和 PC_2 的散点图进行绘图可以通过 DimPlot 函数来完成。
DimPlot(pbmc, reduction = "pca")
输出结果如下。
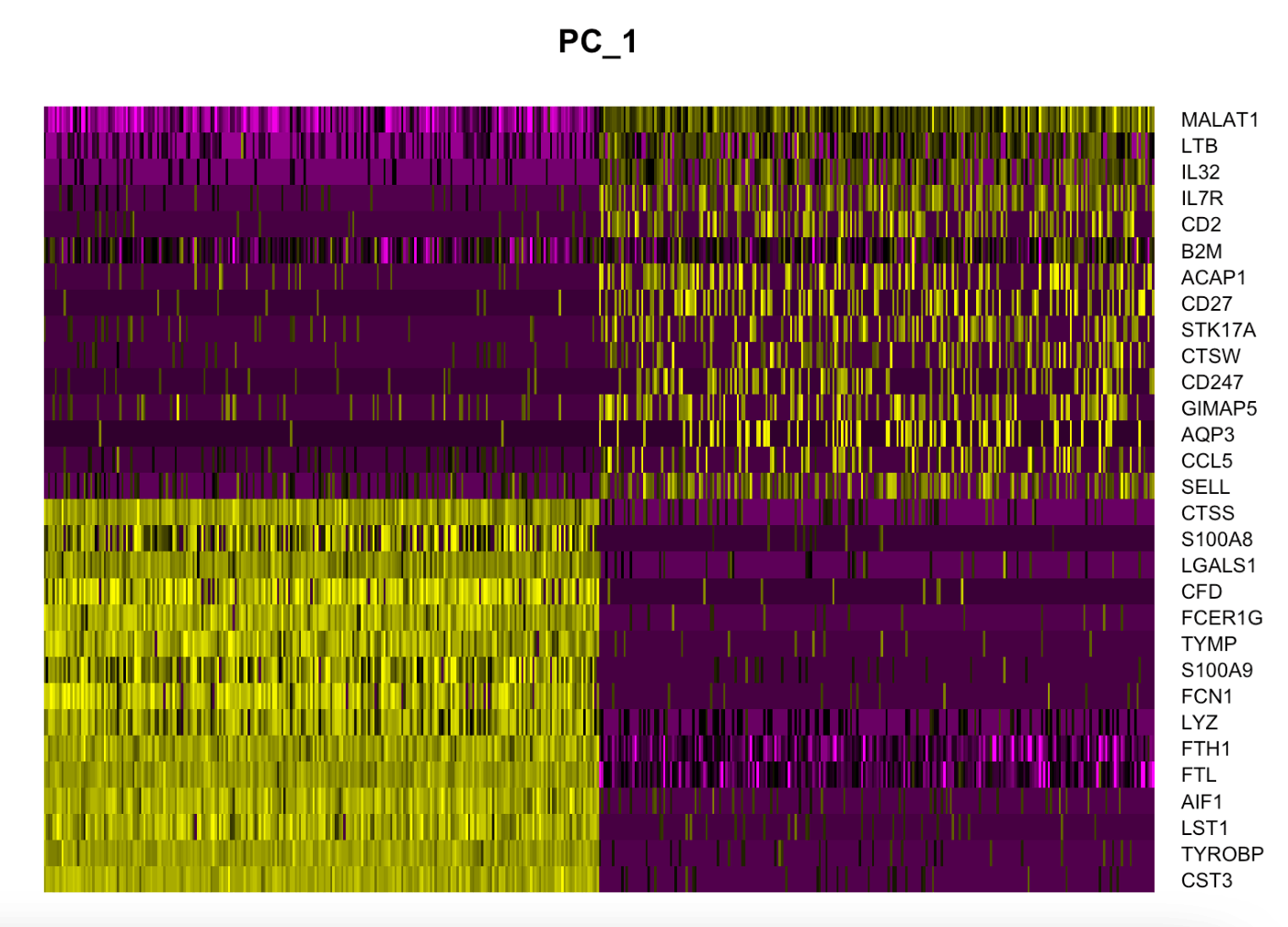
DimHeatmap¶
通过 DimHeatmap 函数,可以以热图的形式对主成分进行调查。可以通过 dims 参数控制要显示的主成分。以下是显示 PC1 的代码。
DimHeatmap(pbmc, dims = 1, cells = 500, balanced = TRUE)
确定数据集的“维度”
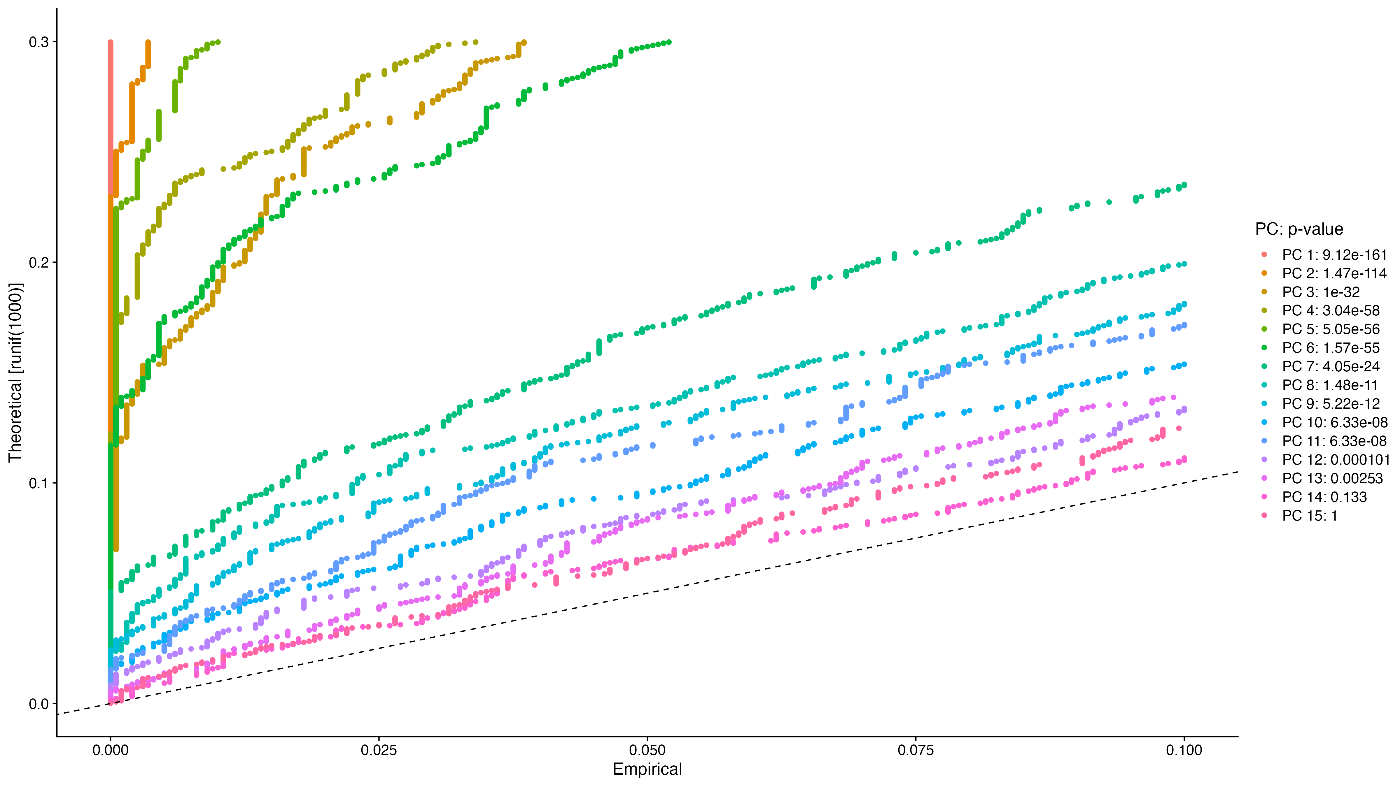
JackStrawPlot¶
通过 JackStrawPlot 可以计算并可视化每个主成分的 p 值。
注意:此代码执行可能需要一些时间。
pbmc <- JackStraw(pbmc, num.replicate = 100)
pbmc <- ScoreJackStraw(pbmc, dims = 1:20)
JackStrawPlot(pbmc, dims = 1:20)
上述代码将计算和可视化前 20 个主成分的 p 值。
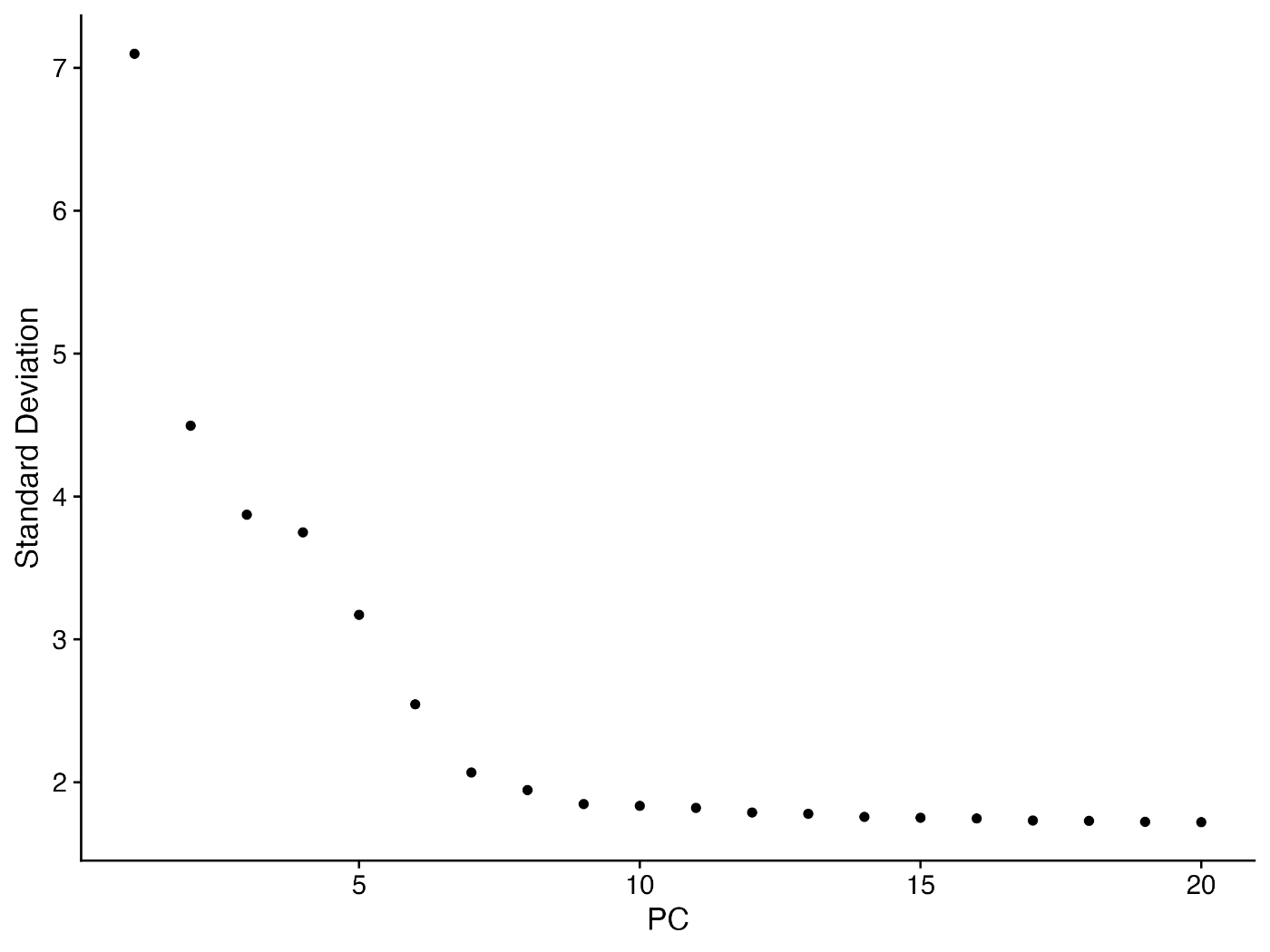
ElbowPlot¶
ElbowPlot 可以用来对各个主成分的贡献比例进行排序,非常方便用于可视化各个主成分的贡献程度。
ElbowPlot(pbmc)
输出结果如下。可以观察到在 PC9-10 附近有一个“肘部”,表明大部分真实信号可以由前 10 个主成分解释。
非线性方法降维
UMAP (Uniform Manifold Approximation and Projection)¶
接下来进行细胞的聚类。对于 scRNA-seq 分析,许多人会想到使用 UMAP/tSNE 进行非线性降维。请执行以下命令进行 UMAP 降维。
# 进行细胞的聚类
pbmc <- FindNeighbors(pbmc, dims = 1:10)
pbmc <- FindClusters(pbmc, resolution = 0.5)
# 执行UMAP
pbmc <- RunUMAP(pbmc, dims = 1:10)
各个函数的详细解释如下。虽然内容较为复杂,但只需理解这是在优化模块并进行聚类即可。
FindNeighbors 函数¶
基于 PCA 空间中的欧氏距离构建 KNN 图,并根据在局部邻域中的共享重叠(Jaccard 相似度)细化任意两个细胞之间的边权重。输入为数据集的预定义维度(前 10 个 PC)。
FindClusters 函数¶
为了进行细胞聚类,应用 Louvain 算法(默认)或 SLM 等模块优化技术,以优化标准模块函数为目标,反复对细胞进行分组。包含设置下游聚类“粒度”的 resolution 参数,值越大,聚类数越多。
可视化 UMAP 结果¶
使用 DimPlot 函数可视化 UMAP 的结果。
DimPlot(pbmc, reduction = "umap")
运行上述代码后,你将看到 UMAP 降维后的可视化结果。
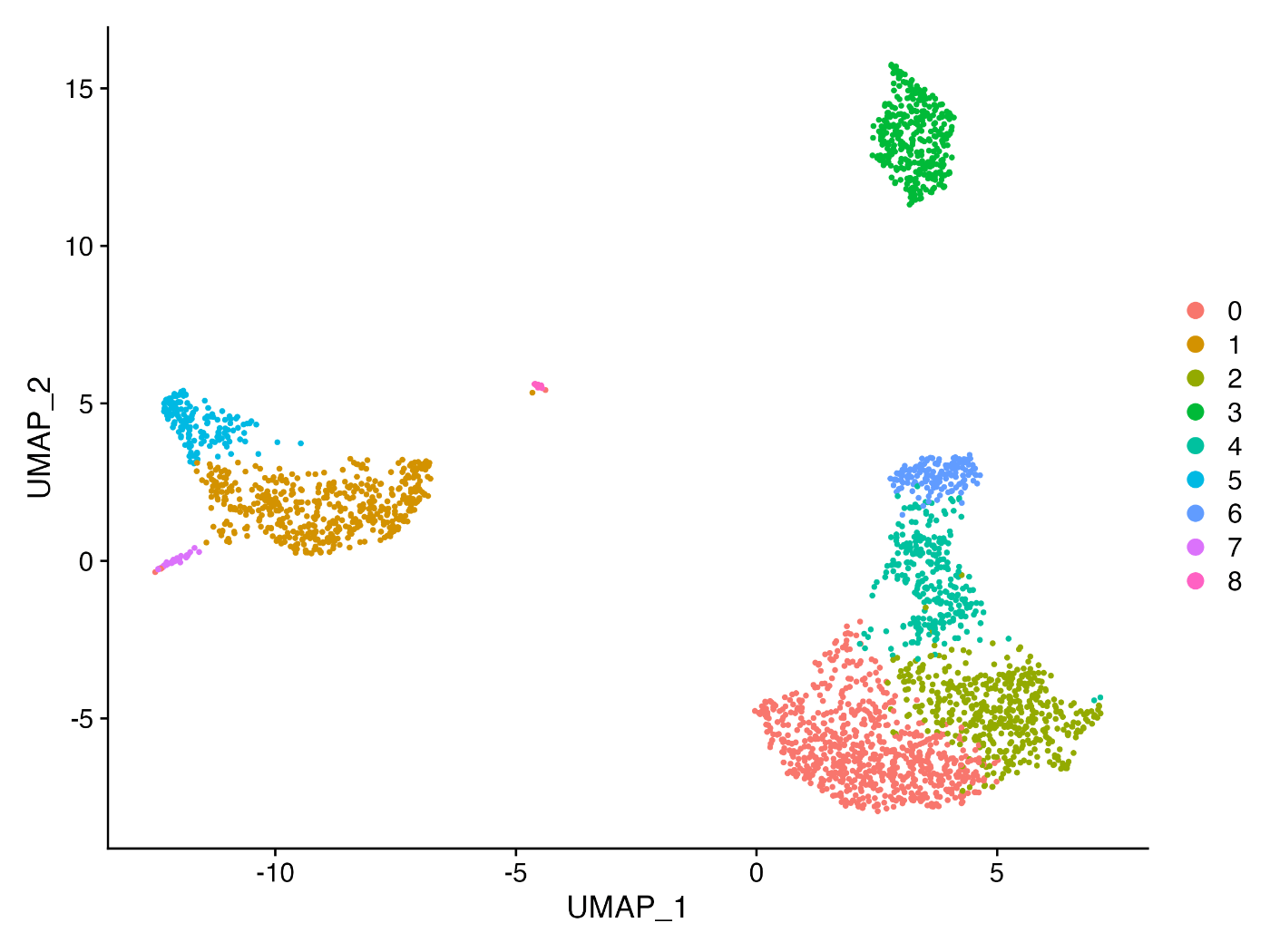
成功进行 UMAP 分析!
到此为止,您已经掌握了使用 Seurat 进行 scRNA-seq 分析的基础知识。教程中还介绍了更多的高级分析内容,感兴趣的朋友可以继续挑战。
如果按照命令操作仍出现错误¶
如果按照命令操作仍出现错误,可能是由于 pbmc 中的值错误。遇到错误时不要慌张,请按以下步骤重新操作:
- 质量检查(QC)
- 规范化
- 提取变异基因
- 降维和聚类
确保按顺序输入命令,这样可以避免错误。