5.1 如何查找单细胞 RNA seq 数据
从这里开始,我们将从公共数据中查找 scRNA-seq 数据,并进行质量检查分析。在质量检查中,我们将使用 Seurat。因此,建议您阅读这篇文章,以便对 Rstudio 和 Seurat 有一些预先的了解。
【scRNA-seq】使用 Seurat 开始 scRNA-seq 分析的方法(前篇)【Seurat】
查找 scRNA-seq 数据¶
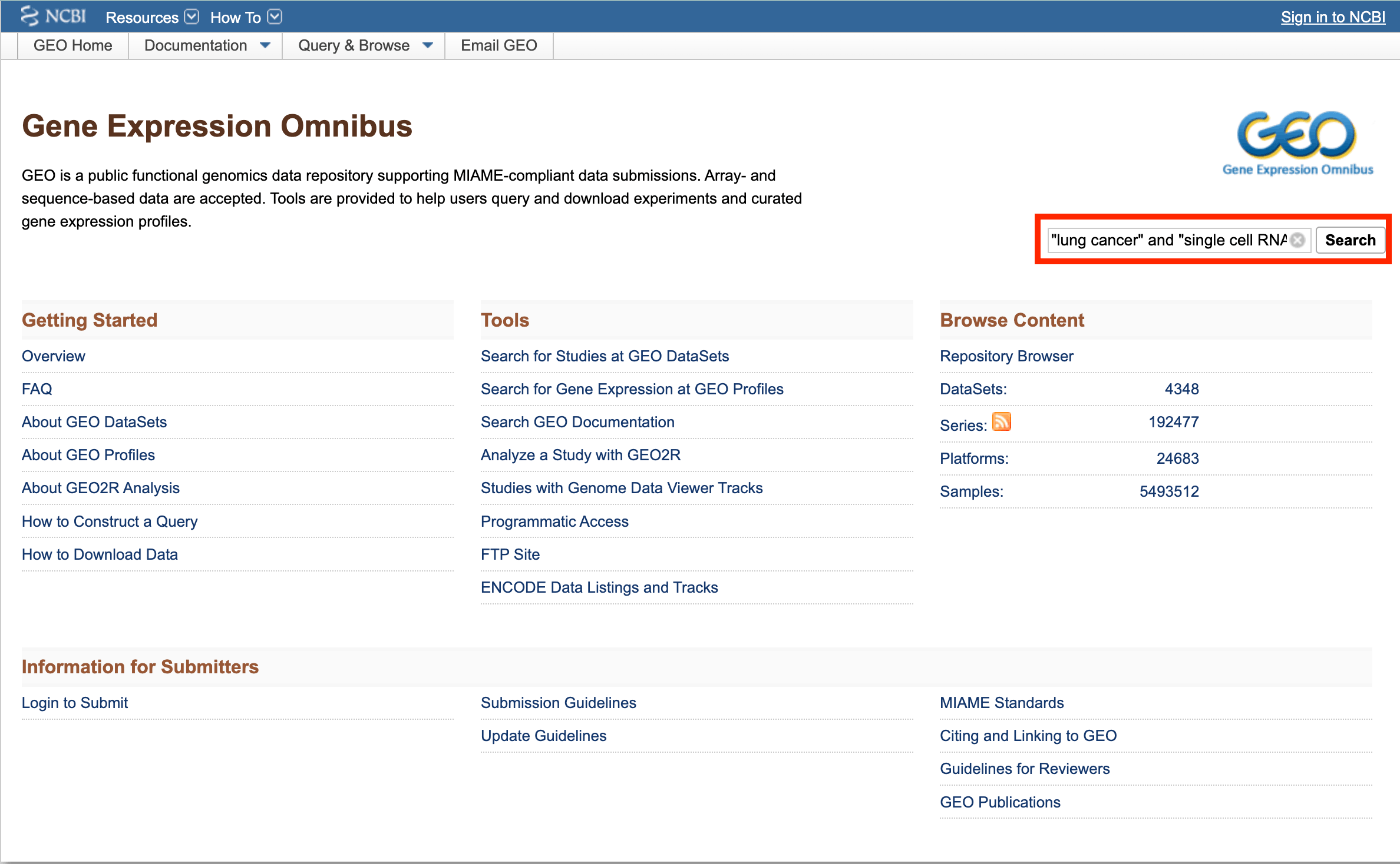
我们将从 NCBI GEO 数据库中查找 scRNA-seq 数据。请访问以下网址:
在右上角的搜索框中输入搜索词。搜索词设置如下:
"[想要搜索的词]" and "single cell RNA-seq"
这次,我们将搜索词设置为“lung cancer” and “single cell RNA-seq”。
开始您的搜索吧!
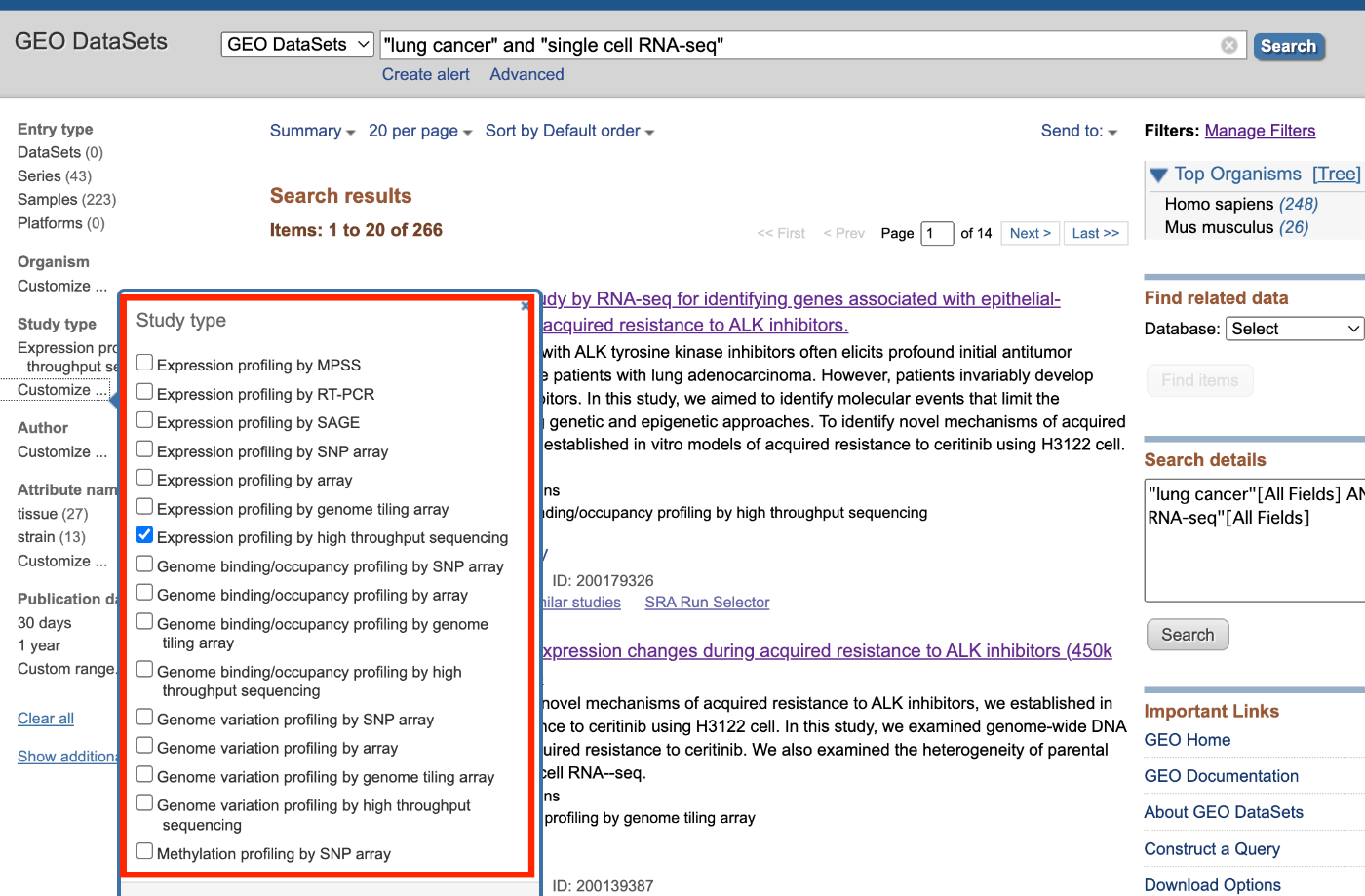
点击左侧边栏的 " 自定义 ",然后勾选 " 通过高通量测序进行表达谱分析 "。
选中后,显示 "Expression profiling by high througput sequencing"(通过高通量测序进行表达谱分析),点击它。
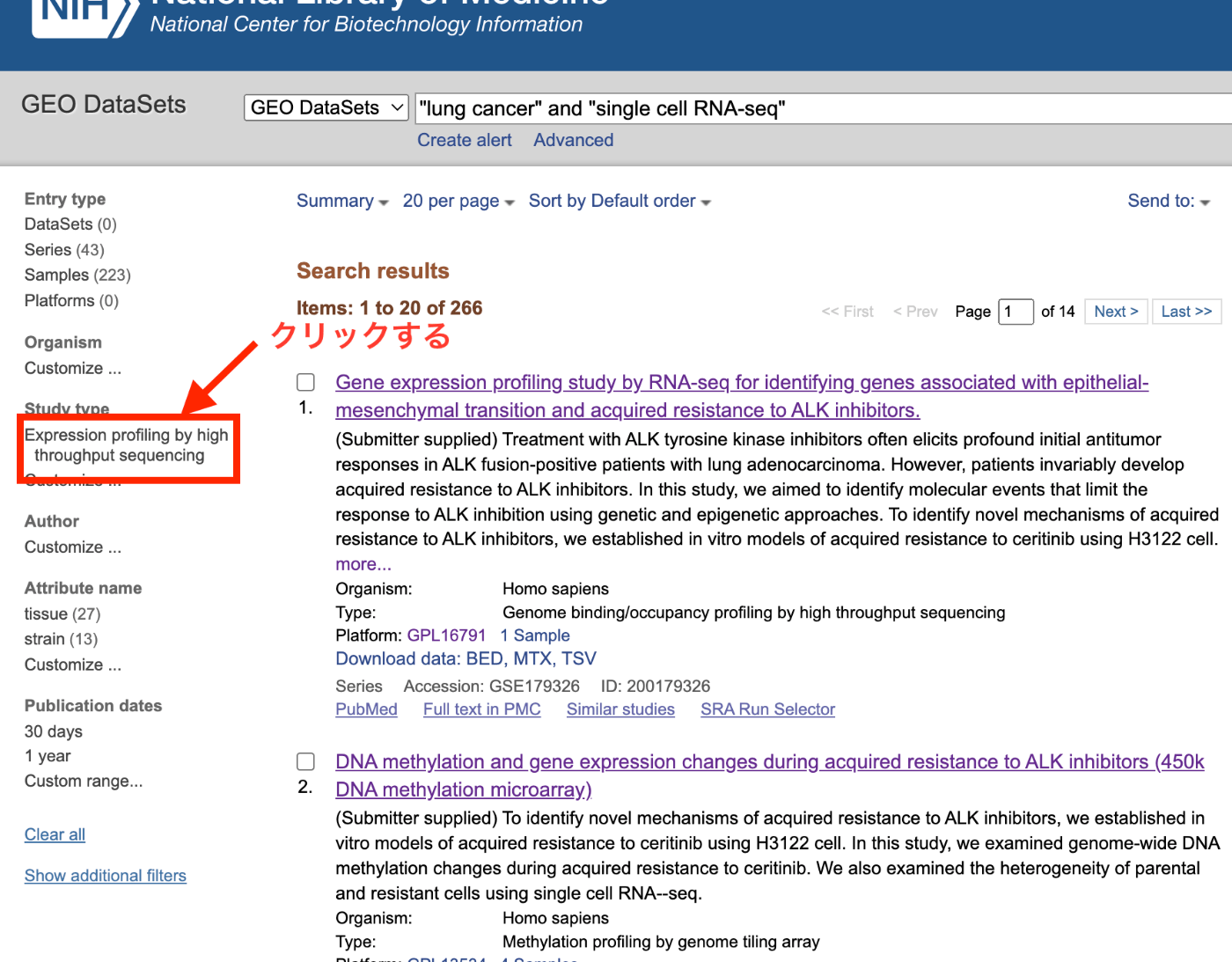
准备工作至此完成。接下来,寻找可以进行 scRNA-seq 分析的样本。请在顶部尝试 ALK 抑制剂获得性抗性过程中的 DNA 甲基化和基因表达变化(scRNA-seq)。
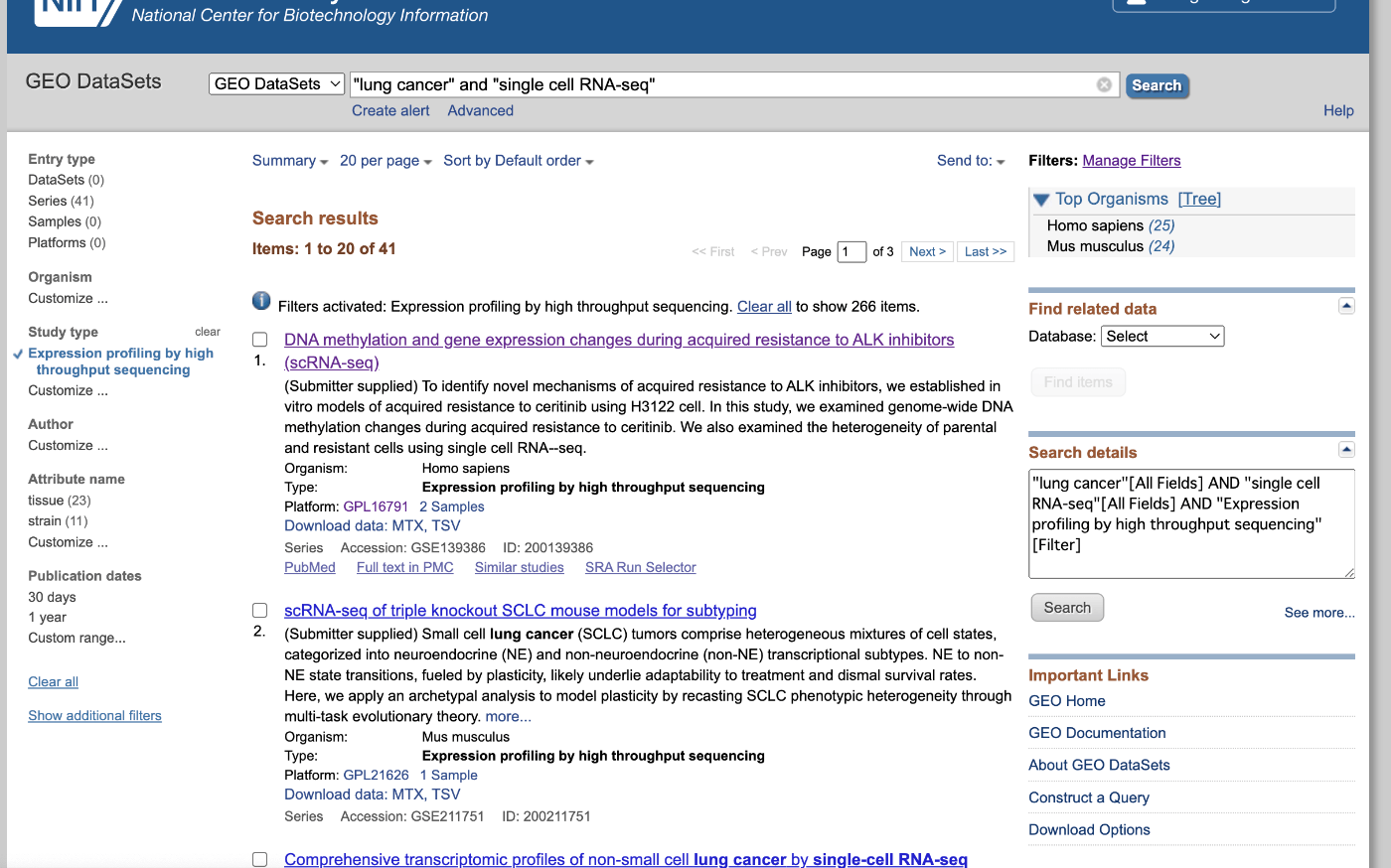
向下滚动,您将找到有关补充文件的信息。点击 " 自定义 " 浏览文件内容。在该样本中,10X 基因组格式文件的条形码、特征和矩阵均可用,可用于该分析。
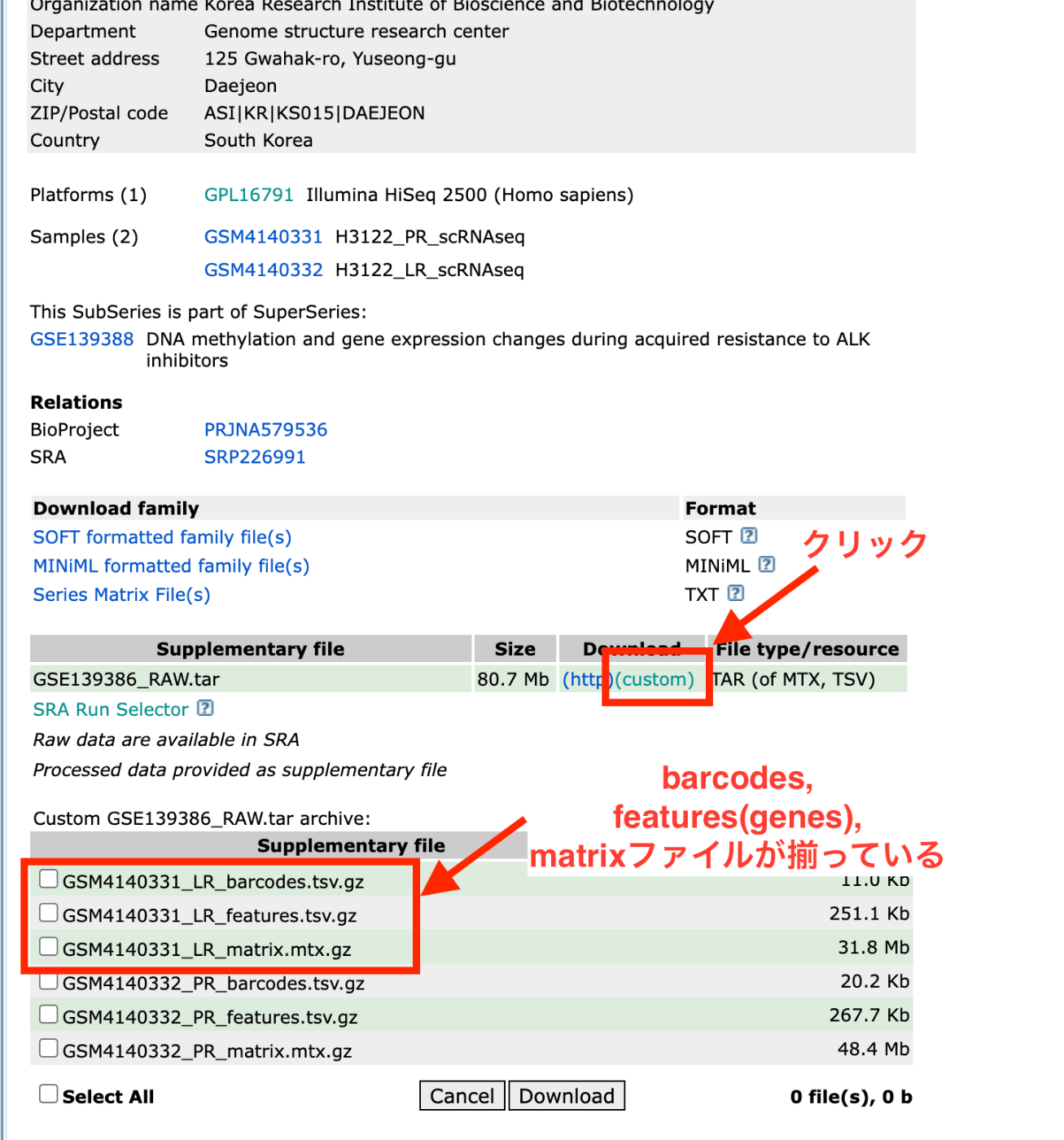
下一步是引入目前分析无法使用的模式。请看 RAW.tar 文件中的搜索结果 [Comprehensive transcriptomic profiles of non-small cell lung cancer by single-cell RNA-seq] (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE119911)。您可以看到它包含一个类似这样的 txt.gz 文件。这种模式不能用于本分析,应避免使用。我们下次再介绍。
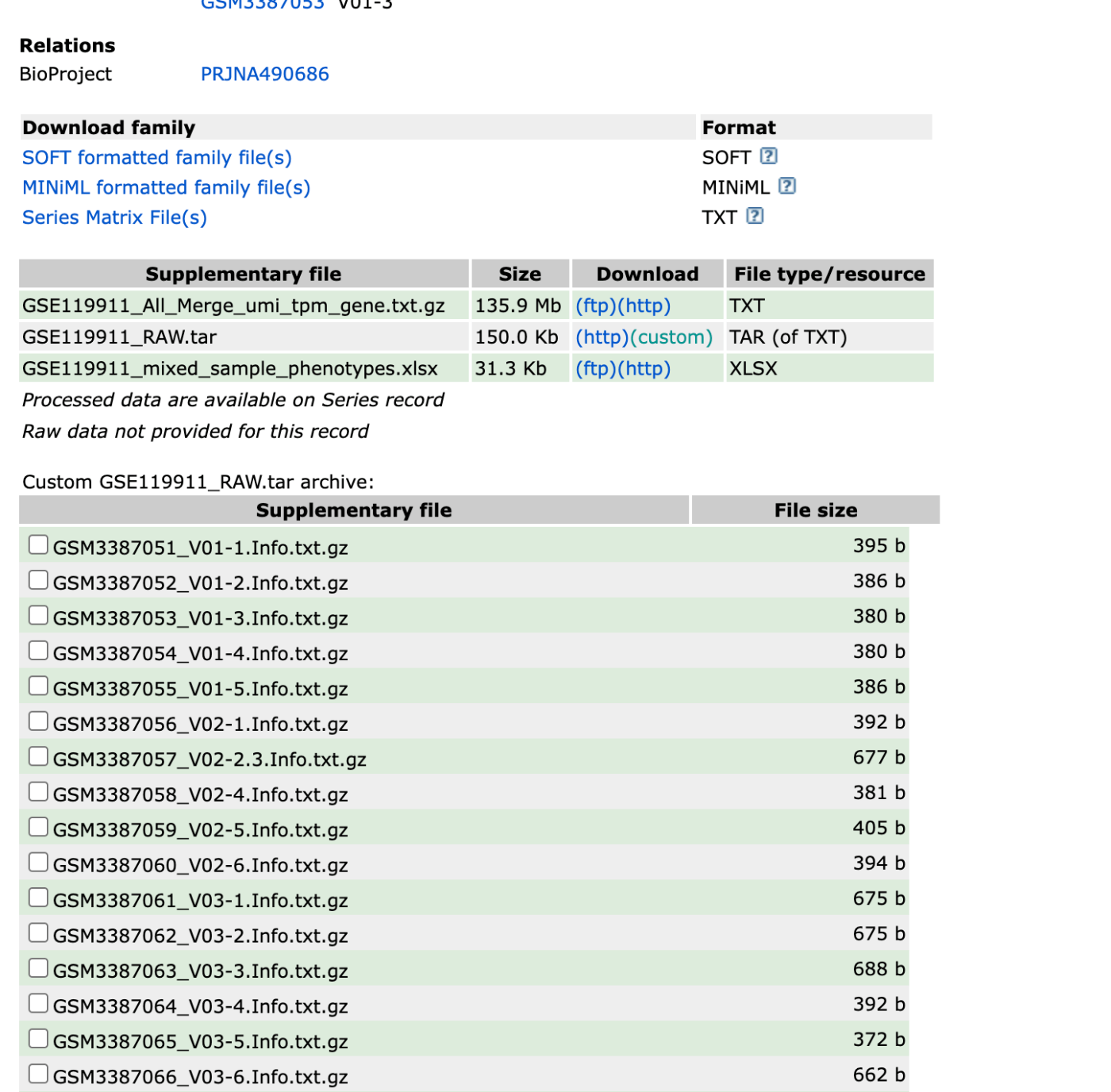
现在,让我们实际分析一下 ALK 抑制剂获得性耐药性过程中的 DNA 甲基化和基因表达变化(scRNA-seq)。选中该文件的复选框,点击下载,将其下载为 tar 文件,并解压到桌面上。
下载的样本内容如下。
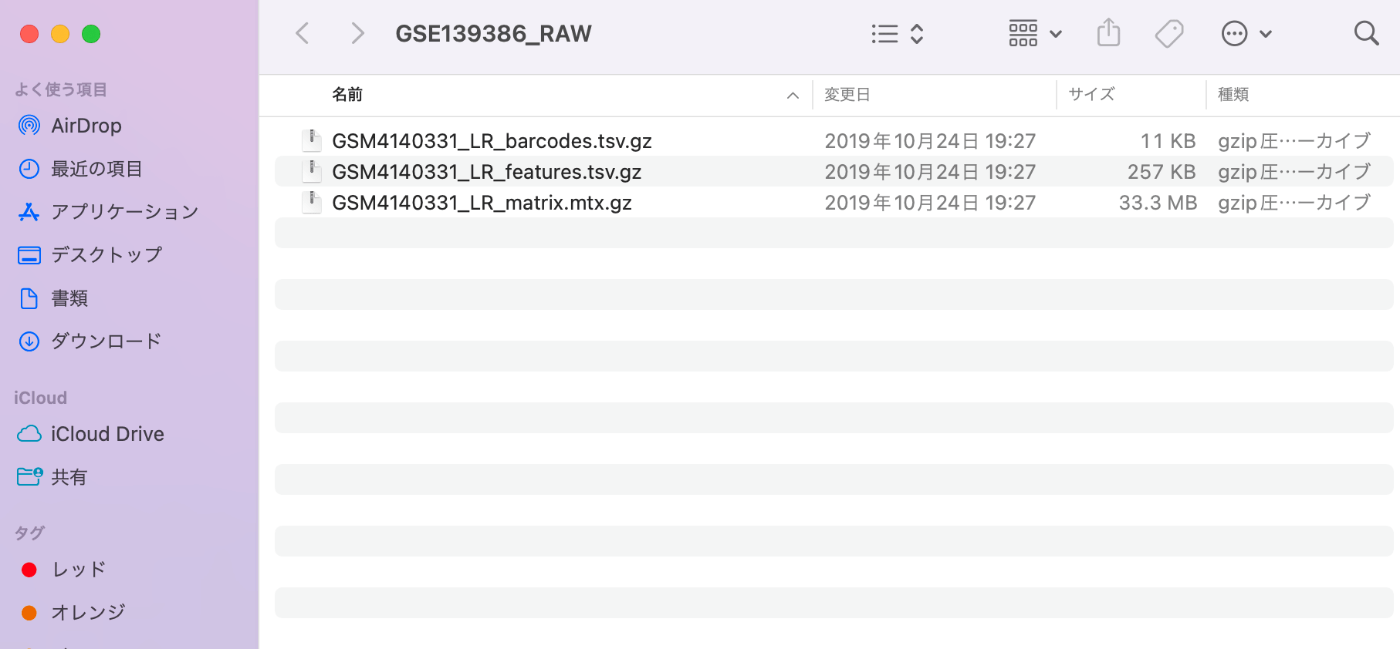
然后将文件分别重命名为 barcodes.tsv.gz、features.tsv.gz 和 matrix.mtx.gz。如果不这样做,就无法在 Seurat 中读取它们。顺便提一下,你不必解压缩这些文件,因为它们仍然是 gz 格式。
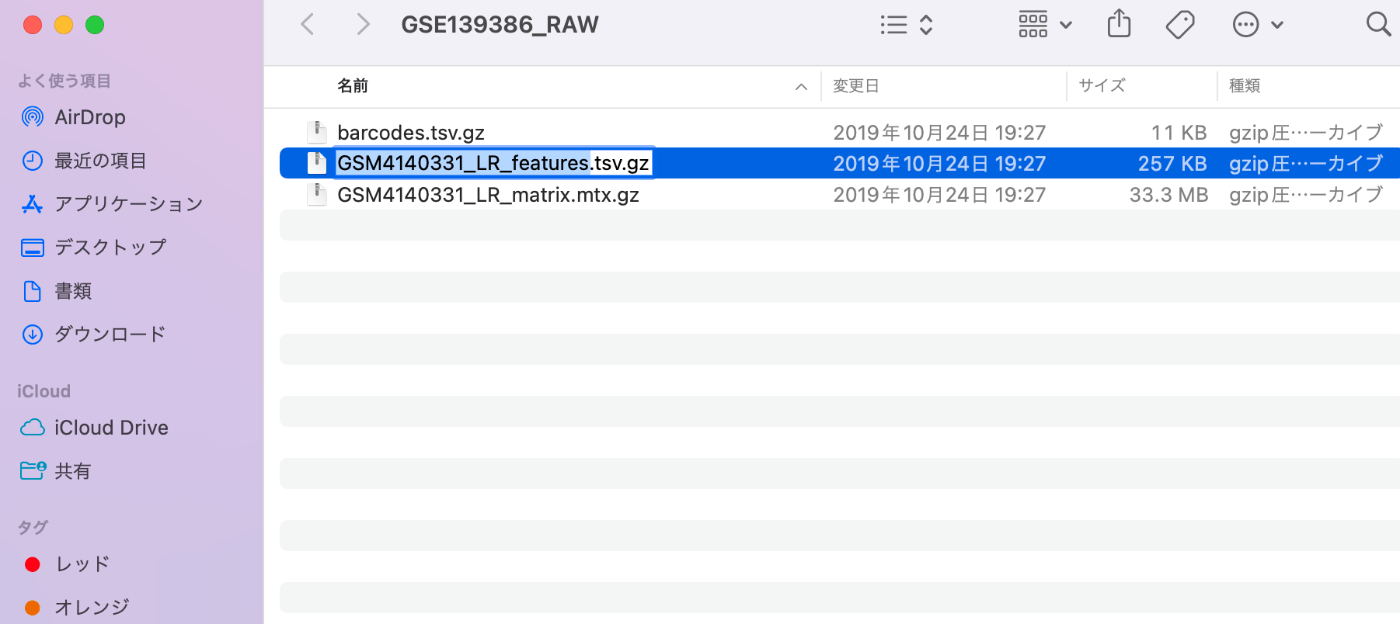
准备工作完成
必要的代码如下。请启动 Rstudio 并加载所需的库。
install.packages("dplyr")
install.packages("Seurat") # 使用Docker镜像的用户建议跳过此步骤
install.packages("patchwork")
library(dplyr)
library(Seurat)
library(patchwork)
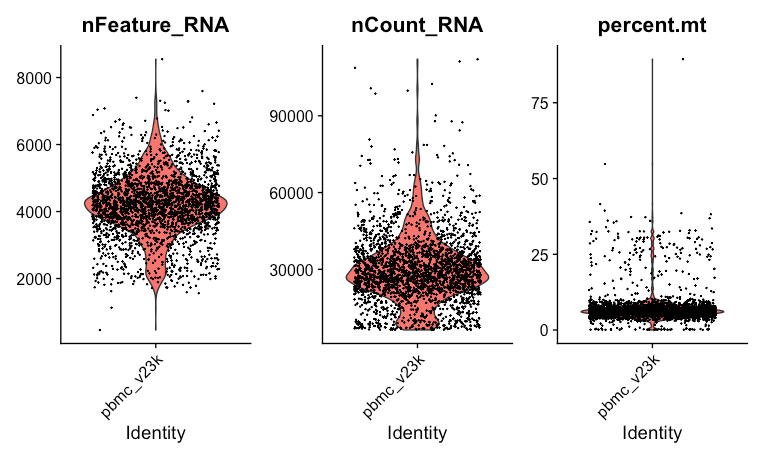
使用以下代码读取样本并显示用于质量检查的小提琴图。
# 读取样本
pbmc_v2.data <- Read10X(data.dir = "./GSE139386_RAW/")
# 显示小提琴图
pbmc_v2 <- CreateSeuratObject(
counts = pbmc_v2.data,
project = "pbmc_v23k",
min.cells = 3,
min.features = 200
)
pbmc_v2[["percent.mt"]] <- PercentageFeatureSet(pbmc_v2, pattern = "^MT-")
VlnPlot(
pbmc_v2,
features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3
)
如果成功,将会显示如下所示的小提琴图!