A visual language foundation model for computational pathology
摘要¶
随着数字病理学的加速应用和深度学习的进步,已经开发出了适用于多种病理学任务的稳健模型,这些任务涵盖了多种疾病和不同的患者群体。然而,由于医学领域标签稀缺,模型训练常常面临困难,且模型的使用受限于其训练的特定任务和疾病。此外,大多数组织病理学模型只利用图像数据,这与人类教学和推理组织病理实体的方式形成了鲜明对比。我们提出了一种基于对比学习的视觉 - 语言基础模型 CONCH(CONtrastive learning from Captions for Histopathology),该模型使用多样化的组织病理学图像、生物医学文本以及超过 117 万幅图像 - 字幕对进行任务不可知的预训练。在一系列 14 个不同的基准测试中评估,CONCH 能够转移到涉及组织病理学图像和/或文本的广泛下游任务中,实现组织学图像分类、分割、字幕生成、以及图像到文本和文本到图像的检索方面的最新性能。CONCH 在当前组织病理学视觉 - 语言预训练系统中取得了重大进步,具有直接促进需要最少或无需进一步监督微调的基于机器学习的工作流程的潜力。
正文¶
在众多疾病的诊断中,病理学家对组织的检查仍然是金标准。近年来,计算病理学的兴起利用人工智能(AI)解决病理学中的问题,显示出在许多任务中的显著进步,包括转移瘤检测、癌症亚型分析、生存预测、未知原发部位预测、图像搜索以及分子变化预测等。此外,当前该领域的进展是在开发针对特定任务的模型的范式下进行的,这些任务使用大规模的标记训练样本,例如在淋巴结转移检测和前列腺癌分级中。然而,整体滑片图像(WSIs)的数据收集和标注过程非常劳动密集,且不易扩展到开放集识别问题或罕见病,这两者在病理学实践中很常见。由于存在数千种可能的诊断和许多其他任务,为病理工作流的每个步骤训练单独的模型是不切实际的。尽管这些任务多样,但它们都涉及视觉数据的分析,或包括‘组学’和其他多模态数据源的结构化信息。然而,病理学实践和病理学发现的交流广泛使用自然语言,无论是病理学家为患者和其治疗临床医生准备的报告,还是详细说明新的组织病理实体的期刊文章,或是教导住院医师如何实践病理学的教科书章节。
一般机器学习社区在使用视觉和语言信息的基础模型上取得了巨大进展。代表性的工具如 CLIP、ALIGN 和 CoCa 等,使用大规模图像 - 标题对进行视觉语言基础模型的预训练——这些任务不可知的预训练模型在下游视觉和视觉语言任务中表现出强大的性能。在更广泛的生物医学成像领域,视觉语言数据已被用于多种任务,包括 X 光报告生成、零样本分类和检索等。然而,在计算病理学中将视觉和语言数据整合用于表示学习的研究数量很少,最近的研究显示使用成对的图像 - 标题数据学习有意义的视觉表征,并为组织病理学开发基础模型的潜力,这些模型可以在零样本设置中转移到多个下游任务,即不使用任务特定的训练数据。然而,这些研究在组织病理学特定的预训练数据规模上受限,由于该领域缺乏现成的图像 - 标题对,导致从相对较差的表现中获得的实际效用有限。此外,这些模型的更广泛能力仍有待深入探索。
鉴于任务的多样性、获取大规模罕见病数据集或发现组合的困难以及语言在病理学实践中的核心作用,我们有必要开发(1)利用大规模预训练并能够跨任务通用的高性能视觉语言基础模型;(2)广泛研究这些模型的潜在应用范围,以理解它们的实用性和限制。我们介绍了 CONtrastive learning from Captions for Histopathology(CONCH),这是一个使用多源组织病理学图像、生物医学文本和超过 117 万个图像 - 标题对通过任务不可知的预训练开发的视觉语言基础模型。基于 CoCa 的最先进的视觉语言基础预训练框架,CONCH 采用图像编码器、文本编码器和多模态融合解码器,并通过一种对比对齐目标的组合进行训练,该目标寻求在模型的表示空间中对齐图像和文本模态,以及一个学习预测与图像相对应的标题的标题生成目标。我们调查了 CONCH 在包括图像块分类、千兆像素 WSIs、跨模态图像到文本和文本到图像检索、图像分割和图像标题生成等 14 个不同基准上的能力。我们证明,我们的模型在所有基准中相对于其他视觉语言基础模型(包括 PLIP、BiomedCLIP 和 OpenAICLIP)都实现了最先进的性能,并且经常大幅度超过同时期的基线。

a, 自动化数据清洗流程。教育资源(EDU)和 PubMed Central 开放获取数据集(PMC OA)的部分数据经过人工清洗,用于训练一个目标检测器以检测组织病理学图像,一个语言模型以分割指向多个图像的标题,以及一个匹配模型以将检测到的图像与其对应的标题匹配。清洗过程产生了 179 万个图像 - 文本对的数据集,随后我们筛选掉指向非人类对象的对,创建了我们的 CONCH(仅限人类)预训练数据集,共 117 万对(有关数据清洗的详细信息,请参阅方法部分;关于数据筛选的消融实验讨论,请参阅讨论部分)。b, 人类预训练数据集中图像 - 文本对的估计分布按主题分类。请注意,预训练数据涵盖了多样的病理学主题。插图,PMC-Path 与 EDU 的标题长度分布比较(有关各类别标题的词云,请参见扩展数据图 1)。c, 视觉 - 语言预训练设置。CONCH 包括一个图像编码器、一个文本编码器和一个多模态文本解码器。预训练过程采用对比和标题生成两种目标。对比目标通过最大化配对图像和文本嵌入之间的余弦相似度分数来对齐图像和文本编码器,而标题生成目标则最大化在图像和之前生成的文本条件下生成正确文本的可能性(有关详细信息,请参阅方法部分)。\
表示句子开始;attn 表示注意力机制;\ 表示句子结束。d, 雷达图比较了 CONCH 与基线在各种下游任务上的表现。CONCH 在一系列任务上显著超越基线,这些任务包括零样本分类、检索和零样本分割(有关每个任务和度量的详细描述,请参阅结果部分)。
结果¶
多样组织和疾病的零样本分类¶
通过对比对齐的视觉 - 语言预训练使得模型能够直接应用于下游分类任务,无需进一步的标记样本进行监督学习或微调。这种零样本转移能力使得一个预训练的基础模型能够即开即用地应用于具有任意数量类别的不同下游数据集,与目前为每个新任务训练一个新模型的范式相比。虽然我们不期望零样本分类当前对大多数临床用例足够准确,但在某些任务中,我们发现 CONCH 表现出色,可能作为传统监督学习的强大基线,特别是在训练标签稀缺时。
针对一个任务,我们首先使用一组预定的文本提示来表示类别或类名集合,每个提示对应一个类别。然后通过将图像与模型共享的图像 - 文本表示空间中最相似的文本提示匹配来对图像进行分类(见图 2a;详细方法请参见方法部分)。在实践中,同一概念在文本中经常有多种表述方式(例如,“乳腺浸润性小叶癌(ILC)”和“乳腺 ILC”);因此,我们在预测时为每个类别创建了多个文本提示的组合,这通常比使用单一文本提示能够提高预测性能(扩展数据图 2)。此外,尽管之前的研究主要集中在感兴趣区域(ROI)级别的分类任务上,我们还利用 MI-Zero 技术探索了我们模型在千兆像素 WSI 上的零样本能力,该技术将 WSI 划分为较小的 batches,并随后将各个 batches 级别的得分汇总成幻灯片级预测(见图 2b)。
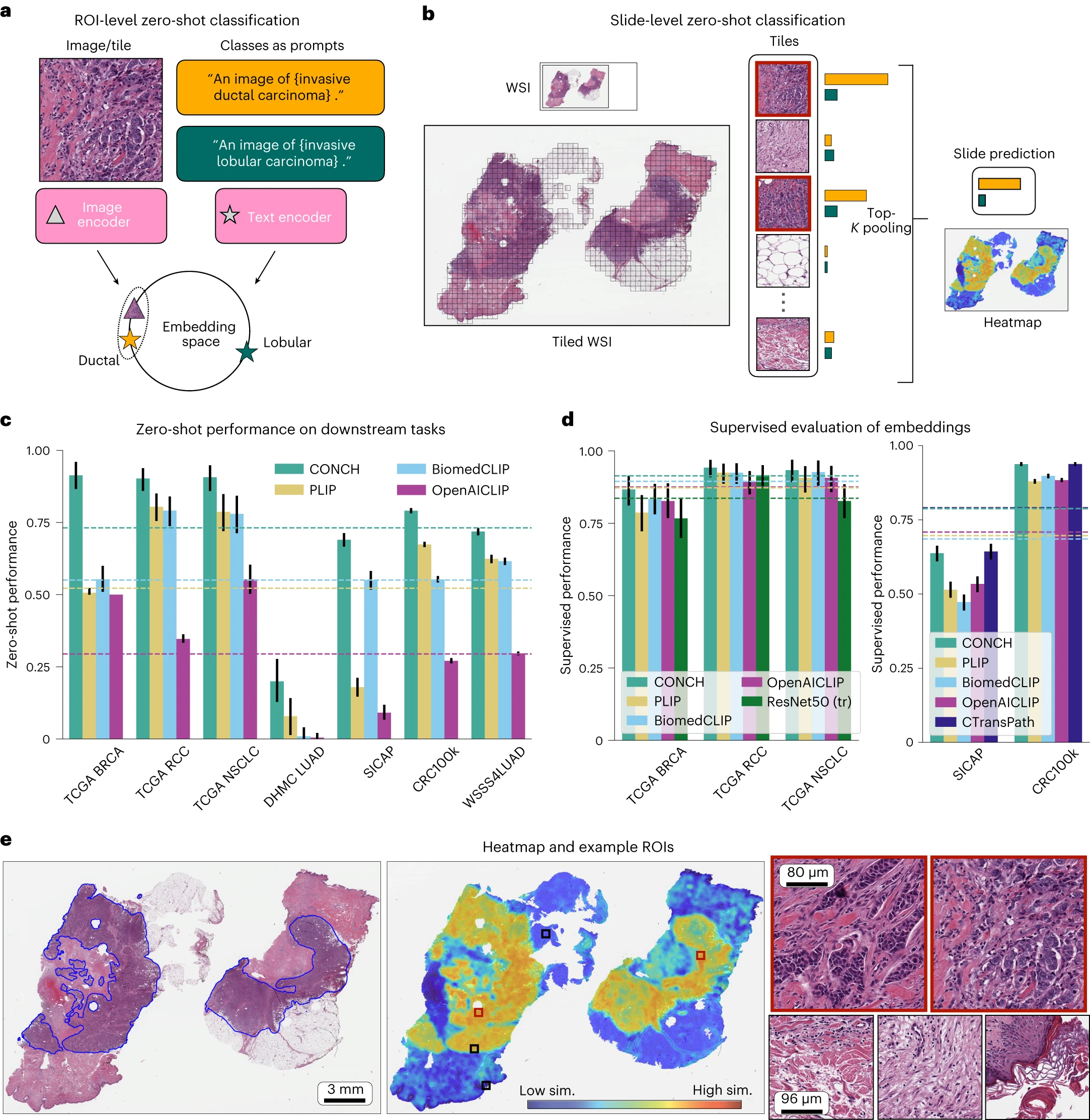
a, 使用对比对齐的图像和文本编码器进行零样本分类的示意图。为每个类别构建一个提示,图像根据与图像在共享嵌入空间中最接近的提示的嵌入进行分类。b, WSI 的零样本分类。每个 WSI 被划分成 batches 并按照 a 中的方法处理。batches 的相似度分数通过顶 K 汇总法聚合成幻灯片级相似度分数,其中最高的对应于幻灯片级预测。在 c 和 d 中,虚线表示任务平均值。误差条代表 95% 置信区间,中心点对应每个度量的计算值,具体如下所述。c, 下游亚型(TCGA BRCA, n = 150; TCGA RCC, n = 225; TCGA NSCLC, n = 150; DHMC LUAD, n = 143; CRC100k, n = 7,180; WSSS4LUAD, n = 4,693)和分级(SICAP, n = 2,122)任务的零样本表现。DHMC LUAD 报告了 Cohen's κ,SICAP 报告了加权 Cohen's κ,其他所有任务报告了平衡准确度。补充表 1-7 报告了其他度量。d, 每个模型嵌入的监督评估。ROI 级任务(CRC100k 和 SICAP)使用线性探测,而幻灯片级任务使用 ABMIL,报告的度量与 c 中相同(更详细的结果见补充表 15-19)。e, 从左到右:病理学家标注的 IDC、相应的热图和放大的选定 batches。热图根据幻灯片中每个 batches 与预测类别标签对应的文本提示之间的余弦相似度得分进行着色。我们发现标注图像与高相似度区域之间有极好的一致性,高相似度区域的 batches 展示了经典的 IDC 形态,而低相似度区域包含乳腺的间质或其他正常成分。
总体而言,我们在四个幻灯片级别的分类任务上评估了 CONCH:癌症基因组图谱(TCGA)BRCA(侵袭性乳腺癌亚型分类)、TCGA NSCLC(非小细胞肺癌亚型分类)、TCGA RCC(肾细胞癌亚型分类)和达特茅斯希区柯克医学中心(DHMC)LUAD(肺腺癌组织模式分类);以及三个感兴趣区域(ROI)级别的任务:CRC100k(结直肠癌组织分类)、WSSS4LUAD(LUAD 组织分类)和 SICAP(Gleason 模式分类)。我们采用平衡准确度作为 TCGA NSCLC、TCGA RCC、TCGA LUAD、CRC100k 和 WSSS4LUAD 的主要评估指标,这考虑了类别不平衡,通过均等加权每个类别的准确度得分。按照社区标准,我们分别使用 Cohen 的κ和二次加权 Cohen 的κ作为 LUAD 模式分类和 Gleason 模式分类的主要指标,因为这些任务被认为更加主观,通常意味着更高的评分者间变异性。更详细的模型性能报告见补充表 1-14,评估数据集的详细描述见方法部分。
在幻灯片级别的基准测试中,CONCH 在所有任务上均显著优于其他先进的视觉语言基础模型(PLIP、BiomedCLIP 和 OpenAICLIP),通常差距较大(见图 2c)。例如,在 NSCLC 亚型分类和 RCC 亚型分类中,CONCH 分别实现了 90.7% 和 90.2% 的零样本准确率,并且分别比下一个表现最好的模型 PLIP 高出 12.0% 和 9.8%,P<0.01(方法部分的“统计分析”)。在更具挑战性的 BRCA 亚型分类任务中,CONCH 实现了 91.3% 的零样本准确率,而其他模型的表现接近随机机会,准确率从 50.7%(PLIP)到 55.3%(BiomedCLIP),几乎比 CONCH 低 35%(P<0.01)。最后,在 LUAD 模式分类任务中,CONCH 实现了 0.200 的κ分数,比下一个表现最佳的模型 PLIP 高出 0.12,尽管未达到显著性(P=0.055)。在 ROI 级别的基准测试中,我们观察到类似的结果,其中 CONCH 在 SICAP 上实现了 0.690 的零样本二次κ(比 BiomedCLIP 高出 0.140,P<0.01),在 CRC100k 上实现了 79.1% 的零样本准确率(比 PLIP 高出 11.7%,P<0.01),在 WSSS4LUAD 上实现了 71.9% 的零样本准确率(比 PLIP 高出 9.5%,P<0.01)。这些结果表明,除了在相对容易的任务上实现更准确的预测外,CONCH 还能在其他模型可能特别挣扎的一些更具挑战性的任务上实现有意义的预测。
在使用零样本转移对 WSI 进行分类时,除了计算聚合的幻灯片级预测外,我们还可以创建热图来可视化幻灯片中每个 batches 与预测类别标签对应的文本提示之间的余弦相似度得分。高相似度得分的区域被模型认为与诊断(例如侵袭性导管癌(IDC))匹配较近,而低相似度得分的区域则不匹配诊断(见图 2e)。在一个乳腺 IDC 幻灯片的示例中,我们发现热图中突出显示的区域与病理学家注释的肿瘤区域非常相似(见图 2e,左侧和中间)。由于幻灯片级预测得分是给定类别的顶 K batches 的相似度得分的简单平均,热图通过直接突出显示模型决策过程中涉及的区域,使人类可解释性成为可能,可以高分辨率显示给人类用户进行检查(见图 2e,右侧)。其他示例在扩展数据图 3-5 中可视化。这些发现表明我们的模型的零样本识别能力有可能用于 WSI 上的粗粒度组织分割,我们在结果中对此进行了量化评估(“零样本分割”)。
使用特定任务的监督学习进行少样本分类¶
对比预训练的视觉语言模型在病理组织学中的零样本识别能力使得可以有效且快速地将单一基础模型应用于潜在的广泛任务,无需经历繁琐的训练数据收集、标注和监督模型训练的过程。然而,有时可能仍希望使用带标签的训练样例专门化模型以最大化特定任务的性能,理想情况下使用尽可能少的标签。在本节中,我们探讨了使用视觉语言基础模型的图像编码器主干的预训练表示进行特定任务的监督分类时的标签效率。对于使用监督训练的每个基准测试,我们使用官方提供的训练集(如果提供的话)或在保留用于零样本评估的案例集后的数据集中剩余的案例(方法部分的“下游评估数据集”)。对于幻灯片级任务,我们根据广泛使用的基于注意力的多实例学习(ABMIL)算法训练了弱监督分类模型。对于 ROI 级任务,我们在每个编码器的全局(例如,分类(\

a-c,我们研究了在少样本设置中不同视觉语言预训练编码器的标签效率,其中我们变更了每个类别的训练标签数量(nc),对于 nc=1、2、4、8、16……直到训练集中可用标签的最大数量。对于每个 nc,我们抽样了五组不同的训练样本,并在每个训练集上使用幻灯片级标签训练了一个弱监督 ABMIL 模型(详见方法部分的“监督和弱监督分类实验”)。我们通过箱线图显示了它们在 BRCA 亚型分类(a)、RCC 亚型分类(b)和 NSCLC 亚型分类(c)的单独模型性能(每个箱体 n=5),以研究在使用极少训练样本进行监督学习时模型性能的变异。箱体显示四分位数值,须部延伸到 1.5 倍四分位距范围内的数据点。为参考,每个模型的零样本性能以虚线显示在同一图表上。就少样本监督学习而言,CONCH 在不同大小的训练集和所有任务中都比其他编码器取得了更好的性能(基于五次运行的中位准确率)。此外,CONCH 的零样本性能出乎意料地具有竞争力,超过了 PLIP、BiomedCLIP 和 OpenAICLIP 在 BRCA 和 NSCLC 亚型分类中使用多达 64 个标签的少样本性能。Sup.代表监督学习。
应用于罕见疾病分类¶
虽然之前的研究主要关注在相对狭窄的任务上评估视觉语言预训练模型的零样本和少样本性能,这些任务对应的可能类别集小(2-5 个类别),但据我们所知,这些模型在涉及罕见病的大规模、潜在的细粒度疾病分类中的有效性尚未被研究。在这里,我们探讨了 CONCH 在识别多达 30 种类别的脑瘤的能力,所有这些脑瘤根据 RARECARE 项目定义为罕见癌症,即每 10 万人中年粗发病率小于 6,这一定义被国家癌症研究院的监测、流行病学和最终结果(SEER)计划采用。我们使用 EBRAINS 数据集构建了一个大规模的亚型基准,并评估了各种模型的零样本和监督学习的有效性。
在零样本分类中,CONCH 在 30 类亚型问题上达到了 37.1% 的平衡准确率(扩展数据图 7 和补充表 20),远超过 3.3% 的随机机会基线以及表现第二好的视觉语言预训练零样本分类器 BiomedCLIP(+17.0%,P<0.01)。然而,这些模型的总体低零样本性能表明,当前代的视觉语言基础模型可能还不能直接在野外环境中执行,即开放集识别病理学中的多样性疾病,当评估涉及多类别和罕见实体的更具挑战性的基准时,它们可能只能实现有限的性能。
接下来,我们研究了我们视觉编码器预训练表征的质量,用于训练弱监督 ABMIL 分类模型。与前一部分类似,我们还包括了额外的预训练视觉编码器基线,包括 CTransPath、KimiaNet 和截断的 ResNet50(以 ImageNet 初始化权重)。我们发现,尽管由于任务的挑战性,CONCH 的零样本性能有限,但冻结的 CONCH 编码器的图像嵌入可以用来开发性能强大的分类模型,当与弱监督学习结合时。具体来说,CONCH 结合 ABMIL 实现了 68.2% 的平衡准确率(扩展数据图 7a 和补充表 21),超过了仅使用视觉的自监督学习(SSL)预训练的 CTransPath 模型(+6.8%,P<0.01),以及其他所有测试的视觉语言预训练模型(PLIP +10.7%,P<0.01;BiomedCLIP +14.4%,P<0.01;OpenAICLIP +17.8%,P<0.01)。这些结果展示了强大的预训练视觉语言模型作为标准弱监督学习计算病理学工作流程中有效的仅图像编码器的潜在用途,即使任务主要涉及罕见疾病。最后,我们还研究了各种模型的少样本学习性能,这一研究受到了由于数据可用性有限而训练罕见病诊断模型需要高标签效率的需求的驱动。我们观察到与所有其他测试模型相比,CONCH 的标签效率更高,其他模型通常需要大约四倍的标签才能达到可比的性能(扩展数据图 7b)。
零样本跨模态检索¶
通过学习对齐的视觉和语言嵌入的潜在空间,我们的模型能够在零样本设置中执行跨模态检索,即根据图像查询检索相应的文本条目(图像到文本,简称“i2t”)或反之亦然(文本到图像,简称“t2i”)。这项任务自然适合于图像搜索应用,这在生物医学领域非常有用,例如识别研究队列或临床试验中的案例、帮助罕见病呈现或形态学,以及收集案例或帮助创建教育资源。为执行文本到图像的检索(图像到文本的方向类似),我们使用文本编码器嵌入作为查询的文本输入。然后我们使用查询文本嵌入在潜在空间中检索相似的图像(见图 4b)。
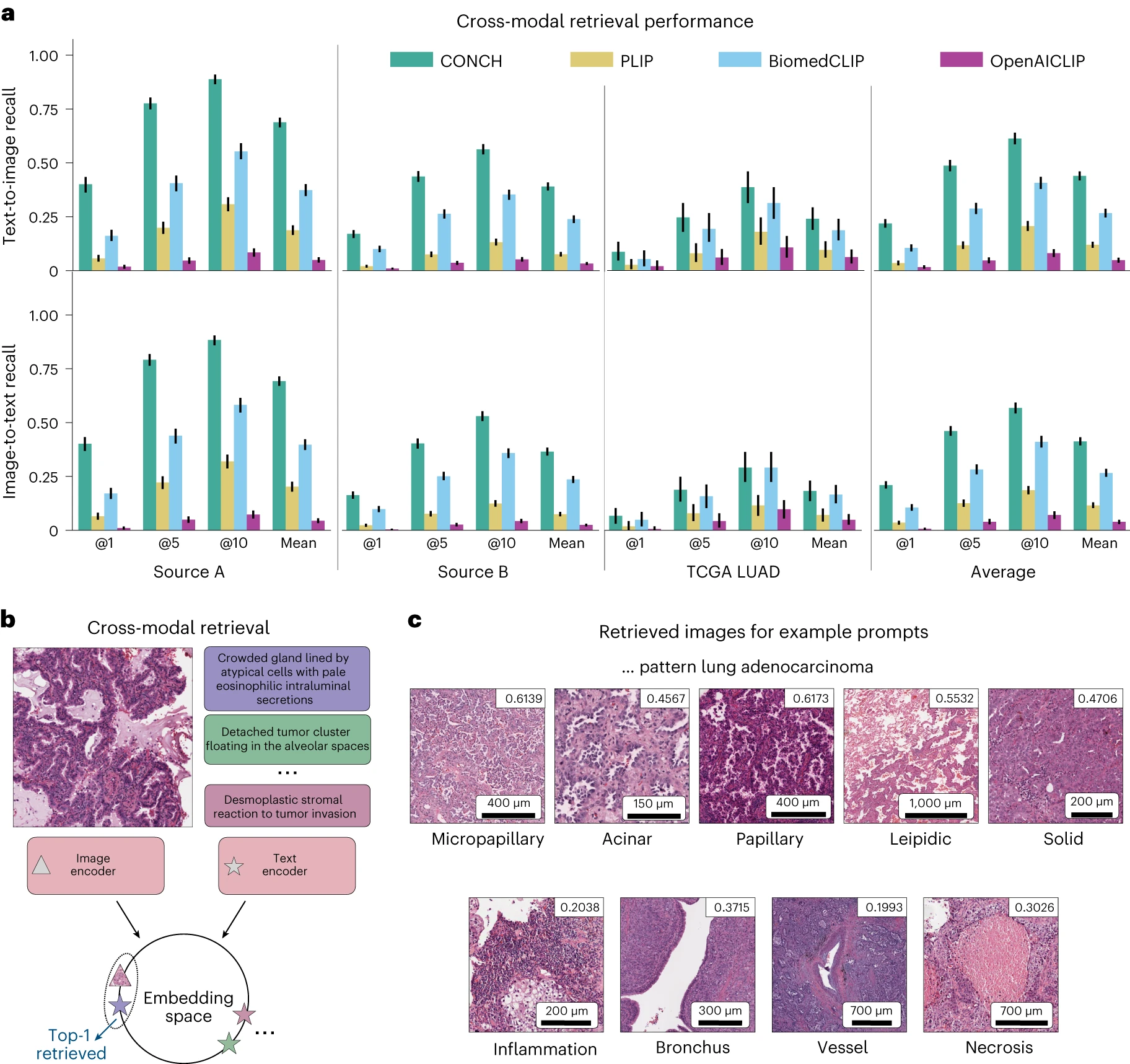
a, 我们评估了模型在三个图像 - 文本对数据集上的跨模态检索性能(来源 A,n=797;来源 B,n=1755;TCGA LUAD,n=165)。在嵌入空间中计算查询图像与数据库中所有文本样本之间的相似度。检索最相似的前 K 个文本。我们报告了 K ∈ {1, 5, 10}的召回率@K 和平均召回率,后者对 K 进行了平均。我们展示了每个检索任务(列)的文本到图像(顶行)和图像到文本(底行)检索。最右列报告了每个度量的任务平均值。CONCH 在所有检索任务上均优于其他基准。误差条显示 95% 置信区间。b, 零样本图像到文本检索的示意图(文本到图像的方向类似)。c, 使用与 LUAD 相关的查询从 TCGA LUAD 中检索的前五张图像的示例,右上角显示了余弦相似度分数。使用更多样化查询的其他数据集的示例显示在扩展数据图 7 中。总的来说,我们发现模型检索的图像与文本提示中描述的内容相匹配。
我们在三个图像 - 文字配对数据集上评估了我们的模型,即来源 A 和来源 B(这两个都是模型预训练期间保留的源,涵盖了多种一般病理学概念)以及 TCGA LUAD(这是一个更为特定的数据集,由从 TCGA 中提取的 LUAD 切片构成,并在内部用文字标注)。根据先前的研究,我们使用召回率@K 作为跨模态检索的评估指标(更详细的检索数据集描述见方法部分)。
平均而言,在这三个数据集上,CONCH 显著超过了基线模型,取得了 44.0% 的文本到图像检索的平均召回率,并且以 17.3% 的优势超过了表现第二好的模型 BiomedCLIP,P<0.01(图 4a)。对于来源 A 和来源 B,CONCH 在文本到图像检索的平均召回率分别达到了 68.8% 和 39.0%,分别以 31.5% 和 15.1% 的优势超过了表现第二好的模型 BiomedCLIP(两者 P<0.01)。对于 TCGA LUAD,CONCH 的文本到图像平均召回率为 24.0%,超过了表现第二好的模型 BiomedCLIP 5.3%,但这一差距在统计上不显著(P=0.22)。然而,CONCH 显著优于 PLIP 和 OpenAICLIP(P<0.01)。所有三个数据集的图像到文本检索表现趋势与文本到图像检索相同,统计显著性也相同,除了 TCGA LUAD 中 CONCH 和 BiomedCLIP 之间的差距略小(1.6%)。更详细的模型性能报告请参阅补充表 22-27。基于这些结果,CONCH 能够比基线模型更准确地执行跨模态检索。
除了使用配对的文字作为查询外,我们还展示了使用简单的与 LUAD 相关的文本提示(例如,‘实性模式 LUAD’)在 TCGA LUAD 数据集上使用 CONCH 检索的结果示例(图 4c)。为了提供更复杂的文本查询示例,如‘cribriform prostatic adenocarcinoma’,我们使用了从 1620 例中抽样的 321,261 块切片的高度多样化数据集,涵盖 108 个 OncoTree 编码(扩展数据图 8)。然而,由于该数据集没有配对的文本数据,我们无法量化检索性能。呈现的示例已经由病理学家确认与文本查询密切相关。
零样本分割¶
尽管 WSI 可以是千兆像素大小,它们通常是异质的,包括多种细胞类型、形态和组织结构,每种通常只占幻灯片的一小部分。因此,在幻灯片级别上进行分割是一项困难且有用的任务,它可以根据感兴趣的特征识别 WSI 的不同区域,并可以减少下游应用所需的切片数量。然而,因为在子幻灯片级别上收集标注数据既昂贵又费力,所以一种能在零样本设置中执行幻灯片级分割的通用模型具有价值。在这项工作中,我们探索了在没有标记示例的情况下,直接使用我们模型所展示的零样本检索和分类能力来执行 WSI 上的粗粒度组织分割的可能性。
给定一个 WSI,我们将组织区域分成较小的图像切片,并将给定的分割任务视为使用零样本分类对每个切片进行分类,并将预测的类别标签分配给切片中的所有像素,对所有切片进行操作(图 5a)。为了最小化相邻切片边界处预测值的突然转变,我们对 WSI 进行了 75% 重叠的切片,并在重叠区域平均预测得分,以在预测的分割图中实现更平滑的外观。我们在 SICAP 上评估了我们的模型,用于前列腺肿瘤与正常组织的分割,在 DigestPath 上用于 CRC 标本的恶性与良性组织的分割。我们报告了广泛使用的 Dice 得分,以及每项任务的精确度和召回率,对每个数据集中的所有图像进行宏观平均(更多详情见方法部分)。更详细的模型性能结果请参阅补充表 28 和 29。
CONCH 在两项任务中均优于其他模型(图 5b,c)。在 SICAP 中,CONCH 的平均 Dice 得分为 0.601(PLIP 0.549, P=0.08;BiomedCLIP 0.484, P<0.01),平均召回率为 0.751(PLIP 0.644, P<0.01;BiomedCLIP 0.557, P<0.01),平均精确度得分为 0.672(PLIP 0.605, P=0.024;BiomedCLIP 0.536, P<0.01)。在 DigestPath 中,CONCH 的平均 Dice 得分为 0.615(PLIP 0.426, P<0.01;BiomedCLIP 0.446, P<0.01),平均召回率为 0.709(PLIP 0.541, P<0.01;BiomedCLIP 0.601, P<0.01),平均精确度得分为 0.663(PLIP 0.526, P=0.024;BiomedCLIP 0.581, P<0.01)。此外,我们发现,尽管该方法的粗粒度和零样本特性,该模型在某些情况下能够产生相当准确的像素级分割掩模,如图 5d,e 所示。
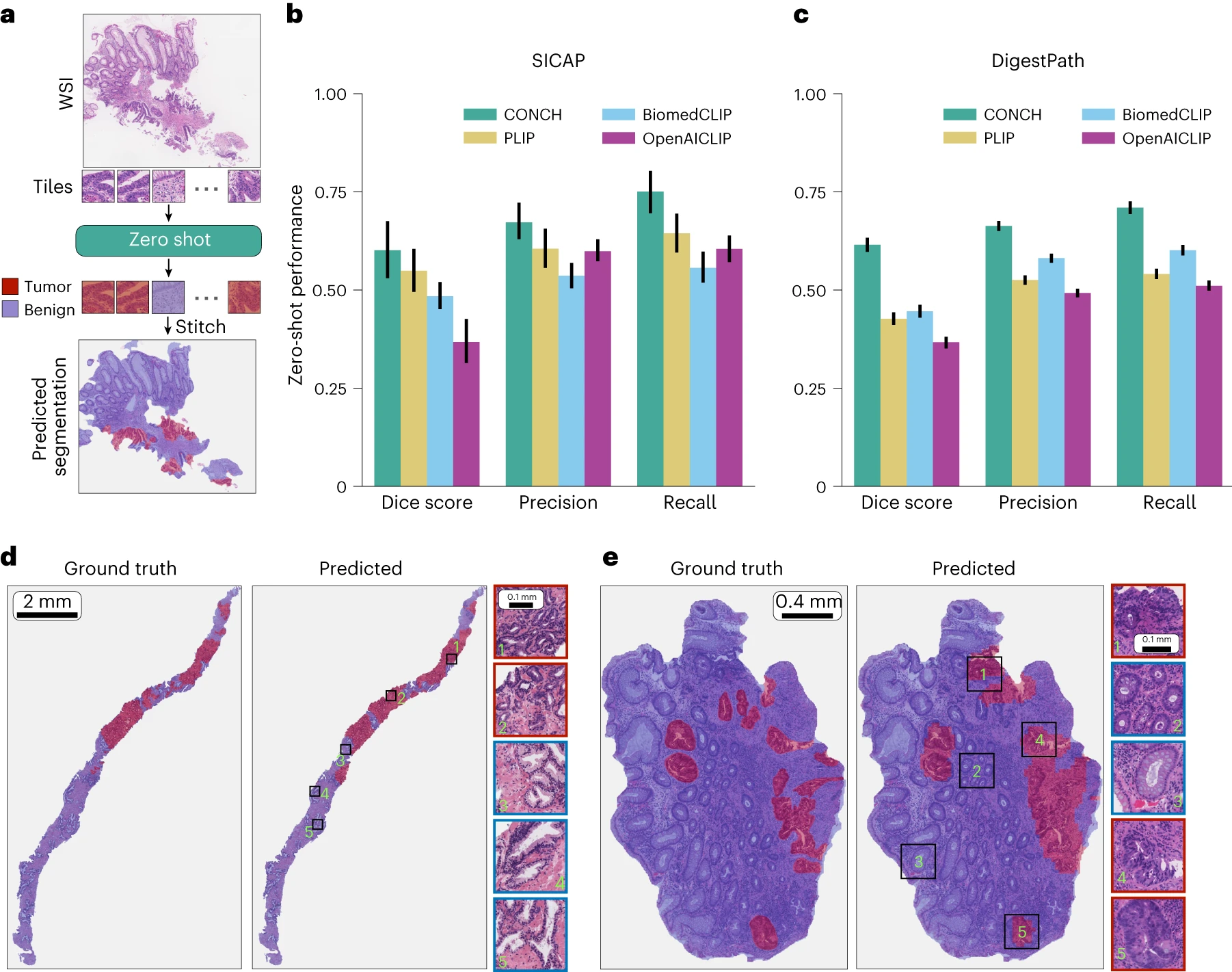
a, 示意图展示了在 WSI(或大型组织切片)上进行零样本分割的过程。为了进行分割,我们将每个 WSI 划分成多个 batches,并使用零样本分类来预测每个 batches 的标签。这些 batches 级预测被拼接起来,形成预测的分割掩模。 b,c, 在 SICAP(n=31)(b)和 DigestPath(n=250)(c)数据集上,CONCH 及基线模型的零样本分割性能。报告了宏观平均的 Dice 得分、精确度和召回率。误差条表示 95% 置信区间。 d,e, CONCH 在 SICAP(d)和 DigestPath(e)数据集上 WSI 的分割预测示例。左侧面板显示了真实情况,右侧面板显示了预测的分割掩模,示例区域已放大。红色和蓝色分别指示肿瘤和正常组织。总的来说,在这些示例中,CONCH 对肿瘤区域显示出极好的敏感性,尽管特异性略低,但大多数 CONCH 划分为肿瘤的非肿瘤区域实际上邻近癌变腺体或包含与癌症相关的间质,这一点在 SICAP 和 DigestPath 上都是如此。
讨论¶
在计算病理学领域,大多数先前的工具试图从图像数据和/或结构化的患者数据(如基因组数据)中提取有意义的模式和辨别信号,并忽略了病理学的文本方面。然而,这些方法没有利用图片描述中存在的大量信息,这种信息允许病理学培训者从少数实例图像泛化到现实世界中常常更为多样的图像。尽管近期几项研究试图利用社交媒体或生物医学研究文章中的图像和字幕数据来构建适用于组织病理学领域的视觉语言基础模型,我们发现在许多任务中,这些模型的零样本和监督分类性能仍然有限,阻碍了它们作为组织病理学通用识别或检索系统的实用价值。此外,除了在小型感兴趣区域(ROI)上的工作外,模型在更复杂环境中的表现能力(例如,罕见疾病分类或在异质性千兆像素 WSI 上的肿瘤分割)仍然未被充分探索。
在本研究中,我们展示了通过使用目前最大的特定于组织病理学的配对图像 - 文本数据集(超过 117 万个例子)进行任务不可知预训练,我们能够构建一个高性能的视觉语言基础模型,然后在广泛的临床相关下游任务中展示其实用性,如分类、检索和组织分割。我们的模型配备了强大的零样本识别能力,这可以潜在地减轻为许多特定分类任务标注训练示例的负担,并且我们展示了其零样本性能经常与这些任务中的传统监督学习基线相当甚至超过,在少样本设置下尤为如此。此外,我们模型显著改善的零样本图像到文本和文本到图像的检索能力,将有可能赋予医学实习生、医师和研究者更准确、更灵活地根据图像或自然语言查询检索相关病例或教育示例的能力,一旦能有效地实施到医疗系统或数据库中。配备多模态解码器的我们的视觉语言基础模型还提供了进一步在涉及语言生成(例如,图像字幕;见方法部分的“带微调的字幕制作”以及扩展数据图 9 和补充表 30 中的探索性结果)和/或基于视觉和文本输入的多模态推理的下游任务中进行微调的灵活性。然而,尽管在选定任务中取得了有希望的结果,我们还发现当前的视觉语言预训练模型,包括 CONCH,在涉及大量类别和罕见疾病的具有挑战性的零样本问题上的表现仍然较差(相对于它们的监督学习对应物)。这些观察表明,我们在实现能够真正普遍进行零样本识别或检索的组织病理学基础模型的目标上可能还有很长的路要走。
此外,我们还进行了消融实验,以探索数据过滤、不同预训练算法和单模态预训练对我们模型性能的影响。最值得注意的是,我们发现进行单模态预训练(尤其是视觉编码器 SSL 预训练)可以在大多数任务中改善模型在零样本分类和检索中的表现(更多详情见扩展数据图 10)。
还有一个相对未被充分探索的方面是视觉语言预训练基础模型与针对特定任务的常规端到端监督学习的兼容性。对于一些广泛研究的单病种模型任务,如前列腺腺癌 Gleason 分级,世界各地的多个团队已经付出了巨大努力来构建具有详细 ROI 或像素级标注的大型多样化数据集,以适用于端到端的监督机器学习。鉴于标注数据的丰富,一个自然的问题是,预训练一个在多种组织类型和疾病上的图像和字幕数据上的基础模型是否仍能为这些特定任务带来实际好处?我们尝试通过组建一个来自多个公开来源的超过 200,000 个标记 ROI 的大型多样化数据集来回答这个问题,用于前列腺癌 Gleason 分级的任务,并对我们的视觉编码器进行端到端微调,以及其他一些预训练的标准卷积神经网络(CNN)和视觉变换器(ViT)模型,包括像 KimiaNet 和 CTransPath 这样的领域特定编码器。在我们的实验中,我们发现,即使是配对了数以十万计的标记 ROI 并从 ImageNet 权重或 SSL 预训练中进行迁移学习,微调后的 CONCH 模型仍然可以提供相当大的改进,即使与更大的 ViT-Large 模型相比也是如此(补充表 31)。
虽然最近的一项研究发现,当前的视觉语言预训练基础模型在使用一种特定算法进行 WSI 到 WSI 匹配的特定场景中可能表现不如较小的编码器,但我们在罕见疾病的少样本和弱监督分类中的实验,以及端到端微调表明,CONCH 可以作为组织病理学图像的最先进的视觉编码器,除此之外,还提供了一个解锁额外多模态能力的共享图像 - 文本潜在空间。然而,这些发现强调了持续研究和评估的重要性,以更好地了解基础模型在计算病理学中的优势和局限。
我们研究的一个主要限制是数据预训练的规模,与通用机器学习社区开发大规模视觉语言基础模型所使用的十亿规模数据集相比仍然相形见绌;因此,通过增加组织病理学图像 - 字幕数据集的数量和质量,我们可能会看到零样本识别能力、表征质量和鲁棒性的进一步潜在改进。然而,鉴于预训练中使用的数据规模不断增加,预训练数据和下游测试数据之间的潜在重叠可能性越来越高,这也是以前视觉 - 语言预训练方法在生物医学领域共有的限制。检测和移除重复及近重复通常依赖于启发式方法和人工评估,这在生物医学领域尚未得到充分探索,是未来工作的一个开放研究问题。在本研究中,我们通过确保没有任何公开可用的测试数据集直接源自任何训练来源,并且只在来源级别保留数据,来尽量减少数据重叠的可能性。另一个研究限制是,我们没有调查零样本分类(对图像 ROI 和 WSI 都是如此)在潜在不同数据队列中的鲁棒性,这些数据队列可能具有不同的染色变异、组织制备协议和扫描器特定的成像特征,与使用常规监督学习或参数高效微调技术相比。此外,虽然我们展示了简单地整合少量由病理学家编写的模板和类名已经可以很好地适用于几项任务,但我们没有尝试明确地基于模型的表现(例如,使用验证集)来工程化提示。我们注意到,在小型验证集上明确寻找“好”的提示在实践中可能更有效,同时仍然保留不需要微调模型的好处,尽管这将不再严格被视为零样本转移。此外,作为 WSI 的零样本分类算法,MI-Zero 最适合于每个类的定义形态模式是互斥的任务,并且可能不适用于具有特定假设或指导方针的任务。这包括像 Gleason 评分这样的任务,可能需要考虑主要和次要模式来信息分类,或者肿瘤与正常分类,在这种情况下,只要识别到包含肿瘤的区域,幻灯片就可能被适当标记为“阳性”。我们注意到,对于这些类型的任务,可以调整 MI-Zero 的汇聚函数以更好地适应任务的性质,并将其实施和评估留给未来的研究。最后,虽然目前组织病理学的视觉语言基础模型的研究主要集中在图像级任务上,但这些模型在区域级别(即细胞或亚细胞级别)识别细粒度视觉概念的能力尚未被研究,这意味着其他重要任务,如有丝分裂检测、细粒度组织分割或细胞计数,目前仍不在它们的下游能力范围内。