A whole slide foundation model for digital pathology from real world data
摘要¶
数字病理学提出了独特的计算挑战,因为标准的千兆像素切片可能包含数万个图像切片。之前的模型通常通过对每张切片进行小部分切片的子采样,从而忽略了重要的切片级上下文。在此,我们提出了 Prov-GigaPath,这是一种全切片病理学基础模型,预训练于 Providence 的 171,189 张全切片中的 13 亿个 256×256 病理图像切片。Providence 是一个由 28 个癌症中心组成的大型美国健康网络。切片来源于超过 30,000 名患者,涵盖 31 种主要组织类型。为了预训练 Prov-GigaPath,我们提出了 GigaPath,一种用于预训练千兆像素病理切片的新型视觉变换器架构。为了实现 GigaPath 在具有数万个图像切片的切片级学习中的扩展,GigaPath 采用了新开发的 LongNet 方法用于数字病理学。为了评估 Prov-GigaPath,我们构建了一个数字病理学基准,包括 9 个癌症亚型任务和 17 个病理组学任务,使用了 Providence 和 TCGA 数据。通过大规模预训练和超大上下文建模,Prov-GigaPath 在 26 个任务中有 25 个任务上达到了最先进的性能,并在 18 个任务上显著超越了第二好的方法。我们进一步通过结合病理报告展示了 Prov-GigaPath 在病理学视觉 - 语言预训练中的潜力。总之,Prov-GigaPath 是一个开放权重的基础模型,在各种数字病理学任务中实现了最先进的性能,展示了现实世界数据和全切片建模的重要性。
主要内容¶
计算病理学有潜力通过支持多种临床应用来变革癌症诊断,包括癌症亚型划分、癌症分期、诊断预测和预后预测。尽管现有计算方法的表现令人鼓舞,但这些方法通常是为特定应用开发的,并且需要大量注释数据用于监督学习。数据注释既昂贵又耗时,已成为计算病理学的一个重要瓶颈。近年来,自监督学习在利用未标记数据预训练基础模型方面显示出令人鼓舞的结果,可以大大减少对任务特定注释的需求。由于其强大的泛化能力,基础模型已经在标注数据稀缺但未标记数据丰富的生物医学领域得到了开发,这种情况恰好描述了计算病理学。
病理学基础模型在现实世界临床应用中的发展和使用面临三个主要挑战。首先,公开可用的病理学数据相对稀少且质量参差不齐,这限制了在此类数据上预训练的基础模型的性能。例如,现有的病理学基础模型主要是在癌症基因组图谱(TCGA)的全切片图像(WSI)上进行预训练的,这是一种专家策划的数据集,包含大约 30,000 张切片和 2.08 亿个图像切片。尽管这些数据资源丰富,但 TCGA 数据可能不足以完全解决临床实践中现实世界数字病理学的挑战,如异质性和噪声伪影,导致在使用基于 TCGA 的预测模型和生物标志物时在分布外样本上的性能大幅下降。其次,设计一个能够有效捕捉单个切片中局部模式和整个切片中全局模式的模型架构仍然具有挑战性。现有模型通常将每个图像切片视为独立样本,并将切片级建模形式化为多实例学习,从而限制了它们在千兆像素全切片中建模复杂全局模式的能力。一个显著的例外是层次图像金字塔变换器(HIPT),它探索了切片上的层次自注意力。第三,在少数情况下,在大规模现实世界患者数据上进行了预训练,所得的基础模型通常不向公众开放,从而限制了它们在临床研究和应用中的更广泛适用性。
我们开发了 Prov-GigaPath,一个开放权重的病理学基础模型,以应对这三个挑战(补充表 1)。首先,Prov-GigaPath 是在 Providence 健康网络的 28 个癌症中心的大型数字病理学数据集 Prov-Path 上预训练的。Prov-Path 包含来自 171,189 张 H&E 染色和免疫组织化学病理切片的 1,384,860,229 个图像切片,这些切片来自于超过 30,000 名患者的活检和切除手术,涵盖 31 种主要组织类型。就图像切片的数量而言,Prov-Path 比 TCGA 大五倍以上,就患者数量而言,Prov-Path 比 TCGA 大两倍以上。据我们所知,我们的预训练利用了所有 13 亿个图像切片,构成了迄今为止最大规模的预训练努力。这些大规模、多样化的现实世界数据构成了预训练 Prov-GigaPath 的基础。Prov-Path 还包含了宝贵的信息层次,包括组织病理学发现、癌症分期、基因突变谱以及相关的病理报告。
其次,为了在整个切片中捕捉局部和全局模式,我们提出了 GigaPath,一种用于在千兆像素病理切片上预训练大型病理学基础模型的新型视觉变换器。关键思想是将图像切片嵌入为视觉标记,从而将切片转化为长序列标记。变换器是一种强大的序列建模神经架构,通过提取标记间的任意复杂模式来实现。然而,我们不能直接将传统的视觉变换器应用于数字病理学,因为一个病理切片可能包含数万个切片(在 Providence 数据中多达 70,121 个),并且在变换器中自注意力的计算随序列长度呈二次方增长。为了解决这个问题,我们通过调整我们最近开发的 LongNet 方法来利用扩展的自注意力。预训练首先使用标准视觉变换器和 DINOv2 进行图像级自监督学习,然后使用 LongNet 和掩码自动编码器进行全切片级自监督学习。
最后,为了加速数字病理学研究的进展,我们将 Prov-GigaPath 完全开放,包括源代码和预训练模型权重。
为了系统地研究 Prov-GigaPath 作为病理学基础模型在现实世界场景中的有效性,我们建立了一个综合的数字病理学基准,涵盖 26 个预测任务,如病理组学和癌症亚型划分,使用了 Providence 和 TCGA 的数据。我们将 Prov-GigaPath 与现有的最先进的公开病理学基础模型进行比较,包括 HIPT、CtransPath 和 REMEDIS。结合大规模预训练和超大上下文建模,Prov-GigaPath 在 26 个任务中有 25 个任务上达到了最先进的性能,并在 18 个任务上显著超越了第二好的方法(补充表 2)。例如,在 TCGA 数据集上的 EGFR 突变预测任务中,Prov-GigaPath 在 AUROC 上比第二好的模型 REMEDIS 提高了 23.5%,在 AUPRC 上提高了 66.4%。这尤其值得注意,因为 REMEDIS 是在 TCGA 数据上预训练的,而 Prov-GigaPath 则不是。在癌症亚型划分方面,Prov-GigaPath 在所有九种癌症类型中都优于其他所有模型,并在六种癌症类型中显著优于第二好的方法。这表明它在各种癌症类型中的广泛适用性。最后,我们通过结合每张切片的相关病理报告来探索视觉 - 语言预训练,继续使用视觉 - 语言对比学习对 Prov-GigaPath 进行预训练。我们展示了结果表明 Prov-GigaPath 在标准的视觉 - 语言建模任务中表现出最先进的能力,如零样本亚型划分和突变预测,说明了其在多模态综合数据分析中的潜力。总之,Prov-GigaPath 展示了利用大规模机器学习模型来辅助临床诊断和决策支持的可能性。
Prov-GigaPath 概述¶
Prov-GigaPath 将病理切片中的图像切片作为输入,并输出可用于多种临床应用的切片级嵌入。Prov-GigaPath 通过提取多种局部病理结构和整合整个切片的全局特征,在千兆像素病理切片的长上下文建模中表现出色。Prov-GigaPath 由一个切片编码器和一个切片编码器组成,用于捕捉局部特征和全局特征。切片编码器将所有切片单独投射到紧凑的嵌入中。切片编码器然后输入切片嵌入序列,并使用变换器生成考虑到整个序列的上下文嵌入。切片编码器使用 DINOv2 进行预训练,这是最先进的图像自监督学习框架。切片编码器结合了使用 LongNet 进行掩码自动编码器预训练的方法,这是我们最近开发的用于超长序列建模的方法。在下游任务中,切片编码器的输出使用简单的 softmax 注意力层进行聚合。Prov-GigaPath 是一种适用于高分辨率成像数据的通用预训练方法,可以扩展到其他生物医学问题,包括大规模 2D 和 3D 图像和视频的分析。我们在 Prov-Path 的庞大且多样化的现实世界数据上预训练了 Prov-GigaPath。对于下游任务,预训练的 Prov-GigaPath 使用任务特定的训练数据进行微调,作为使用基础模型的标准。所得的任务特定模型然后可以在给定任务的测试数据上进行评估。与之前最先进的公开病理学基础模型相比,Prov-GigaPath 在 17 个病理组学任务和 9 个亚型划分任务中取得了显著的改进。我们的预训练数据集 Prov-Path 由 1,384,860,229 个 256×256 图像切片组成,这些切片来自于 171,189 张 H&E 染色和免疫组织化学病理切片,涉及超过 30,000 名患者的活检和切除手术,涵盖 31 种主要组织类型。我们总结了人口统计学信息,包括性别、年龄和种族分布(补充表 3-5)以及突变率(补充表 6)。
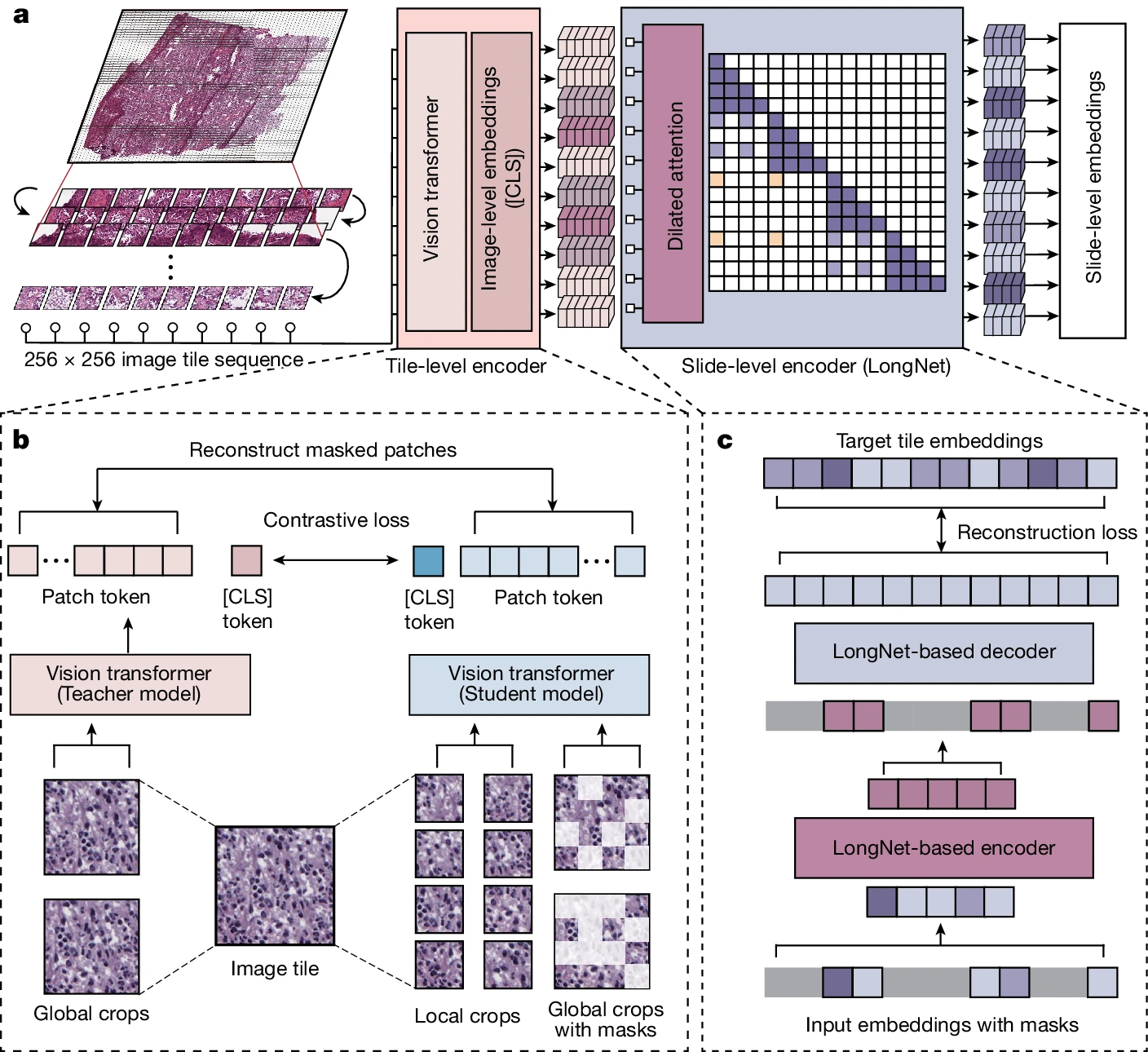
a,显示 Prov-GigaPath 模型架构的流程图。Prov-GigaPath 首先将每个输入的 WSI 序列化为一系列按行优先顺序排列的 256×256 图像切片,并使用图像切片级编码器将每个图像切片转换为视觉嵌入。然后,Prov-GigaPath 应用基于 LongNet 架构的切片级编码器生成上下文嵌入,这些嵌入可以作为各种下游应用的基础 b,使用 DINOv2 进行图像切片级预训练。 c,使用掩码自动编码器和 LongNet 进行切片级预训练。[CLS] 是分类标记。
Prov-GigaPath 改善了突变预测¶
多种功能改变的体细胞基因突变是癌症进展和发展的基础,因此可能在癌症诊断和预后中具有重要作用。尽管测序成本已经大幅下降,但在全球范围内获取肿瘤测序的医疗差距依然存在。因此,从病理图像中预测肿瘤突变可能有助于指导治疗选择并增加个性化医疗的应用。我们将 Prov-GigaPath 与竞争方法在五基因突变预测基准上进行了比较(图 2 和扩展数据图 1-4),将此任务表述为图像分类任务。首先,我们检查了在泛癌设置中最频繁突变的 18 个生物标志物的预测(图 2a、f、l 和扩展数据图 1)。与最佳竞争方法相比,Prov-GigaPath 在这 18 个生物标志物上的宏观接收者操作特征曲线下面积(AUROC)提升了 3.3%,宏观精确率 - 召回曲线下面积(AUPRC)提升了 8.9%。鉴于已知的特定肿瘤突变与整体肿瘤组成和形态之间的关联,我们将这种改进归因于 LongNet 有效捕捉全局图像模式的能力。接下来,我们专注于肺腺癌(LUAD),这是图像突变预测中研究最广泛的癌症类型之一(图 2b、g 和扩展数据图 2)。我们专注于文献中与 LUAD 诊断和治疗密切相关的五个基因(EGFR、FAT1、KRAS、TP53 和 LRP1B)。Prov-GigaPath 表现最佳,平均宏观 AUROC 为 0.626,超过了所有竞争方法(P 值<0.01)。在泛癌分析中,Prov-GigaPath 在这五个基因上也优于最佳竞争方法,宏观 AUROC 提高了 6.5%,AUPRC 提高了 18.7%(图 2c、h 和扩展数据图 3)。
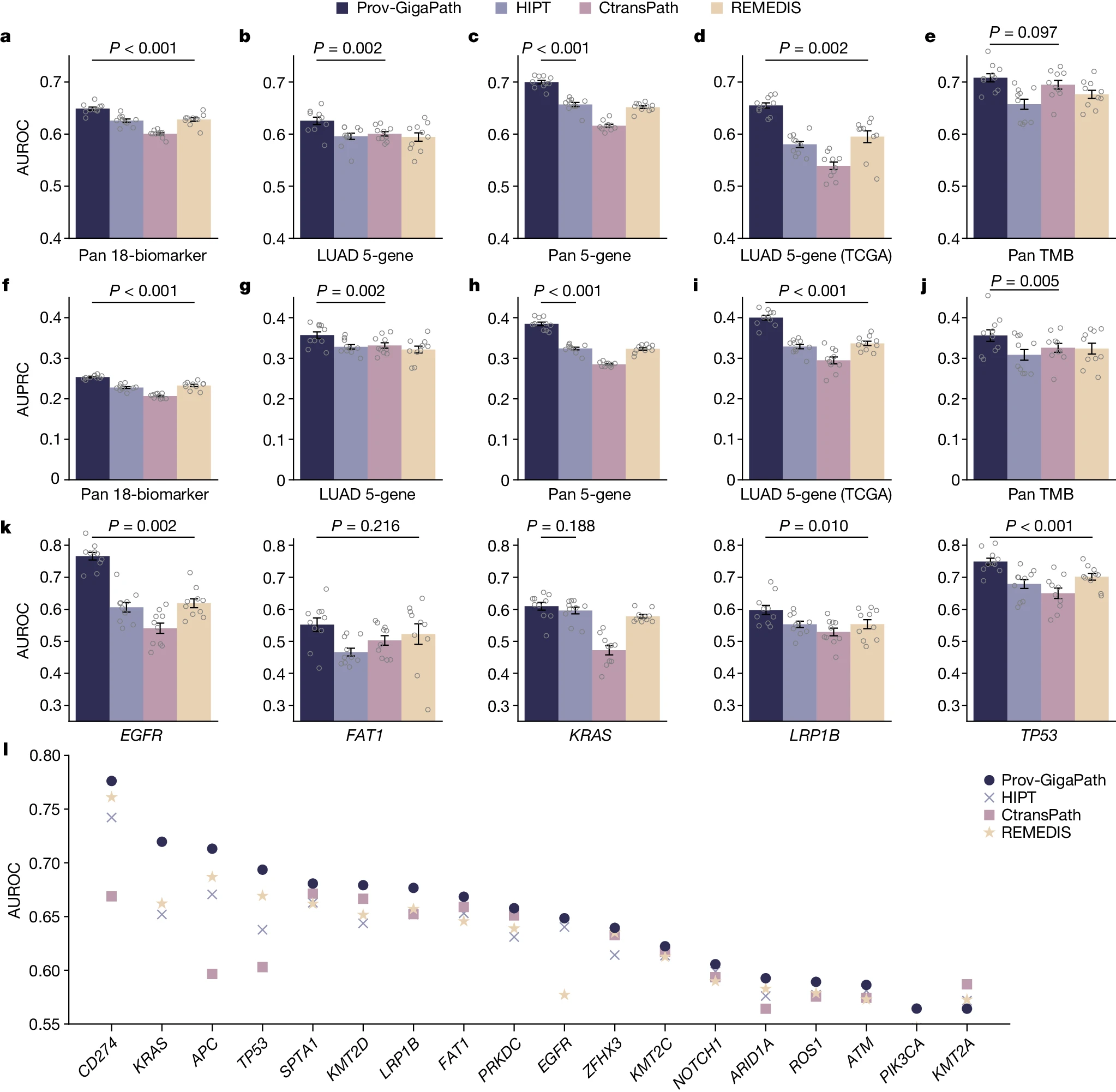
a−j,条形图比较了 Prov-GigaPath 与竞争方法在以下任务中的 AUROC 和 AUPRC 得分:泛癌 18 个生物标志物预测(a,f),LUAD 特异性 5 基因突变预测(b,g),泛癌 5 基因突变预测(c,h),TCGA 中的 LUAD 特异性 5 基因突变预测(d,i)和泛癌肿瘤突变负荷(TMB)预测(e,j)。k,条形图显示了 TCGA 中每个基因在 LUAD 特异性五基因突变预测中的 AUROC。a−k,数据是 10 次独立实验的均值 ± 标准误(s.e.m.)。所列 P 值表示 Prov-GigaPath 优于最佳比较方法的显著性,使用单侧 Wilcoxon 检验。l,比较了泛癌 18 个生物标志物预测中各个生物标志物的 AUROC 得分。
我们还在 TCGA 数据上进行了各方法的对比,以检验 Prov-GigaPath 的泛化能力。我们再次使用 LUAD 特异性五基因突变预测作为关键评估任务(图 2d、i 和扩展数据图 4)。我们观察到 Prov-GigaPath 相对于竞争方法的显著优势,这一点尤为显著,因为所有竞争方法(如 35,41,42)都是在 TCGA 上进行预训练的。为了进一步测试 Prov-GigaPath 的泛化能力,我们从 Providence 收集了一组新的 403 名结直肠癌患者的数据。这些数据是在 2023 年 3 月之后收集的,而用于预训练 Prov-GigaPath 的所有数据是在 2023 年 3 月之前收集的。我们发现,Prov-GigaPath 在这一队列中再次优于竞争方法。我们还注意到,其性能与以前的结直肠癌患者数据没有显著差异(扩展数据图 5)。最后,我们检查了整体肿瘤突变负荷(TMB)的预测,这是固体肿瘤中特别与免疫治疗相关的预测生物标志物。Prov-GigaPath 以平均 AUROC 0.708 的最佳性能显著超过了第二好的方法(图 2e, j)。
我们观察到,在测试 TCGA 中的 LUAD 特异性五基因突变时,基于 Prov-Path 预训练的 GigaPath 相比于基于 TCGA 数据预训练的同一模型架构取得了显著提升,这表明了 Prov-Path 的高质量(扩展数据图 6)。我们进一步发现,当两者都在 Prov-Path 上训练时,GigaPath 优于 HIPT,这表明了 GigaPath 框架的有效性(扩展数据图 7 和 8)。为了进一步评估我们方法的预训练策略,我们观察到使用 DINOv2 预训练比使用基于对比学习的 SimCLR26 和掩码自动编码器 45 预训练效果更好(补充图 4),这表明我们的预训练策略的有效性。Prov-GigaPath 还优于使用 ImageNet 训练模型的监督学习方法,这证明了我们自监督学习框架的必要性(补充图 4)。
总体而言,Prov-GigaPath 在各种病理组学任务上显著优于之前最先进的病理学基础模型。我们假设这种显著的改进反映了我们全切片建模中的差异化优势。
Prov-GigaPath 改善癌症亚型划分¶
鉴于病理图像在分配肿瘤亚型中的整体效用,我们接下来检查了 Prov-GigaPath 是否能够准确预测图像中的癌症亚型。我们在 Prov-Path 中的九种主要癌症类型的亚型划分上评估了我们的方法(图 3)。Prov-GigaPath 在所有九种癌症类型中都优于所有竞争方法,并在六种癌症类型上显著优于第二好的方法,这表明我们的切片编码器和全切片编码器协同工作以提取区分微小病理模式的有意义特征。HIPT 与 Prov-GigaPath 之间的一个关键区别在于图像切片嵌入的聚合层。Prov-GigaPath 相对于 HIPT 的显著改进展示了使用 LongNet 在全切片中高效且有效地聚合超大图像切片集合的潜力。
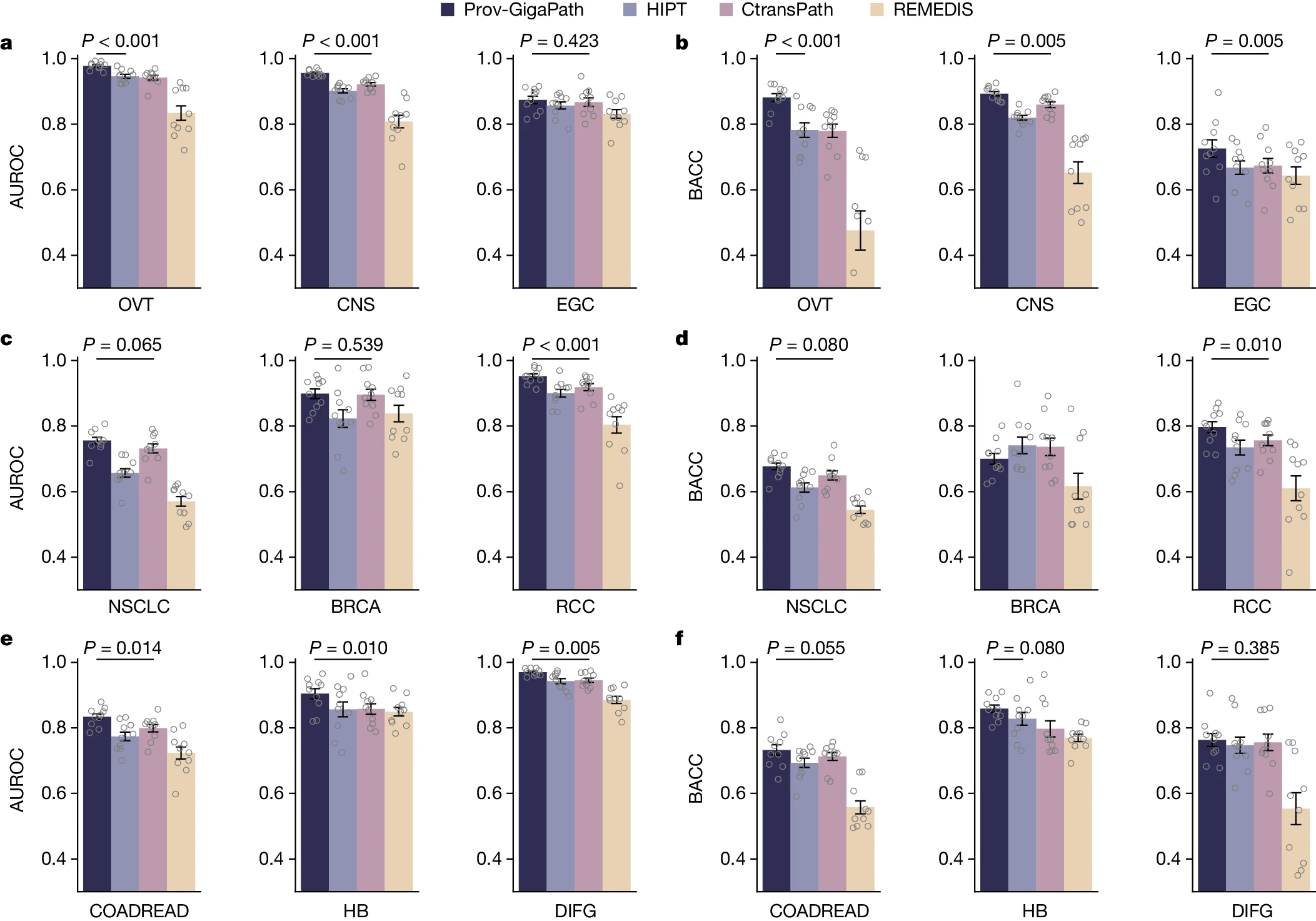
a–f,条形图比较了九种癌症类型的亚型划分性能,以 AUROC(a、c、e)和平衡准确率(b、d、f)衡量。数据是 10 次独立实验的均值±标准误(s.e.m.)。所列 P 值表示 Prov-GigaPath 优于最佳比较方法的显著性,使用单侧 Wilcoxon 检验。BACC,平衡准确率。BRCA,乳腺浸润性癌;CNS,中枢神经系统;COADREAD,结直肠腺癌;DIFG,弥漫性内在桥脑神经胶质瘤;EGC,早期胃癌;HB,肝胆;NSCLC,非小细胞肺癌;OVT,卵巢癌;RCC,肾细胞癌。
最后,我们进行了消融研究,以系统评估 Prov-GigaPath 各组件对其在癌症亚型划分性能的贡献(补充图 5)。为了检验 LongNet 预训练的重要性,我们将 Prov-Path 上预训练的 LongNet 编码器替换为随机初始化的模型。我们观察到平均 AUROC 从 0.903 大幅下降到 0.886(P 值<2.0×10^-3),这表明预训练的 LongNet 编码器能够更好地捕捉切片级的癌症异质性。我们观察到冻结和解冻 LongNet 编码器在癌症亚型划分任务上表现相当,这表明我们的预训练方法可以有效学习高质量表示,减少了对 LongNet 进一步微调的需求。为了验证使用 LongNet 编码器在整个切片上聚合图像模式的优越性,我们测试了一个替代方法,通过移除 LongNet 并仅通过基于注意力的深度多实例学习(ABMIL)层进行聚合。平均来看,ABMIL 层无法达到与 LongNet 相似的切片编码器性能(P 值<0.012),这证实了在病理切片中建模长程依赖性的必要性。
切片级视觉 - 语言对齐¶
Prov-GigaPath 在病理图像上的令人鼓舞的结果进一步激励我们在多模态视觉 - 语言处理方面探索 Prov-GigaPath。以往在病理学视觉 - 语言建模的工作往往专注于病理图像和文本的切片级对齐,因为他们的研究受到图像 - 文本对的来源限制(例如教科书示例或 Twitter 数据)。相比之下,我们通过利用每个切片的相关报告,检查了病理图像和文本的切片级对齐(图 4a)。这种自然出现的切片 - 报告对可以潜在地揭示更丰富的切片级信息,但建模相当具有挑战性,因为我们没有单个图像切片和文本片段之间的细粒度对齐信息。我们在 Prov-GigaPath 的持续预训练中使用标准的跨模态对比损失作为视觉编码器,并使用最先进的生物医学语言模型 PubMedBERT 作为文本编码器(图 4b)。
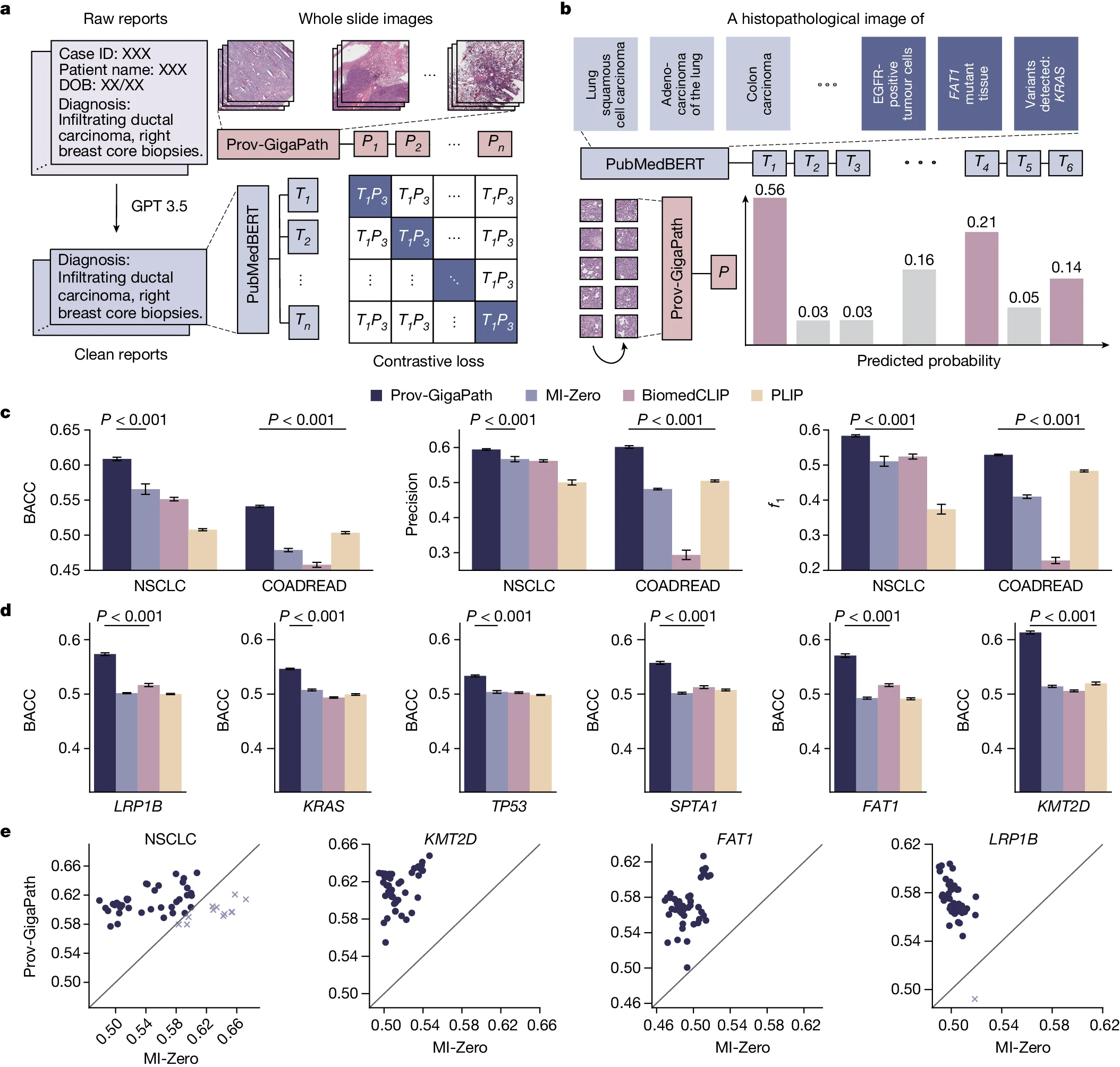
a, 流程图显示了使用病理报告对 Prov-GigaPath 进行微调的过程。使用 OpenAI 的 GPT-3.5 处理真实世界的病理报告,去除与癌症诊断无关的信息。我们进行了基于 CLIP 的对比学习,以对齐 Prov-GigaPath 和 PubMedBERT。 b, 微调后的 Prov-GigaPath 可以用于零样本癌症亚型划分和突变预测。Prov-GigaPath 的输入是一系列从 WSI 分割出的图像切片,文本编码器 PubMedBERT 的输入是手动设计的代表癌症类型和突变的提示词。基于 Prov-GigaPath 和 PubMedBERT 的输出,我们可以计算输入的 WSI 被分类为特定癌症亚型和突变的概率。 c, 条形图比较了在 NSCLC 和 COADREAD 上的零样本亚型划分性能,以 BACC、精度和 F1 值衡量。 d, 条形图比较了微调模型在六个基因上的突变预测性能。 c, d, 数据为 50 次实验的均值±标准误(s.e.m.)。所列 P 值表示 Prov-GigaPath 优于最佳比较方法的显著性,使用单侧 Wilcoxon 检验。 e, 散点图比较了 Prov-GigaPath 和 MI-Zero 在零样本癌症亚型划分中的 BACC 表现。每个点表示具有特定文本查询公式的一次试验。
我们在 NSCLC 和 COADREAD 的零样本癌症亚型划分中评估了 Prov-GigaPath,使用了与最先进的病理学视觉 - 语言模型 MI-Zero 相同的设置。在零样本设置中,没有为任何目标癌症亚型提供训练图像。切片和相应的癌症亚型从 Prov-Path 中收集。与三种最先进的病理学视觉 - 语言模型相比,Prov-GigaPath 在两种癌症类型的所有三个指标上都达到了最佳的零样本分类结果(图 4c,e,扩展数据图 9 和补充图 6),这表明 LongNet 支持的切片级对齐确实具有优势。Prov-GigaPath 在 NSCLC 上的改进比在 COADREAD 上的改进更大,这可以归因于在 Prov-Path 中肺组织的更普遍存在。Prov-GigaPath 以显著优势超越了 PLIP,这可能反映了现实世界临床数据优于 Twitter 数据的优势。
接下来,我们在相同的零样本设置中检查了使用视觉 - 语言预训练 Prov-GigaPath 预测基因突变的可能性(图 4d,e 和扩展数据图 9)。我们采用了用于癌症亚型划分的提示词,并将癌症类型名称替换为我们想要预测的二进制突变状态的基因名称。Prov-GigaPath 在我们研究的所有六种突变中大幅超越了最先进的病理学视觉 - 语言模型(P 值<0.001)(图 4d,e)。我们的方法在突变预测上的改进比在癌症亚型划分上的改进更大,这可能部分归因于现实世界数据中的病理报告中包含了更丰富的突变信息,而不是 Twitter8 和科学论文 50 中的文本评论。据我们所知,这是第一次在病理学视觉 - 语言建模中评估零样本基因突变预测。Prov-GigaPath 在这一新任务上的出色表现预示着在研究罕见癌症类型和新突变方面的潜在应用。
讨论¶
我们介绍了 Prov-GigaPath,这是一种用于广泛数字病理学应用的病理学基础模型。Prov-GigaPath 在从 Providence 健康系统派生的现实世界大数据集 Prov-Path 上进行了预训练,该数据集类型和质量多样。Prov-Path 比 TCGA 大得多,包括来自约 30,000 名患者的 171,189 张全病理切片中的 1,384,860,229 个图像切片。我们提出了用于预训练的 GigaPath,它采用了最先进的 LongNet5 作为视觉变换器,以促进千兆像素 WSI 的超大上下文建模。在对 Providence 和 TCGA 数据集的综合评估中,我们展示了 Prov-GigaPath 在各种病理组学和癌症亚型划分任务以及视觉 - 语言处理方面的最先进性能。Prov-GigaPath 有潜力辅助临床诊断和决策支持,GigaPath 可以广泛应用于高分辨率图像的高效自监督学习,适用于更广泛的生物医学领域。
我们注意到,不同任务中我们方法的性能有显著差异。首先,亚型划分的性能显著优于突变预测的性能。尽管由于训练样本数量的不同,不同任务不可比较,但我们的观察表明基于图像的突变预测更具挑战性。一个特别的原因可能是病理图像信息不足以预测某些突变。因此,我们计划在未来利用其他模式和特征来增强预测。尽管如此,我们的方法在突变预测任务上优于现有方法,为改进诊断和预后提供了机会。此外,我们发现基础模型(包括我们的方法和竞争方法)比任务特定模型(例如补充图 4 中的 SL-ImageNet)更有效,这表明这些基础模型中自监督学习框架的必要性。我们目前在预处理中选择了 20 倍放大倍率。更高的放大倍率将使处理时间增加四倍,但也会揭示更多图像细节。因此,我们有兴趣在未来探索其他放大倍率。在对文本数据建模时观察到了大语言模型中的缩放规律。我们观察到,基于较大 Prov-Path 数据预训练的 GigaPath 优于基于较小 TCGA 数据预训练的 GigaPath(扩展数据图 6)。尽管有不同的模型架构,我们还观察到参数更多的 GigaPath 在两者都在 Prov-Path 上预训练时优于 HIPT。这两个结果表明,较大的预训练数据和较大的模型的有效性,部分表明随着更多预训练标记,模型性能可能会进一步提高。我们有兴趣在病理学基础模型的背景下,通过比较不同大小的模型和不同大小的预训练数据来进一步验证缩放规律。
虽然初步结果令人鼓舞,但还有很多发展机会。首先,通过比较使用不同尺寸的视觉变换器的性能,研究病理学基础模型的缩放规律将是有趣的。特别是,我们发现使用 2300 万个参数的小版本 Prov-GigaPath 也达到了优于现有方法的性能,这表明两种模型在现实世界诊所中的应用:一个用于快速推理和微调的小模型,和一个用于更准确推理的大模型(Prov-GigaPath)。其次,预训练过程可以进一步优化。在切片级自监督学习中,我们在预训练切片级编码器时冻结了切片级编码器,以减少内存成本,这可能并不理想。我们计划在更大的 GPU 集群上探索端到端预训练,在这些集群上我们可以即时计算图像编码,并进行全面微调。第三,我们在视觉 - 语言预训练上进行了初步探索,并在零样本亚型划分和突变预测中展示了令人鼓舞的结果,但这仍然远未达到作为临床医生对话助手的潜力。未来,我们计划将先进的多模态学习框架(如 LLaVA-Med52)纳入我们的工作。
方法¶
预处理 WSIs¶
我们首先建立了对 171,189 张 H&E 染色和免疫组化病理切片的预处理流程。各器官的切片和患者统计数据如补充图 1 和 2 所示。首先,我们进行组织分割以过滤背景区域。按照 HIPT 的方法,我们在下采样分辨率(例如 1,024 像素)下运行 Otsu 图像阈值分割算法,以其计算效率和在区分组织和背景方面的有效性。其次,我们使用 pyvips 库将 WSIs 调整到每像素 0.5 μm(即 20×放大倍率)的标准分辨率。这一步是必要的,因为某些切片根据扫描仪设置具有更高的分辨率。最后,将图像裁剪为 256×256 像素的图像切片。通过 Otsu 算法确定占用值小于 0.1 的切片被丢弃,以集中关注覆盖组织的区域。我们在一个最多可容纳 200 个节点的集群上执行这些操作,每个节点配备 32 个 CPU 内核和 256 GB RAM,预处理大约在 157 小时内完成。任务被并行化,每个节点独立处理一组切片。最终,我们总共收集了 1,384,860,229 个切片,每个 WSI 中的切片数量如补充图 3 所示。
Prov-GigaPath 预训练的详细信息¶
Prov-GigaPath 切片编码器使用 ViT 模型架构,采用标准的 DINOv2 设置。我们在 1,384,860,229 个分割切片上对模型进行预训练,将每个切片视为一个数据实例。DINOv2 预训练的基本学习率设置为 4×10^-3。我们将每个 GPU 设备上的批量大小设置为 12,总有效批量大小为 384。Prov-GigaPath 切片编码器使用 LongNet 模型架构,采用标准设置。为了离散化切片坐标,我们将网格大小 dgrid 设置为 256,将行和列的数量 ngrid 设置为 1,000。对于输入序列的增强,我们将裁剪比率设置为 0.875。移动距离是随机生成的,使用均匀分布保持所有切片在创建的网格覆盖范围内。我们以 0.5 的概率水平翻转每个切片的坐标。为了使用掩码自动编码器对 Prov-GigaPath 切片编码器进行预训练,我们将学习率设置为 5×10^-4,每个 GPU 设备上的批量大小设置为 4。我们还将训练周期设置为 30,初始周期作为预热阶段。切片编码器预训练利用 16 个节点,每个节点配备 4×80 GB A100 GPU,预训练大约在 2 天内完成(3,072 A100 GPU 小时)。对一个 WSI 的推理时间平均为 0.7 秒,包括计算切片嵌入的 0.4 秒和 LongNet 推理的 0.3 秒。
竞争方法和基准测试¶
我们将 Prov-GigaPath 与 4 种比较方法进行了比较。HIPT 是一个预训练模型,在 TCGA 的 10,678 张千兆像素 WSI 上进行了预训练。它采用了分层图像金字塔变换器架构,具有 256×256 和 4,096×4,096 的图像视图。我们也可以将 HIPT 模型视为一个切片编码器,在 4,096×4,096 视图上有一个额外的嵌入聚合编码器。由于它使用 DINO 自监督学习方法训练 256×256 图像编码器和 4,096×4,096 图像编码器,因此 HIPT 的切片编码器预训练与 Prov-GigaPath 相同。HIPT 和 Prov-GigaPath 之间的主要区别在于聚合机制。Prov-GigaPath 使用带有切片编码器的长序列表示学习进行聚合,而 HIPT 在 4,096×4,096 图像视图上使用第二阶段的 ViT 进行聚合。CtransPath\({}^{41}\) 结合了 CNN 模型和多尺度 SwinTransformer。CtransPath 使用语义相关的对比学习目标预训练模型,将每个输入图像及其增强视图视为正样本对,并将 \(\mathcal{S}\) 检索到的语义相关图像作为伪正样本对。REMEDIS\({}^{42}\) 使用 Resnet 作为骨干网络,并在从 29,018 张 TCGA 切片中随机采样的 5000 万病理图像上使用 SimCLR 方法进行预训练。在我们的实验中,我们选择了 Resnet 152×2 模型进行评估。
我们在不同的下游任务上微调了 Prov-GigaPath 和其他基线模型。对于 Prov-GigaPath,我们冻结了切片编码器,仅微调 LongNet 切片级编码器。对于每个切片,LongNet 生成一组上下文化的切片嵌入。这些嵌入通过浅层 ABMIL 层聚合,得到切片嵌入,然后在下游预测任务的附加分类器中使用。在应用 HIPT 模型时,我们按照默认设置冻结了 256×256 和 4,096×4,096 图像编码器,并调整额外的变压器层和 ABMIL 层的参数。由于 CtransPath 和 REMEDIS 都是切片级编码器,我们直接应用一个 ABMIL 层来获取切片级嵌入,并主要调整 ABMIL 层和分类器。
突变预测¶
从 Prov-Path 中,我们构建了 5 个基因突变预测任务:泛癌 18 个生物标志物预测、LUAD 5 个基因突变预测、泛癌 5 个基因突变预测、TCGA 中的 LUAD 5 个基因突变预测和整体 TMB 预测(补充表 7 和 9)。18 个生物标志物预测是一个 18 类多标签分类问题,每个类别是一个突变或 PD-L1。每个基因的阳性状态表示其发生突变或 PD-L1(由 CD274 编码)高表达。5 个基因突变预测任务是 5 类分类问题。包括 EGFR、FAT1、KRAS、TP53 和 LRP1B 的 5 个基因突变预测任务被表述为多标签预测任务,模型被要求预测所有基因的突变状态。整体 TMB 预测是一个 2 类分类(高 TMB 对低 TMB)。我们将此任务表述为图像二元分类任务,其中每个图像根据肿瘤的体细胞突变数量被标注为“高 TMB”和“低 TMB”。这些评估反映了模型提取 WSIs 上不同分子模式的能力。对于每个通常具有多个 WSI 的患者,我们选择最大的 WSI。这在将数据集划分为训练、验证和测试集时自然启用了患者级别的分层。我们以基础学习率 2×10^-3 和权重衰减 0.01 微调了 Prov-GigaPath 模型。按照 HIPT 的默认设置,我们以学习率 2×10^-4 训练了比较模型。所有方法的训练批量大小设置为 1,梯度累积步数为 32。我们训练了所有方法 20 个周期。性能通过 10 折交叉验证使用 AUROC 和 AUPRC 评估。
癌症亚型划分¶
我们对九种癌症类型进行了亚型划分评估,包括 NSCLC(LUAD 对 LUSC)、BRCA(IDC 对 ILC)、RCC(CCRCC 对 PRCC 对 CHRCC)、COADREAD(COAD 对 READ)、HB(CHOL 对 HCC)、DIFG(GBM 对 ODG 对 AODG 对 HGGNOS 对 AASTR)、OVT(CCOV 对 EOV 对 HGSOC 对 LGSOC 对 MOV 对 OCS)、CNS(ATM 对 MNG)和 EGC(ESCA 对 GEJ 对 STAD);详情和定义见补充表 8 和 9。我们以基础学习率 4×10^-3、权重衰减 0.001 和层级学习率衰减 0.9 微调了 Prov-GigaPath。训练超参数基于验证集性能选择。所有模型均微调 20 个周期,并使用 10 折交叉验证进行评估。对于 Prov-GigaPath,我们另外在切片级编码器中添加了一个快捷方式,以更多地关注切片级特征。
视觉 - 语言对齐¶
我们构建了 17,383 对病理 WSI- 报告对,并使用 OpenCLIP 代码库进行视觉 - 语言处理。由于现实世界的病理报告杂乱且冗长,我们首先通过去除与癌症诊断无关的信息(包括医院位置、医生姓名和患者姓名)来清理原始病理报告。具体来说,我们首先使用 k-means 将临床报告聚类为四个簇,并选择簇中心作为四个代表性报告。然后,我们手动清理这四个报告,并获得四对原始和清理后的报告。我们使用这四个报告作为上下文学习示例,并要求 GPT-3.5 根据这四个上下文学习示例清理所有其他报告(补充图 9)。过滤前后的整体标记长度分布如补充图 10 所示。使用 OpenAI 的 text-embedding-ada-002 模型计算文本嵌入。最终,我们构建了 17,383 对 WSI 和清理后的报告的视觉 - 语言对。我们保留 20% 的患者用于 CLIP 预训练的零样本预测任务。我们将 CLIP 训练的学习率设置为 5×10^-4,批量大小为 32。我们训练视觉编码器和文本编码器 10 个周期,前 100 次迭代为预热阶段。
在零样本预测任务中,我们选择了 MI-Zero(PubMedBERT)7、BiomedCLIP50 和 PLIP8 作为比较模型。MI-Zero(PubMedBERT)在来自教育资源和 ARCH 数据集的 33,480 对病理图像 - 字幕对上训练。它是一种基于多实例学习的零样本转移方法,通过 top K 池化策略聚合多个切片。BiomedCLIP 在 1500 万对生物医学领域特定的图像 - 字幕对上训练。PLIP 是一个使用 Twitter 上的图像 - 文本对进行预训练的病理学领域特定视觉 - 语言模型。我们在 NSCLC 和 COADREAD 亚型划分任务以及 LRP1B、KRAS、TP53、SPTA1、FAT1 和 KMT2D 突变状态预测上评估了比较方法和 Prov-GigaPath。我们遵循 MI-Zero7 中的设置和提示模板,并使用 50 个随机抽样的提示集评估这些方法。
报告摘要¶
关于研究设计的更多信息,请参见链接到本文的《Nature Portfolio》报告摘要。
数据可用性¶
用于预训练的病理成像数据来自 Providence 的肿瘤病理切片。用于微调和测试的相关临床数据来自相应的医疗记录。这些专有数据无法公开提供。研究人员可以通过合理请求并经过当地和国家伦理批准,从 Providence 健康系统获得一个去标识的测试子集。为了帮助研究人员使用我们的模型,我们在 https://doi.org/10.5281/zenodo.10909616(参考文献 57)和 https://doi.org/10.5281/zenodo.10909922(参考文献 58)提供了一些患者的去标识数据子集。我们还从 NIH 基因数据共享门户收集了公开可用的 TCGA WSI。TCGA-LUAD 数据集,包括完整的病理切片和标签,可以通过 NIH 基因数据共享门户 https://portal.gdc.cancer.gov/projects/TCGA-LUAD 获取。
代码可用性¶
Prov-GigaPath 是一个视觉变换器模型,通过使用 DINOv2 在切片级预训练,然后使用掩码自动编码器和 LongNet 在切片级预训练,处理超过 170,000 张全切片和超过十亿个病理图像切片。在预训练之前,这些病理切片已去除了识别条形码。Prov-GigaPath 可以通过 https://github.com/prov-gigapath/prov-gigapath 访问,包括模型权重和相关源代码。我们在方法和补充信息中包含了详细的方法和实施步骤,以便独立复制。