Disentanglement of single cell data with biolord
摘要¶
Biolord 是一种深度生成方法,用于解析单细胞多组学数据中已知和未知的属性,包括空间、时间和疾病状态。此方法可以揭示不同单细胞方式和生物系统中的解耦生物标志。通过在虚拟环境中转换细胞状态,Biolord 能生成实验上无法获取的样本,其在预测细胞对未见过的药物和基因扰动的反应方面,超越了现有的最先进方法。Biolord 的资源可以在以下链接获取:https://github.com/nitzanlab/biolord。
正文¶
细胞的基因表达谱同时编码了多个属性的信息,如细胞类型、来源组织和分化阶段(见图 1a)。单细胞技术能够为单个细胞提供这类表达谱信息。然而,解码所测量的基因表达并区分各个过程仍然是一个主要挑战。一个解耦的表征能够揭示多样化生物过程的存在和特征,允许重建细胞身份的多个属性,如对扰动的反应和感染进程。早期研究建议使用因子分析(引用 1、2)或非负矩阵分解(引用 3)来识别与不同属性相关的程序。最近,针对特定任务的计算方法被提出来进行解耦;这些任务包括解耦扰动响应(引用 4 至 8)、解构特定群体属性(引用 9)或对单细胞数据进行分布外采样(引用 10、11)。然而,这些方法要么是针对特定任务并没有解决一般解耦问题,要么依赖于线性和独立性假设,不能整合超出单细胞测量的多种类型信息,或者没有提供一般的重建程序。
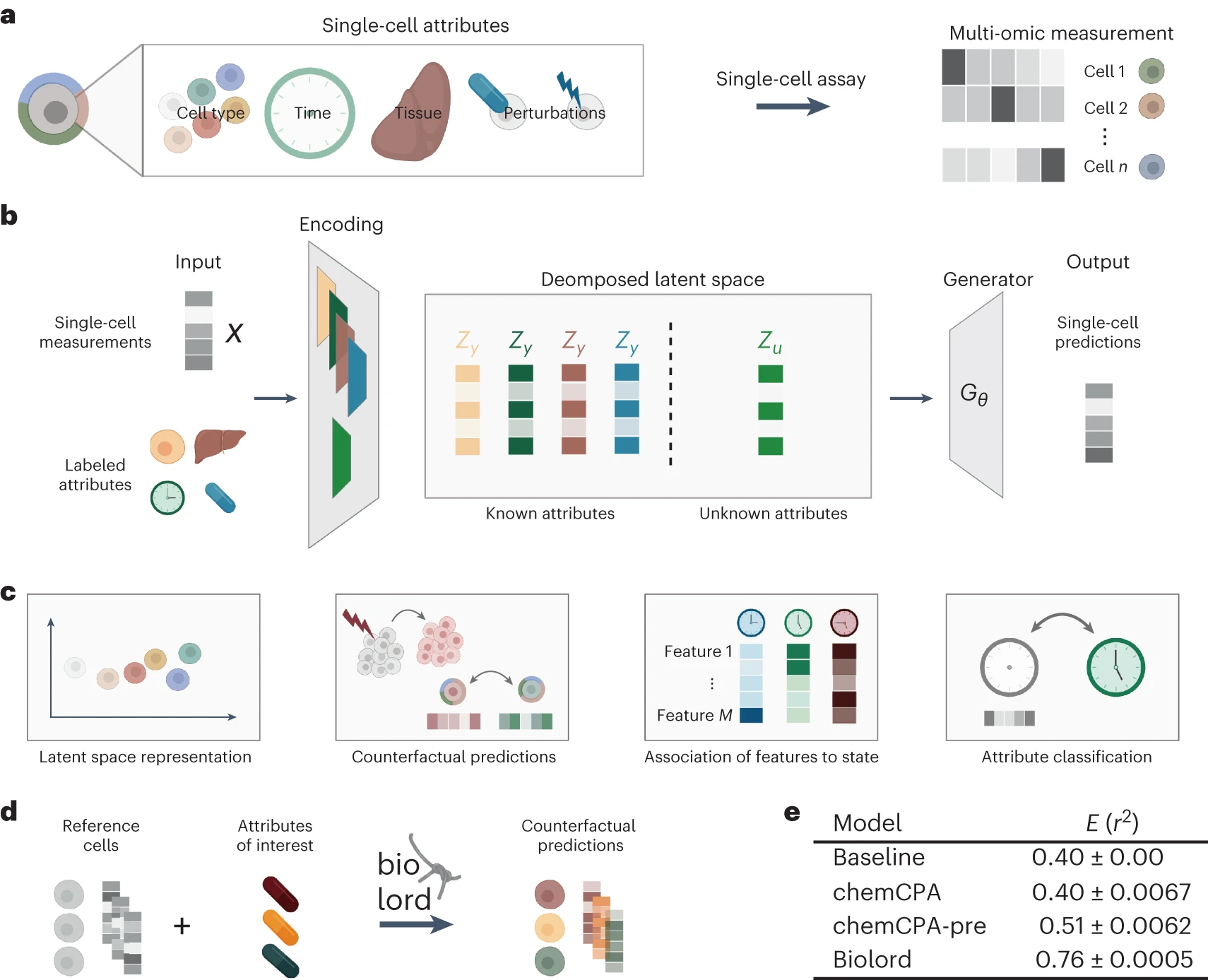
图 1 a, 单细胞数据编码细胞身份的多个属性。 > b, Biolord 模型的示意概览; 给定单细胞测量和观察到的属性标签,Biolord 分别对每个属性进行编码,同时对未知属性进行单一编码。这些定义了一个分解的潜在空间,作为生成模块的输入,提供测量预测。 c, Biolord 可用于多种下游任务。 从左到右—潜在空间表征:分解的潜在空间可用于获取个别属性底层结构的洞察。反事实预测:给定一个对照细胞和未见(目标)标签作为输入,Biolord 可以预测未见细胞状态的基因表达,并研究对应于细胞属性操作的基因表达变化。特征与状态的关联:通过操作已知属性,Biolord 可以识别与不同可能状态相关的测量特征,例如,通过将对照细胞操作为感染状态并识别与感染相关的基因。属性分类:使用半监督的 Biolord 架构,可以为细胞标记缺失的属性。 d, 获取反事实预测的示意概览。 我们以一组参考细胞的测量数据作为输入,这些细胞对于我们进行预测的属性有不同的分配。例如,我们以对照细胞和多种药物作为输入,生成反事实预测,预测如果给予每种药物,这些细胞的基因表达谱将如何变化(方法)。 e, Biolord 在预测未见药物方面的性能评估,使用的是包含约 650,000 个单细胞转录组的 sci-Plex 3 数据集,这些转录组来自三个癌症细胞系,暴露于 188 种化合物 16。 结果报告了 10 微摩尔剂量的情况,这被认为是最严格的设置,因为测量显示与对照状态的最大偏差,这使得它们最难预测。报告了每个模型不同随机种子初始化的平均值和方差。图版 a-d 由 BioRender.com 创建。
在机器学习中,解耦方法将世界视为由未知的前向过程生成,该过程将生成因子(属性)映射到可观察的数据中。例如,一张汽车的图像是由多个属性如型号和姿态生成的。解耦的目标是逆转这一过程,例如,将汽车图像映射到代表其型号和姿态的变量中。然后可以使用解耦表示进行数据操作,生成型号和姿态的未见组合。类似地,在生物环境中,给定标记的单细胞数据,例如细胞类型和年龄注释(已知属性),解耦表示将已知属性,如细胞类型和年龄,与未知属性分离。未知属性对应于细胞特定的标志,例如,与批次效应、生物噪音或未分类的生物过程相关。解耦表示可用于数据生成、操纵和派生生物学洞察(例如,预测未观察到的细胞类型和年龄组合的测量特征或识别某些细胞类型或状态的驱动基因)。
利用计算机视觉领域最近的进展,我们提出了 Biolord(生物表示解耦),这是一个用于学习单细胞数据中解耦表示的深度生成框架。为了将单细胞数据解耦为其基础属性,我们假设一个训练集,包括每个具有部分监督的已知属性的单细胞测量,例如,已知属性可能是细胞类型标签、测量时间或扰动值;属性可能是分类的(离散的;例如,细胞类型)或有序的(连续的;例如,年龄)。在部分监督下,Biolord 找到一个解耦的潜在空间,包括每个已知属性的嵌入和数据中剩余未知属性的嵌入。在这些之上,Biolord 学习一个生成器,将已知和未知属性的表示映射到可观察的单细胞数据中。它反过来可以使用解耦的潜在空间来预测不同细胞状态在内部或外部条件变化下的单细胞测量。通过引入信息约束,获得成功的解耦;模型的损失函数试图最大化重建的准确性(强制完整性)同时最小化未知属性中编码的信息(限制其容量)。我们修改了原始的图像分析框架,以适应单细胞数据的特点,并通过架构和设计选择进行调整。此外,我们提出了框架的一个扩展,Biolord-classify,它可以应用于具有部分标记属性的数据集,并为缺失标签提供分类。
该框架的通用性使其可以应用于可以通过丰富的下游分析任务研究的多种生物环境。利用模型的生成方面,我们可以进行反事实预测,预测未见的细胞状态并进行数据操纵。在预测对未见药物或基因扰动的反应方面,Biolord 超过了专门用于此任务的最先进方法。分解的潜在空间表示允许独立研究不同属性及其内部结构。例如,这种表示揭示了人类胎儿染色体图谱中组织、样本估计的概念后年龄和细胞类型属性之间的关系。此外,我们可以将测量特征与细胞状态关联。最后,Biolord 可以应用于部分标记的数据集,并用于获得整个数据集的标记(属性分类)。我们将这一应用于一个时空疟疾感染图谱,以完成一个独特状态的缺失分类(最初仅为最新时间点提供),从而允许我们研究向感染状态的瞬态轨迹。我们使用 scvi-tools 库实现了 Biolord,并在 https://github.com/nitzanlab/biolord 提供了它。
Biolord 准确预测细胞扰动响应¶
准确预测对药物或基因扰动的分子响应对于我们理解细胞行为和转化医学至关重要。因此,许多计算工具致力于这项任务。在这些工具中,有的是用于药物反应预测的 chemCPA,用于基因扰动的 GEARS,以及同时处理这两者的 PerturbNet。细胞响应预测可以被框定为一个解耦任务,旨在从基础细胞状态中解耦扰动响应,因此可以通过 Biolord 来接近。在药物反应预测任务中,我们使用 sci-Plex 3 数据集,该数据集包括来自三个癌症细胞系的约 650,000 个单细胞转录组,这些细胞暴露于 188 种化合物,并进行了四种不同剂量和对照样本的测量。
为了使对未见药物进行概括,我们利用现有的先验知识,使用 RDKit 特征获得药物的化学信息嵌入。对于每个细胞,将每种药物的特征及其剂量、细胞系和相应的单细胞 RNA 测序测量值作为输入给 Biolord。Biolord 学到的潜在表示在生物学上具有信息量;它揭示了根据已知相应途径的药物组织,并在定性和定量上更好地捕捉了与作为输入的化学信息 RDKit 特征相比的基础药物组织。为了进一步评估 Biolord 的药物表示质量,我们使用参考文献中建议的不确定性度量,评估从嵌入空间的 k- 最近邻 (k-NN) 图预测药物途径的能力。与 RDKit 相比,Biolord 的不确定性度量平均值更低,分布更集中。
我们使用训练好的 Biolord 模型获得九种未见药物的反事实预测。具体来说,我们为控制细胞生成了未见化合物的标签的表达预测。使用 r2 分数评估实际暴露于未见化合物的细胞的实际测量与反事实预测之间的性能。Biolord 的表现超过了一个简单的基线(将未见化合物的实际测量与对照测量进行比较),以及最先进的模型,如 chemCPA 和 chemCPA-pre。尽管没有提供 chemCPA-pre 使用的附加信息,Biolord 提供了更准确的预测。Biolord 也超过了 PerturbNet,并在数据子样本上保持高预测准确性。
反事实预测揭示感染基因程序¶
随着时空单细胞图谱的不断扩展,每个图谱都捕捉到了复杂的生物环境。在计算挑战中,解耦多样的属性,从而将测量特征与不同的细胞状态关联起来,是一项重要任务。聚焦于在小鼠肝脏中疟原虫感染进展的时空单细胞图谱,我们展示了 biolord 如何获得一个解耦表示,该表示有助于揭示与感染相关的属性。包括宿主和寄生体转录组的单细胞数据,从感染后的小鼠中在五个时间点(感染后 2、12、24、30 和 36 小时)收集,以及从未暴露于寄生体的对照小鼠中收集(对照;图 2a 和扩展数据图 3)。为了将肝细胞分类为感染或未感染,作者依赖于寄生体转录组中的 GFP 含量(图 2b)。使用 biolord,我们的目标是将宿主肝细胞中由感染引起的基因表达变化与之前已建立的时空过程中的变异性解耦——无论是沿肝小叶径向轴的空间分区还是沿一天中时间的时间变化(图 2a 和扩展数据图 3)。
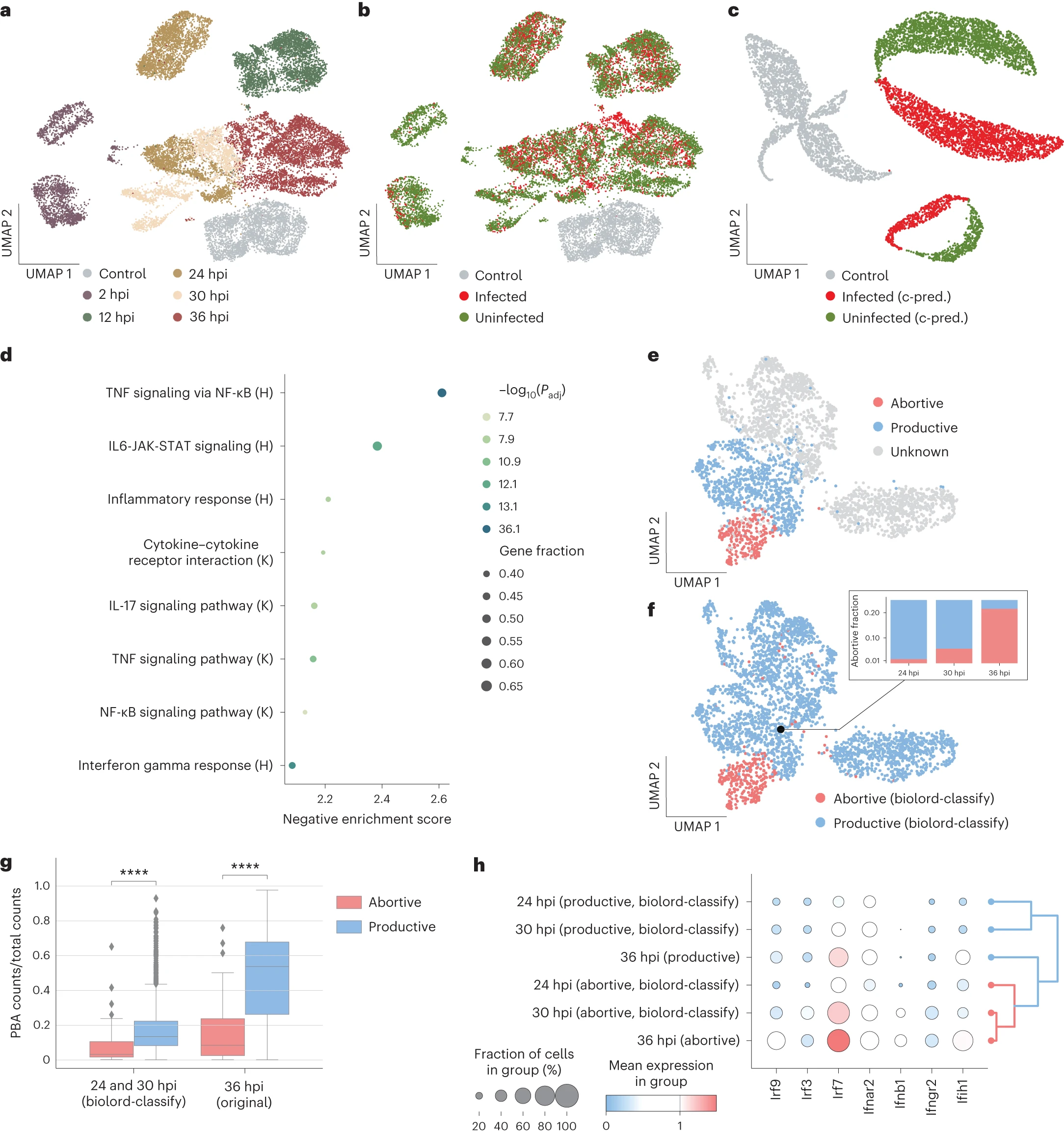
a,b, 疟原虫肝阶段单细胞图谱的 UMAP 显示 细胞根据感染后的时间着色(a)以及感染/未感染及对照细胞的报告分类(b)。 c, 原始对照细胞及其对感染/未感染状态的反事实预测(c-pred)的 UMAP; 细胞按相应状态着色。 d, 基于 biolord 对对照细胞感染状态的反事实预测,发现与感染状态相关的基因的 GSEA(基因集富集分析) GSEA 显示标志基因集(H 表示)和 KEGG 基因集(K 表示)(使用排列测试和 Benjamini-Hochberg 校正计算的 Padj)。 e,f, 疟原虫肝阶段单细胞图谱中从中间到后期时间点的感染细胞的 UMAPs 细胞根据报告的 36 小时后感染细胞的消退/生产性分类着色(e)以及 biolord 对所有感染细胞的消退/生产性分类(f)。插图显示了每个时间点消退细胞的比例(24 小时后 0.016;30 小时后 0.057;36 小时后 0.215)。 g, 比较消退和生产性细胞的箱形图显示,消退的肝细胞在所有时间点保留了较小部分的疟原虫转录组 箱形图中的中线表示中位数;箱体边界表示四分位数范围(IQR);胡须表示 1.5×IQR;最小值和最大值在箱形图中未标示;灰色点表示超出最小或最大胡须的点(使用两侧 Mann-Whitney-Wilcoxon 测试和 Benjamini-Hochberg 校正:24 和 30 小时后(n=1823 个细胞,跨两种状态);biolord-classify < 0.0001, 36 小时后(n=1083 个细胞,跨两种状态);原始 < 0.0001;***P ≤ 0.0001)。 *h, 消退细胞表现出干扰素反应的过表达,表现为干扰素调节因子和反应相关基因的增加** 根据小时后感染(hpi)排序,群组的树状图显示了从生产性到消退细胞的轨迹。IQR 表示四分位数范围。
讨论¶
总之,我们展示了 biolord 在广泛的任务中的应用,涵盖了多种单细胞方式和生物系统,展示了这种解耦表示能提供的洞察范围。
虽然我们在这里关注的是与已知属性相关的解耦表示,但一个引人入胜的后续方向是研究未知属性的表示。此外,类似于其他解耦方法,当属性相关时,期望的结果是不明确的。这在预测以前看到的组合时不会对结果产生不利影响(例如,如果提供了细胞类型 X 在组织 Y 中的测量)。然而,对未见组合的预测可能会产生不可预测的结果,这是神经网络的已知限制。尽管如此,通过提供一个分解的潜在空间,biolord 允许独立提取每个生物属性的底层结构,从而减轻上述限制。最后,与任何深度生成框架一样,biolord 遭受缺乏直接解释性的困扰。我们通过建议使用解耦潜在嵌入和生成模型的各种下游分析工具来克服这一点,提供生物学洞察力和特征(例如,基因表达)空间的可解释性。
总结来说,biolord 提供了解耦单细胞数据中编码的细胞身份的一步。它阐明了不同组件对整体观察到的表达的影响,从而提供了新的见解和更好地利用多组学数据。
方法¶
潜在优化作为解耦中的归纳偏见¶
潜在优化是我们方法的关键组成部分。典型的表示解耦方法使用编码器将原始数据样本映射到潜在代码中。这通常被称为摊销推断。虽然使用编码器网络将样本映射到代码很方便,但 Gabbay 和 Hoshen 展示了这种方法可能会达到次优结果。原因是在训练开始时,编码器(随机初始化)将所有样本属性映射到每个潜在代码,无论是已知的还是未知的。虽然损失函数鼓励解耦(移除已知属性),但编码器的随机初始化导致优化从一个完全纠缠的状态开始。后续训练迭代难以完全去除这种纠缠。
相比之下,随机初始化的潜在代码根本不包含关于已知或未知属性的任何信息。在训练过程中,每个样本对应的潜在代码在未知属性上变得更具信息量,同时解耦目标确保它不会在已知属性上获得信息。直观上,防止获得不需要的信息比失去现有信息更容易。总之,潜在优化通过为学习过程提供更好的初始化,帮助实现更解耦的潜在代码。
值得注意的是,参考文献中的结果直接适用于这里呈现的生物学设置。如上所述,通过潜在优化解决的挑战与标记属性有关。因为这些是与样本(图像或单细胞测量)一起提供的标签,在两种设置中都是相同的。因此,潜在优化使我们能够获得与目标属性相关的有意义的潜在代码。
Biolord 模型¶
Biolord 是一个基于深度学习的生成框架,由多个模块组成,这些模块共同优化。Biolord 的输入是一个数据集 \(D = \left[\left(x_c, y_c\right)\right]_{c=1}^n\),其中 \(n\) 是细胞的数量。对于每个细胞 \(c\),\(x_c \in \mathbb{R}^M\) 代表 \(M\) 个测量特征(例如,来自 \(M\) 个基因的基因表达计数或峰值计数),而 \(y_c\) 是一个大小为 \(K\) 的集合,表示已知的细胞属性,例如细胞类型标签、组织起源或年龄。如下所述,在 \(y_c\) 中,我们在构建其对应的潜在空间时区分分类属性和有序属性。相应地,集合 \(y_c\) 中的每个 \(K\) 元素可能具有不同的维度。鉴于输入数据集 \(D\),Biolord 流程包括两个主要组成部分,同时定义和训练(构建细节在以下小节和补充说明 1 中提供):
- 分解潜在空间 - 对每个已知属性,构建一个专用的子网络。每个子网络的架构基于属性的类型(分类或有序),用户可以修改额外的超参数。我们将每个子网络的输出称为 \(z_y\),它是对应于 \(y_c\) 中的一个属性(分类或有序)的潜在空间,而 \(z_u\) 是未知属性的潜在空间(见图 1b)。
- 生成模块 - 生成器 \(G\) 将连接的分解潜在空间作为输入,并输出对测量特征的预测。
需要注意的是,上述的优化,包括分解潜在空间和生成预测,是联合进行的,这样在分解潜在空间中的嵌入会针对生成器的重建误差进行优化。
已知属性的潜在空间¶
针对已知属性集 \(y_c\),我们为每一个 \(K\) 属性构建了一个专用的子网络来表示其对应的潜在空间。在这里,我们区分了分类属性和有序属性:分类属性中,相似的细胞共享类标签;有序属性中,属性特征之间的距离编码相似性。在我们对有序属性的定义中,我们考虑连续变量以及分类序数变量,因为重要的是属性特征包含结构信息。此外,经常测量的分类序数变量(如年龄)通常代表连续变量的样本。据此,我们按如下方式构建不同的子网络:
- 分类属性子网络 - 这些通过嵌入模块定义,使得潜在代码 \(z_y\) 在属于同一标签的所有细胞之间共享。嵌入直接优化,即通过完整模型的目标函数应用潜在优化。
- 有序属性子网络 - 为了利用每个有序属性的结构,我们使用编码器;默认深度为 \(2\),宽度为 \(256\) 的多层感知机(MLPs)。MLPs 将输入特征映射到相应的潜在空间 \(z_y\),该空间通过完整模型的目标函数进行优化。
未知属性的潜在嵌入¶
我们通过直接优化每个样本的嵌入来学习未知属性的表示。我们使用一个添加了高斯噪声 \(\eta\)(一个随机变量 \(\eta \sim \mathscr{N}\left(0, \sigma^2 I\right)\),固定方差值 \(\sigma\))的正则化嵌入子网络模块。输出是每个细胞的独特潜在代码 \(z_l\),独立于基因表达或已知属性,在训练中使用潜在优化进行优化。
因此,为每个细胞优化一个独特的代码可能会阻碍我们的解耦努力;模型可能会将所有表达信息编码到未知属性的潜在代码中,并忽略属性特定的编码。根据 Gabbay 和 Hoshen 的建议,为了确保已知属性信息不会泄漏到未知属性的表示中,我们以两种方式进行正则化。首先,我们引入嵌入中的加性高斯噪声,其次,我们在损失中添加一个激活惩罚项,以限制嵌入的幅度,从而引入最小性损失项。
最小性损失函数¶
最小性损失函数表示为:
其中,\(\lambda\) 是这一项的超参数权重。这些措施共同确保了未知属性和已知属性表示之间共享信息的最小化。也就是说,未知属性的表示被优化以最小化其包含的关于已知属性的信息。
生成器模块¶
生成器 \(G\) 构建为一个解码网络,由参数 \(\theta\) 确定,它以连接的分解潜在空间为输入,并输出测量特征表达分布的参数化(由均值和方差给出),
根据提供给模型的数据类型,如预处理的对数正态化数据、原始计数或峰值,分布 \(P\) 可能遵循高斯分布、零膨胀完整性损失项,我们对每种分布使用相应的负对数似然损失,NLL \(\left(x \mid G_\theta\right)\)。根据 Gabbay 和 Hoshen 的原始模型,我们加入了一个均方误差项,关于预测均值 \(\mu_\theta\),由 \(G_\theta\) 提供,\(\operatorname{MSE}\left(x, \mu_\theta\right)\),由 \(\tau\)('reconstruction_loss')超参数调整。这使我们能够直接优化均值预测,适用于所有选择的参数分布建模(补充说明 1)。因此,我们可以将完整性项写为,
模型优化¶
综合以上内容,我们可以将完整模型目标写成两个项的组合。第一项通过优化生成器的准确性来诱导完整性,第二项强制执行已知和未知属性表示之间共享的信息的最小性。
Biolord 模型总体损失函数¶
Biolord 模型的总体损失函数由两部分组成,反映了模型各个组件的联合优化目标:
这里,模型中定义的不同组件共同优化,分解的潜在空间中的嵌入以及生成器的预测输出受到输入测量和已知属性标签的影响。
Biolord-classify:具有部分标签的生物表征解耦¶
为了执行半监督解耦,即在我们缺少一部分细胞的标签的情况下,我们采用了参考文献 \(\underline{13}\) 中提出的方法。除了上述的 biolord 模型组件(分解的潜在空间和生成模块)之外,我们还为每个分类属性包括一个分类器 \(C \in \mathcal{C}\),以及为每个有序属性包括一个回归器 \(R \in \mathcal{R}\),这些都与先前的组件一起训练。
分类器(回归器)将基因表达作为输入并输出类标签/特征。对于缺少标签的细胞,分类器(回归器)的输出用于完成分解的潜在表示(扩展数据图 1)。为了训练分类器(回归器),我们在现有的损失函数中添加了一个项,以鼓励对可用标签样本的正确预测。对于分类器,我们使用分类交叉熵损失 \(H(y, C(x))\)。对于回归器,我们使用输出和提供特征之间的均方误差损失 \(\mathrm{MSE}(y, R(x))\)。在所有情况下,损失仅针对提供标签的细胞进行评估(由集合 \(X^S, Y^S\) 标记)。分类损失由以下公式给出:
其中 \(Y_C^S\)(\(Y_R^S\))表示与相应分类器 \(C\)(回归器 \(R\))相关的标签(特征)集。\(\mathcal{L}_{\mathrm{cls}}\) 被添加到 biolord 目标中,使得所有组件现在一起联合训练。
Biolord-classify 模型总体损失函数¶
通过将分类模块(分类器和回归器)作为 biolord 训练程序的一部分,我们允许在半监督设置中训练 biolord 模型,因为分类器和回归器用于插补作为分解潜在空间输入的缺失标签。此外,插补的标签可以用于数据的下游分析(见图 \(\underline{\text{c}}\))。总体损失函数为:
这种设计使得 biolord-classify 模型能够有效地结合已知标签和通过模型推断的标签来优化模型性能和泛化能力。插补的标签不仅帮助改进潜在空间的表征,还增强了模型对未标记数据的理解和处理能力。这种方法在处理具有不完整标签数据的生物学应用中尤为重要,如在异质细胞群中识别新的细胞类型或状态。
通过结合使用生成模块和分类回归模块,biolord-classify 能够更全面地利用可用的数据,不仅重建细胞的表达特征,还能预测缺失的细胞属性,从而在细胞类型分类、疾病状态识别等方面提供更准确的见解。
Biolord 助力下游分析¶
Biolord 通过使用解耦的潜在表征、生成管道和 biolord-classify 模块(图 1c),实现了多样化的下游分析任务。在 Biolord 框架内,我们提供了实用功能以支持这些分析。下游任务如下:
-
潜在空间表征 - 潜在空间的嵌入提供了对特定属性内部及不同属性之间结构的洞见。潜在表征是一组向量,将已知属性的状态映射到 \(n_{\text{latent}}\) 维状态。任何下游分析工具现在都可以应用于揭示状态之间的属性和关系,例如,相关性分析、聚类或低维表征。潜在表征可用于探索给定属性不同标签之间的结构,例如,使用相关性矩阵,或通过考虑连接表征来研究不同属性之间的相互作用。
-
不确定性评估 - 不确定性测量提供了评估模型泛化能力的代理。我们使用参考文献 \(\underline{5}\) 中提出的评估指标来量化属性在其潜在表征上的不确定性,当与属性相关的额外协变量已知时,例如,药物属性的途径关联,并在 biolord 包中提供其实现。不确定性由从属性的潜在空间表征的 \(k\)-NN 图中预测协变量(药物途径)的能力不足来定义。形式上,我们定义:
$$ u_i = \sum_{j \in \mathscr{N}i} \frac{1}{\log d(i, j)} \times H\left(C\right)}_i
$$
其中 \(\mathscr{N}_i\) 是值 \(i\) 的邻集,\(d\) 是距离测量,\(H\) 是香农熵,\(C_{\mathscr{N}_i}\) 是基于潜在表征的 \(i\) 的邻居的协变量向量。
-
反事实预测 - biolord 模块可以获取特定的细胞实例并修改其已知属性。通过 biolord 获得的未知属性嵌入捕获了特定于细胞的嵌入。因此,当将细胞的测量特征与感兴趣的已知属性的不同标签一起作为输入传递时,特定于细胞的属性表征将保持不变(未知属性嵌入),并且只有修改的已知属性的嵌入会发生变化。由于嵌入是生成模块提供预测的输入,所有观察到的变化都是由已知属性的修改引起的。在实践中进行反事实预测时,我们采取一组参考细胞,例如感染数据集中的对照细胞,并使用其测量特征以及任何已知属性的组合(例如,考虑感染情况修改状态标签)作为输入到训练好的 biolord 模型。这允许我们首先采样未见的生物状态,并更重要的是获得一组受控样本,我们可以保证所有观察到的测量特征的变化都是修改属性的结果(图 1d)。
-
特征与状态的关联 - 将反事实预测与统计测试配对,可以恢复一组编码给定观察状态的特征(例如,基因)。在这里,我们明确地将修改的属性与潜在的细胞状态解耦;因此,预测中观察到的变化是由修改的属性引起的。
-
分类 - biolord-classify 模块可以扩展部分标记属性的标记。这提供了数据的完整标记,原则上可以进一步检查并用作各种下游分析管道的输入。
代码可用性¶
我们使用 scvi-tools 库实现了 biolord,并使用 cookiecutter-scverse(https://github.com/scverse/cookiecutter-scverse)作为包的模板。该软件包已作为开源软件发布,在 https://github.com/nitzanlab/biolord 上可获得。文档可在 https://biolord.readthedocs.io 查看。重现结果的代码可在 https://github.com/nitzanlab/biolord_reproducibility 获取。