Inferring super resolution tissue architecture by integrating spatial transcriptomics with histology
摘要¶
空间转录组学(ST)在生成组织内细胞的复杂分子图谱方面展示了巨大的潜力。我们在此介绍 iStar,这是一种基于分层图像特征提取的方法,它整合了 ST 数据和高分辨率组织学图像,以超分辨率预测空间基因表达。我们的方法将 ST 中的基因表达分辨率提高到接近单细胞水平,并且在仅有组织学图像可用的情况下也能进行基因表达预测。
正文¶
空间转录组学(ST)技术的快速发展使得在原始组织环境中测量基因表达成为可能 1,2,3,4,从而使研究人员能够表征空间基因表达模式 5,6,7,研究细胞间通讯 8,9,并解析细胞发育的时空顺序 10。尽管已有许多 ST 平台可用,但没有一个提供全面的解决方案。理想的 ST 平台应具有单细胞分辨率、覆盖整个转录组、捕获大面积组织并具备成本效益。虽然使用现有平台生成这样的 ST 数据仍具有挑战性,但可以采用计算方法在计算机中重建这些数据。
常见的 ST 实验方法包括原位测序或杂交技术,如 STARmap11、seqFISH12,13,14 和 MERFISH15,16,以及后续使用下一代测序技术的空间条形码技术,如 10x Visium、SLIDE-seqV2 (参考文献 17) 和 Stereo-seq18。这些平台在空间分辨率和基因覆盖范围上有所不同。原位测序或杂交技术通常具有更高的空间分辨率和灵敏度,但基因多重性相对较低,而测序技术覆盖整个转录组,但空间分辨率较低,限制了其研究详细基因表达模式的能力。
先前的研究表明,基因表达模式与组织学图像特征相关,这表明可以从组织学图像中预测基因表达 19,20,21。然而,这些现有方法没有充分利用高分辨率组织学图像提供的丰富细胞信息。在实际操作中,病理学家会分层次地检查组织学图像。在这个过程中,第一步是通过检查捕捉到全局组织结构的高层次图像特征来识别感兴趣的区域。在确定感兴趣区域后,会检查反映组织局部细胞结构的低层次图像特征。在本简报中,为了模拟这一过程,我们提出使用分层图像特征提取方法,旨在捕捉局部和全局组织结构。我们进一步开发了一种利用从分层提取的图像特征中获得的高分辨率组织信息的超分辨率基因表达预测模型。所得的基因表达使得细胞类型注释达到近单细胞分辨率。我们在 iStar(推断超分辨率组织结构)中实现了这些过程。
图 1a 显示了 iStar 的概述。我们的方法采用了一种分层视觉转换器 22,23,24(HViT),该转换器已在公开可用的苏木精和伊红染色组织学图像数据集上使用自监督学习(SSL)25,26 进行了预训练。HViT 最初在 16×16 像素尺度上提取组织学特征以捕捉细粒度的组织特征,随后在 256×256 像素尺度上捕捉全局组织结构。随后,通过弱监督学习训练的前馈神经网络使用这些特征来预测超像素级别的基因表达。该模型将每个基因在给定点上的基因表达测量分成多个值,每个超像素分配一个值,由每个超像素的组织学特征辅助。此外,只要有组织学图像,该模型还可以预测点外以及外部组织切片中的超像素级别的基因表达。
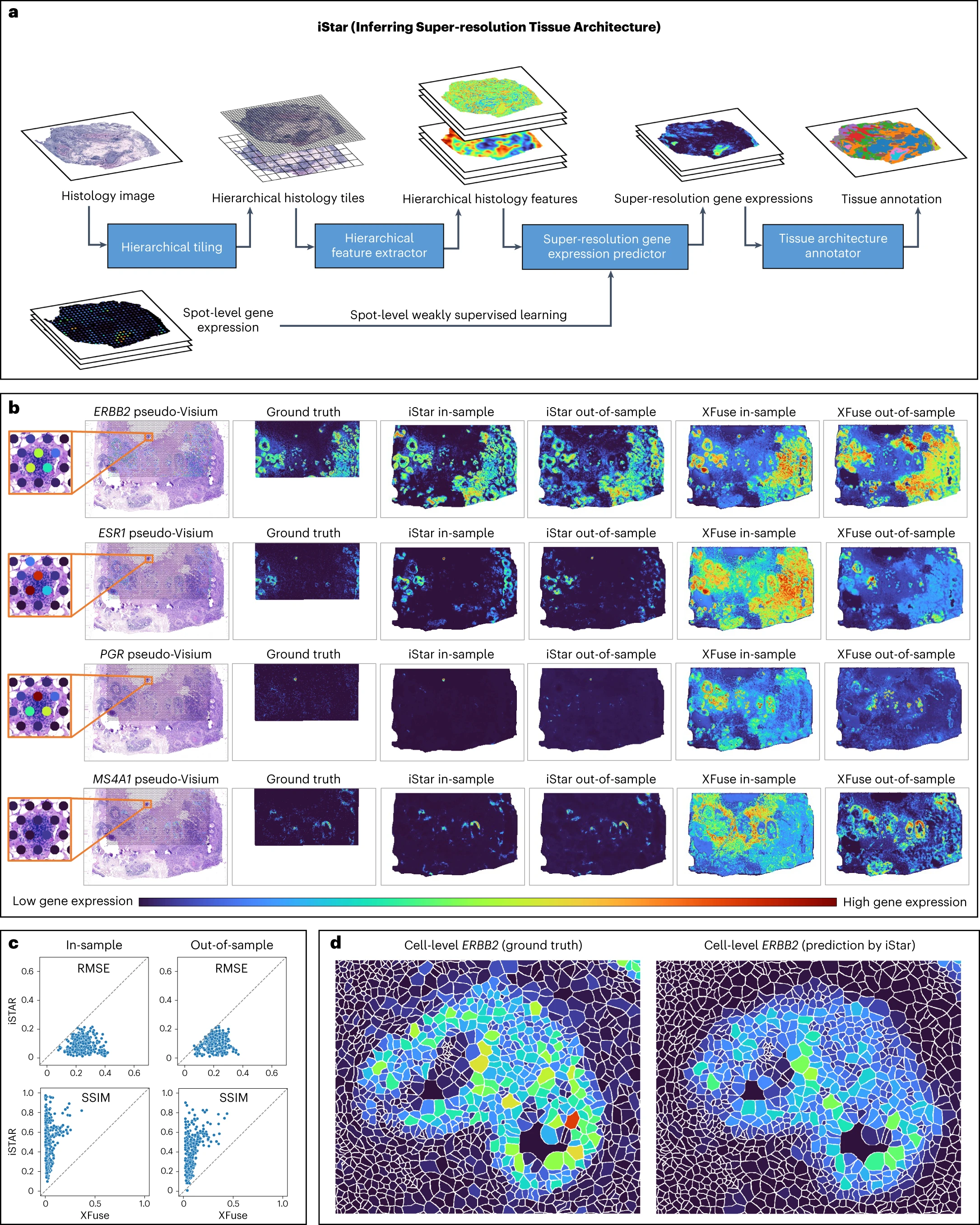
a, iStar 模型总结。组织学图像按层次划分为 patches,然后转换为分层组织学图像特征。这些特征与点级基因表达数据结合使用,以预测超分辨率基因表达。最后,基于超分辨率基因表达预测推断组织结构。
b,c, 使用 Xenium 乳腺癌数据集评估超分辨率基因表达预测的准确性,该数据集包括两个连续切割的组织切片。Xenium 数据作为真实值,模拟基于 Visium 点大小和布局的点级基因表达。对于样本内预测,模型训练和预测均使用第 2 部分的伪 Visium 数据。对于样本外预测,使用第 1 部分进行模型训练,第 2 部分进行预测,在预测时仅使用其组织学图像作为输入。iStar 和 XFuse 的视觉比较。ERBB2、ESR1 和 PGR 是编码乳腺癌预后生物标志物的基因,而 MS4A1 是 B 细胞标记基因。超分辨率基因表达在 8× 分辨率增强比例下进行可视化。更多基因在 8× 分辨率增强比例下的可视化显示在补充图 1-5 中。其他分辨率的可视化显示在补充图 6 和参考文献 40 中。iStar 和 XFuse 在基因表达的 128× 分辨率增强下的数值比较。分辨率增强的程度定义为超分辨率预测中的超像素数与训练数据中的点数的比值。每个点代表 313 个基因之一。其他数值评估报告在补充图 7 中。
d, 预测的单细胞级基因表达,是使用数据集中提供的细胞分割掩膜从预测的超像素级基因表达计算得出的。更多示例显示在补充图 9 中。
为了评估 iStar 在超分辨率基因表达预测方面的准确性,我们将其应用于从由 10x Genomics 生成的 Xenium 乳腺癌数据集衍生的模拟数据集。Xenium 数据集包括 313 个基因的亚细胞 ST 数据,这些数据是在来自单个患者的两个连续切割的组织切片中测量的。为了模拟 Visium 数据,我们根据 Visium 的点大小和布局对 Xenium 基因表达进行分箱。我们评估了样本内和样本外预测的准确性。对于样本内预测,超分辨率基因表达预测是在第 2 部分的伪 Visium 数据上进行的。对于样本外预测,第 1 部分的伪 Visium 数据用作训练数据,并在第 2 部分上进行超分辨率基因表达预测,只使用其组织学图像作为输入。我们将 iStar 的预测准确性与最先进的方法 XFuse 进行比较,视觉上,iStar 的预测与 Xenium 测量的真实值更为接近(图 1b)。为了数值评估性能,我们计算了每个基因的预测和真实基因表达之间的均方根误差(RMSE)和结构相似性指数(SSIM)。iStar 在几乎所有基因和所有分辨率上均优于 XFuse(图 1c,扩展数据图 1-3 和补充图 1-5)。iStar 不仅提高了测量点内的基因表达分辨率,还预测了测量点外的高分辨率基因表达,例如点之间的组织间隙和仅有组织学图像可用的相邻组织切片。我们进一步评估了 iStar 预测单细胞水平基因表达的能力。如图 1d 和扩展数据图 4 和 5 所示,iStar 预测的基因表达与 Xenium 测量的基因表达相似。
接下来,我们评估了 iStar 对多个组织切片的高分辨率组织结构注释的能力。与现有框架通常涉及的复杂图像配准任务不同,我们展示了 iStar 可以绕过图像配准步骤。为了说明这种能力,我们以图 1 中的乳腺癌数据为例,假设伪 Visium 训练数据可用于第 1 部分,但不可用于第 2 部分。为了对两个部分进行超分辨率基因表达预测,我们将它们的组织学图像连接成一个,并在后续分析中将其视为单个图像,以对两部分进行超分辨率基因表达预测。我们使用前馈神经网络的倒数第二层作为 k-means 算法的特征,结果的分割高度符合手动注释,成功将侵袭性癌症(棕色簇)、导管原位癌(DCIS)#1(灰色簇)和 DCIS #2(青色簇)与其余组织分开(图 2a 和补充图 6)。相比之下,使用 XFuse 预测的超分辨率基因表达进行的分割未能将 DCIS #2 与侵袭性癌症或 DCIS #2 与 DCIS #1 分开。此外,iStar 能够对点覆盖组织区域外的组织区域进行注释。最后,第 2 部分的注释与第 1 部分的注释非常相似,展示了 iStar 在多个样本中的一致性。
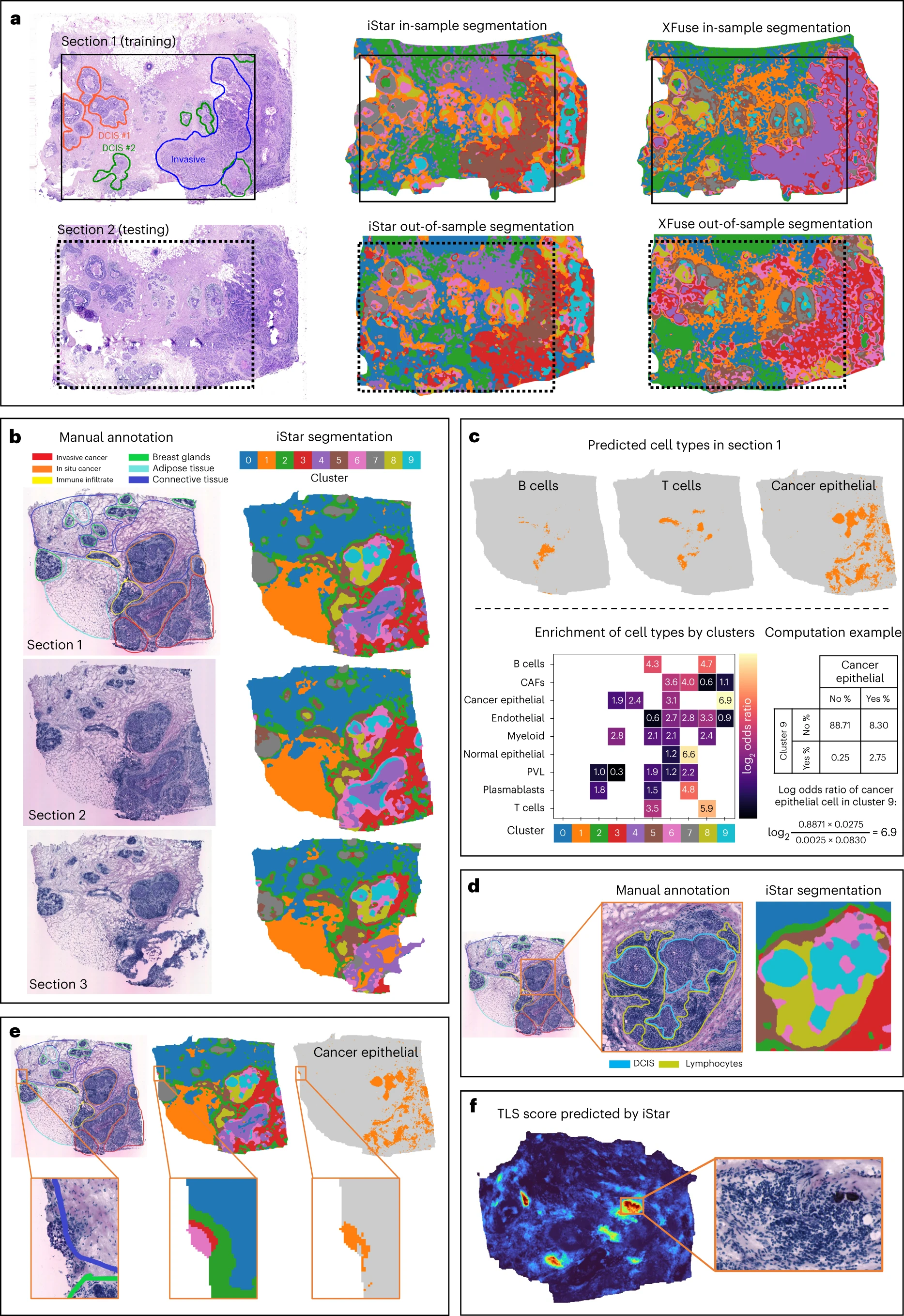
a, iStar 和 XFuse 的无监督组织分割与 Xenium 数据集中乳腺癌患者两个连续切割的组织切片之一的手动注释对比。模型使用从 Xenium 数据的第 1 部分(样本内)模拟的伪 Visium 点级基因表达进行训练。第 2 部分被视为样本外部分,仅使用其组织学图像预测其超分辨率基因表达。超分辨率在 128× 分辨率增强下进行。 b, HER2ST 乳腺癌数据集(受试者 H 的三个连续切割的组织切片)中的乳腺癌组织结构注释。超分辨率在 128× 分辨率增强下进行。 c, iStar 通过执行超像素级细胞类型推断,然后进行细胞类型富集分析,为组织簇分配了生物学上有意义的标签,热图中未显示耗竭,即负富集。CAF,癌症相关成纤维细胞;PVL,血管周围样。 d, iStar 的无监督组织分割揭示了与病理学家的手动注释一致的肿瘤内异质性。 e, iStar 检测到一个手动注释中遗漏的小癌症区域,这是在原始出版物中提供的手动注释中未发现的。 f, iStar 在 HER2ST 乳腺癌数据集(受试者 G)中检测到 TLS。TLS 评分是通过补充表 1 中 TLS 标记基因的标准化超分辨率基因表达的均值计算得出的。
为了评估 iStar 在超分辨率组织分割和注释中的泛化能力,我们将其应用于使用传统 ST 技术生成的另一组 HER2+ 乳腺癌数据集(称为 HER2ST),其空间分辨率低于 Visium(图 2b)。我们考虑了来自受试者 H 的三个连续切割的组织切片,原始出版物中仅对其中一个切片提供了手动注释。为了分割所有三个切片,我们使用与图 2a 相同的方法进行了多样本组织分割,发现 iStar 与粗略的手动注释高度一致,同时提供了更高的粒度(图 2b 和补充图 7-9)。此外,这三个切片显示出相似的结构,证明了 iStar 在多个样本中的一致性。在分割组织后,我们在超像素水平进行了细胞类型注释(图 2c),并根据标记基因的预测基因表达推断细胞类型。细胞类型注释生成了每个组织簇内的细胞类型比例估计,能够评估细胞类型的富集。如图 2c 所示,簇 9(青色)、6(粉色)、4(紫色)和 3(红色)与手动注释的侵袭性和原位癌区域密切匹配,并且富含癌症上皮细胞。此外,簇 8(黄色)和 5(棕色)富含 B 细胞和 T 细胞,这与手动注释结果一致。图 2c 和补充图 10 可视化了被注释为 B 细胞、T 细胞、癌症上皮细胞和其他细胞类型的超像素。每个组织簇的潜在生物学相关性也通过簇中最过表达的基因有所暗示。例如,FABP4(编码脂肪酸结合蛋白)在簇 1(橙色)中富集,CD8A(T 细胞的谱系标记)在簇 5(棕色)中富集,MS4A1(B 细胞的谱系标记)在簇 8(橄榄色)中富集(补充图 11)。
进一步检查 iStar 的无监督分割显示了肿瘤内的异质性,如图 2d 所示,其中精细注释由获得认证的病理学家(E.E.F.)提供。总体而言,超像素级细胞类型注释提供了自动检测到的组织簇的生物学意义解释,与手动标签密切对齐,同时揭示了细微的组织结构。值得注意的是,iStar 甚至能够检测到在原始手动注释中遗漏的阳性手术边缘(图 2e),并且 E.E.F.证实了这一癌症区域的有效性,表明 iStar 能够识别在初始手动注释中被忽略的小区域。我们在此数据集中的发现表明,iStar 即使对于传统 ST 平台也能准确注释组织结构。在组织簇内识别生物学相关基因进一步支持了 iStar 在揭示组织生物学和疾病新见解方面的潜在实用性。
接下来,我们展示了 iStar 可以用于检测多细胞结构,例如三级淋巴组织(TLS),这是一种在非淋巴组织中形成的高度组织化的免疫细胞簇,通常出现在炎症部位,包括各种实体肿瘤。研究表明,TLS 的存在与积极的临床结果和对免疫治疗的响应有关。然而,使用点分辨率 Visium 数据进行 TLS 的手动检测由于 TLS 的小尺寸和细粒度特征,既费力又不精确。为了展示 iStar 自动检测 TLS 的能力,我们分析了 HER2ST 数据集中另一位患者(受试者 G)的三个连续切割的组织切片。为了检测 TLS,我们整理了一份唯一的 TLS 标记基因列表(补充表 1),并通过标准化和平均 TLS 标记基因的预测基因表达计算 TLS 基因特征分数(图 2f)。我们识别了多个 TLS,所有这些都得到了认证病理学家的确认(E.E.F.),TLS 标记基因表达显示在补充图 12 中。相比之下,原始 HER2ST 研究检测到的几个 TLS 分析是基于低分辨率点级基因表达,导致分辨率远低于我们的结果(扩展数据图 6)。
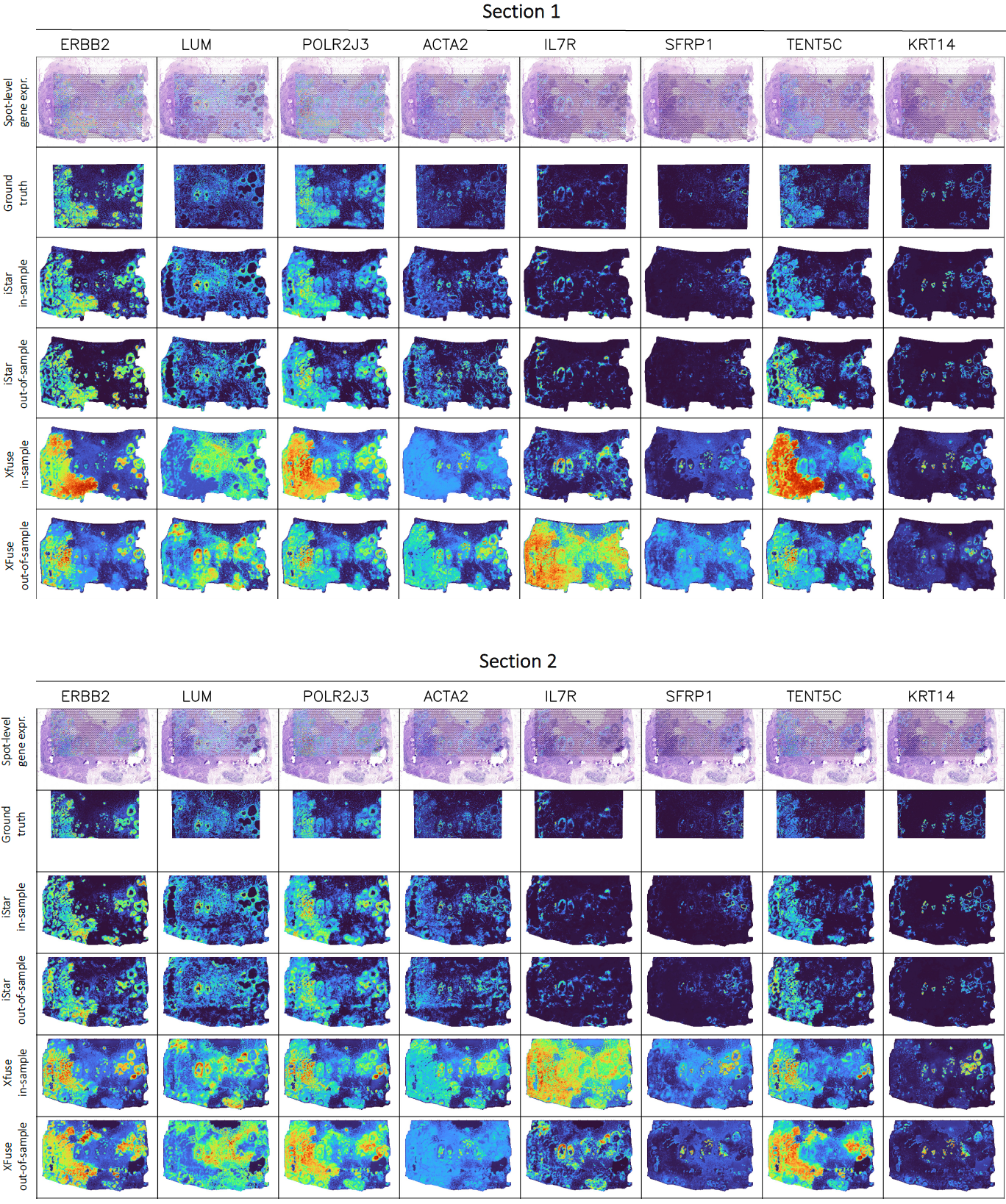
补充图 1:可视化 iStar 和 XFuse 预测的超分辨率基因表达,针对的是在从乳腺癌患者获得的 Xenium 衍生伪 Visium 数据中,313 个基因中方差位于 80%-100% 分位数范围内的 8 个基因。从左到右基因的方差分位数分别是 97.5%、95.0%、…、82.5% 和 80.0%。超分辨率基因表达在 8 倍分辨率增强比例下进行可视化。
除了上述分析的两个乳腺癌数据集外,我们还分析了由 10x Genomics 使用 Visium 生成的另一个乳腺癌数据集。如补充图 13 所示,iStar 揭示了细粒度的组织结构。尽管我们在本研究中主要关注乳腺癌的应用,但 iStar 是一个通用工具,可以应用于各种病变或健康组织类型。为了展示 iStar 在分析健康组织方面的能力,我们使用由 10x Genomics 生成的鼠脑 Xenium 数据集进行了基准评估。基准评估的设计类似于 Xenium 衍生的伪 Visium 乳腺癌数据集的实验。如扩展数据图 7 和 8a 以及补充图 14-17 所示,iStar 在所有分辨率上均实现了高准确性,并且优于 XFuse。此外,我们基于超分辨率基因表达的分割(扩展数据图 8d)与 XFuse 的分割(扩展数据图 8e)相比,揭示了与 Allen Brain Atlas 注释密切匹配的细粒度组织结构(扩展数据图 8b,c)。
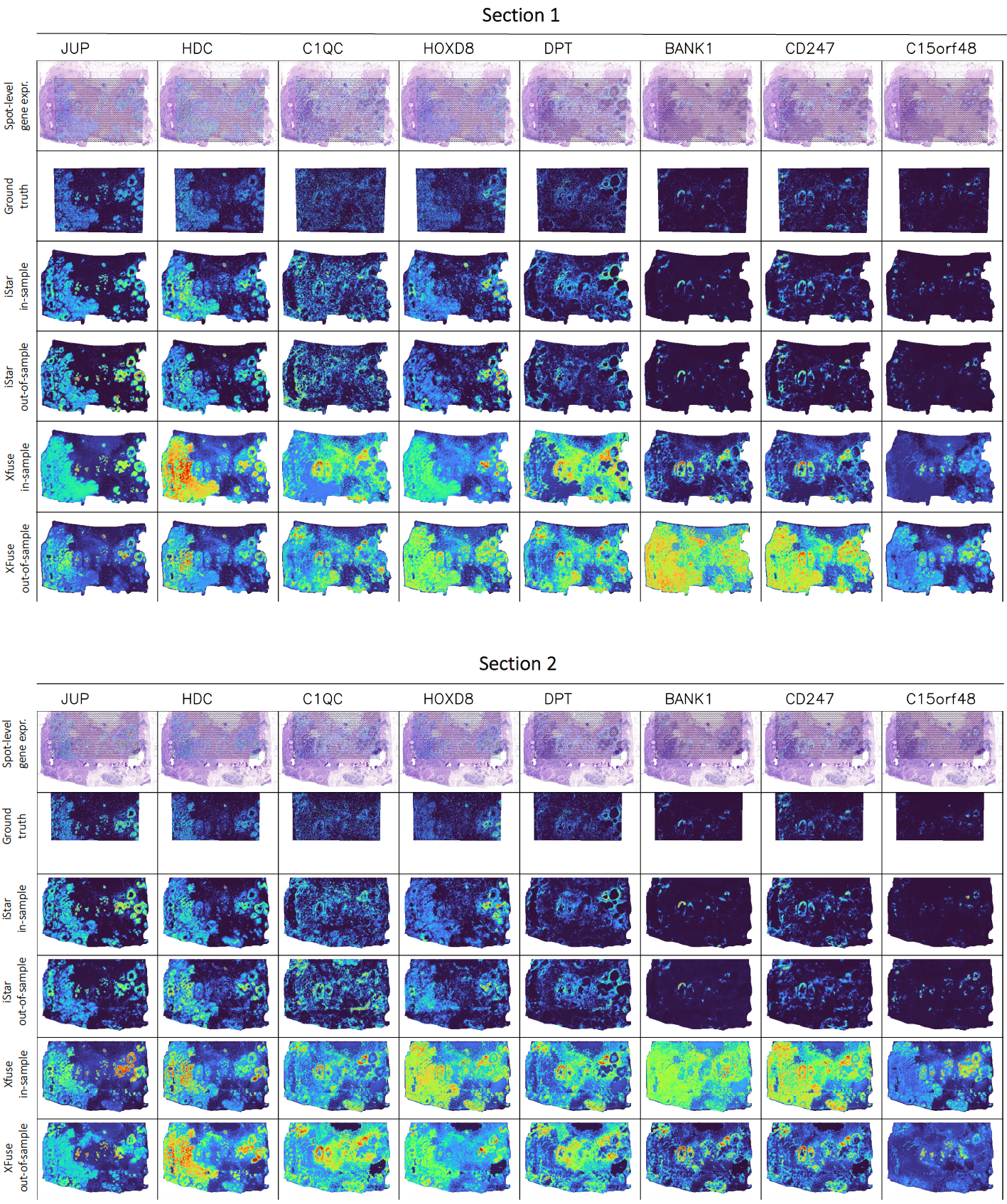
补充图 3:可视化 iStar 和 XFuse 预测的超分辨率基因表达,针对的是在从乳腺癌患者获得的 Xenium 衍生伪 Visium 数据中,313 个基因中方差位于 40%-60% 分位数范围内的 8 个基因。从左到右基因的方差分位数分别是 57.5%、55.0%、…、42.5% 和 40.0%。超分辨率基因表达在 8 倍分辨率增强比例下进行可视化。
最后,为了展示 iStar 对各种癌症和健康组织类型的广泛适用性,我们将其应用于由 Visium 生成的其他数据集,包括鼠脑(扩展数据图 9)、鼠肾(扩展数据图 10a)、前列腺癌(扩展数据图 10b 和补充图 18)、结直肠癌(扩展数据图 10c)和肾癌(扩展数据图 10d)。在所有应用中,iStar 都能够高分辨率地表征组织结构。例如,iStar 准确检测到与病理学家的手动注释一致的肾癌中的 TLS(扩展数据图 10d)。
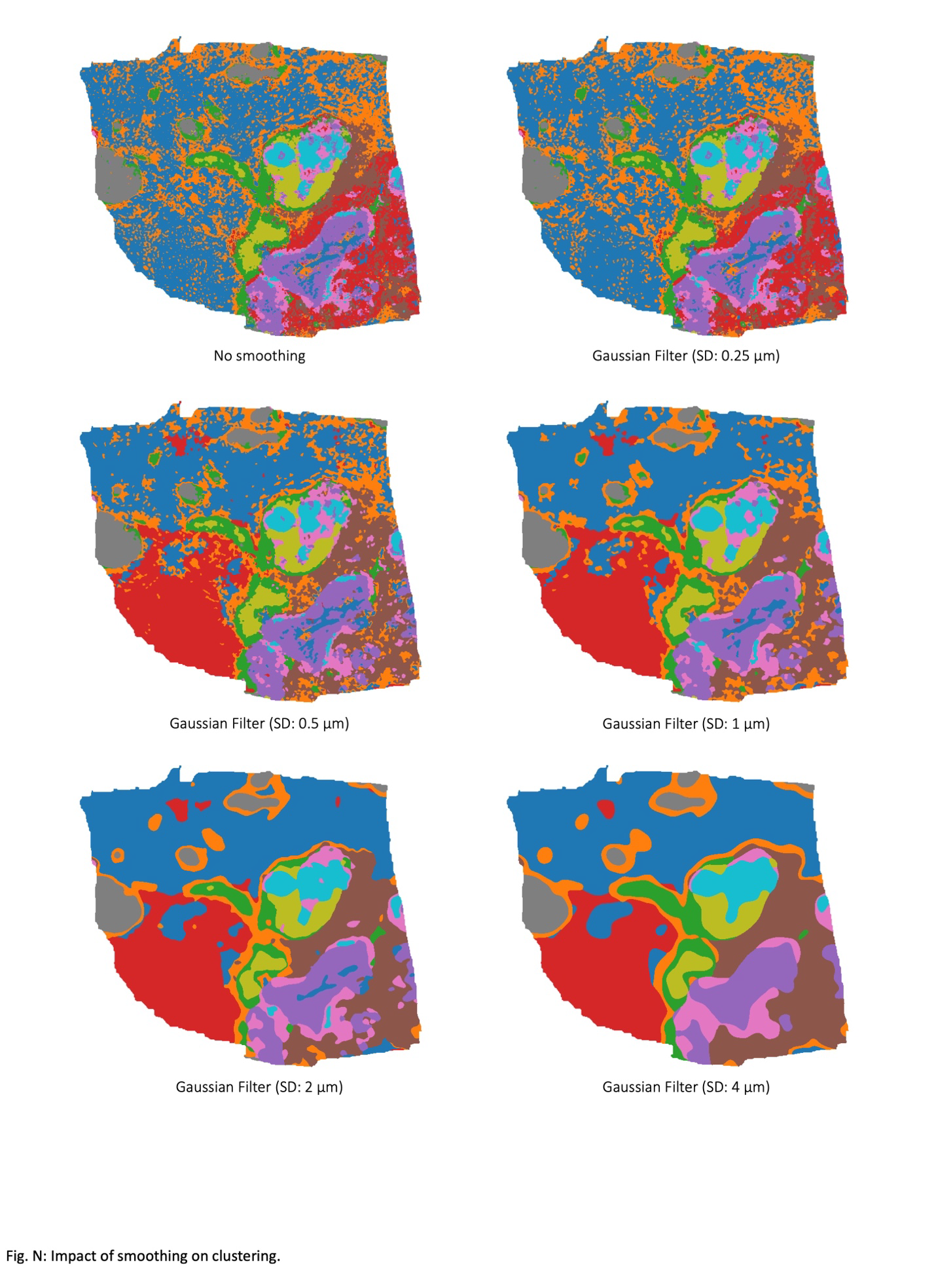
补充图 7:在 HER2ST 乳腺癌数据集的 H1 样本中,iStar 分割在不同高斯滤波器平滑强度下的表现。在此分析中,高斯滤波器平滑了从基因表达预测器的前馈神经网络倒数第二层获得的基因表达嵌入。平滑后的嵌入被用作 K-means 聚类的输入。高斯滤波器中的标准偏差(SD)决定了平滑的程度。
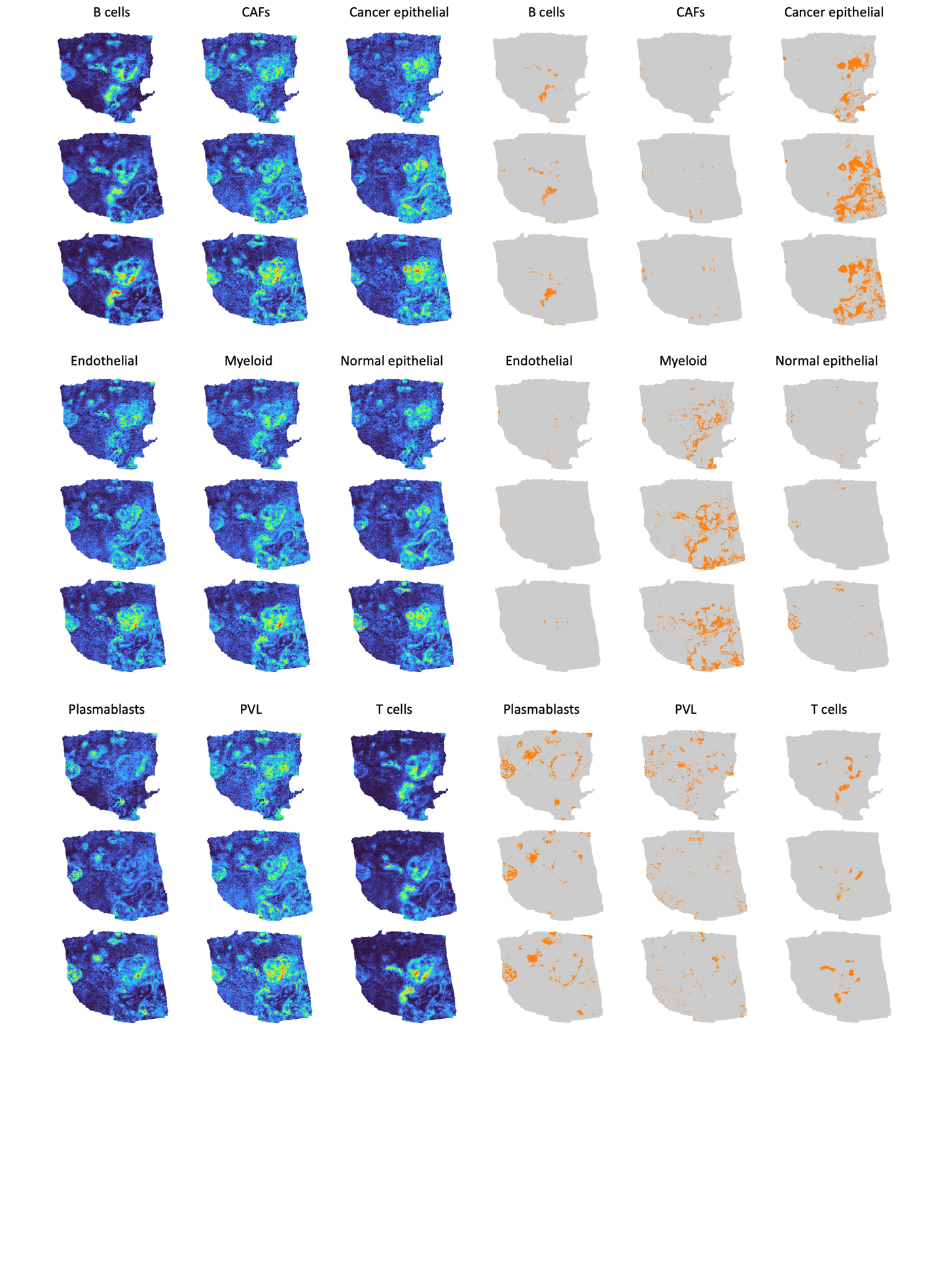
补充图 10:Anderson 等人在 HER2ST 乳腺癌数据集中,受试者 H 的三个连续切割的组织切片中每种细胞类型的基因表达得分和预测的细胞类型图。超分辨率在 128 倍分辨率增强下进行。
总之,我们介绍了 iStar,一种基于缺乏单细胞分辨率的 ST 数据快速注释超分辨率组织结构的方法。这对实际研究具有重要意义,因为现有 ST 平台缺乏单细胞分辨率或全转录组覆盖。然而,iStar 允许我们生成覆盖整个转录组且接近单细胞分辨率的 ST 数据(补充图 19)。iStar 的关键步骤是利用从同一 ST 组织切片中获得的高分辨率组织学图像来重建未观测到的超分辨率基因表达。通过对多个癌症类型和健康组织的数据集的分析,我们证明了 iStar 预测的超分辨率基因表达是准确的。这些预测不仅保留了原始的点级基因表达(补充图 20 和 21),还在各种组织结构推断任务中具有实际应用。此外,我们展示了 iStar 可以对仅有组织学图像的组织切片进行样本外预测。iStar 计算效率高,分析 Xenium 衍生的伪 Visium 乳腺癌数据仅需 9 分钟(补充表 2)。相比之下,XFuse 分析相同数据需要 1,969 分钟,慢了 218 倍。这种计算效率的优势使 iStar 能够从具有组织学图像的大量连续切割的组织切片中生成虚拟 ST 数据,从而全面表征 3D 组织中的基因表达变化。
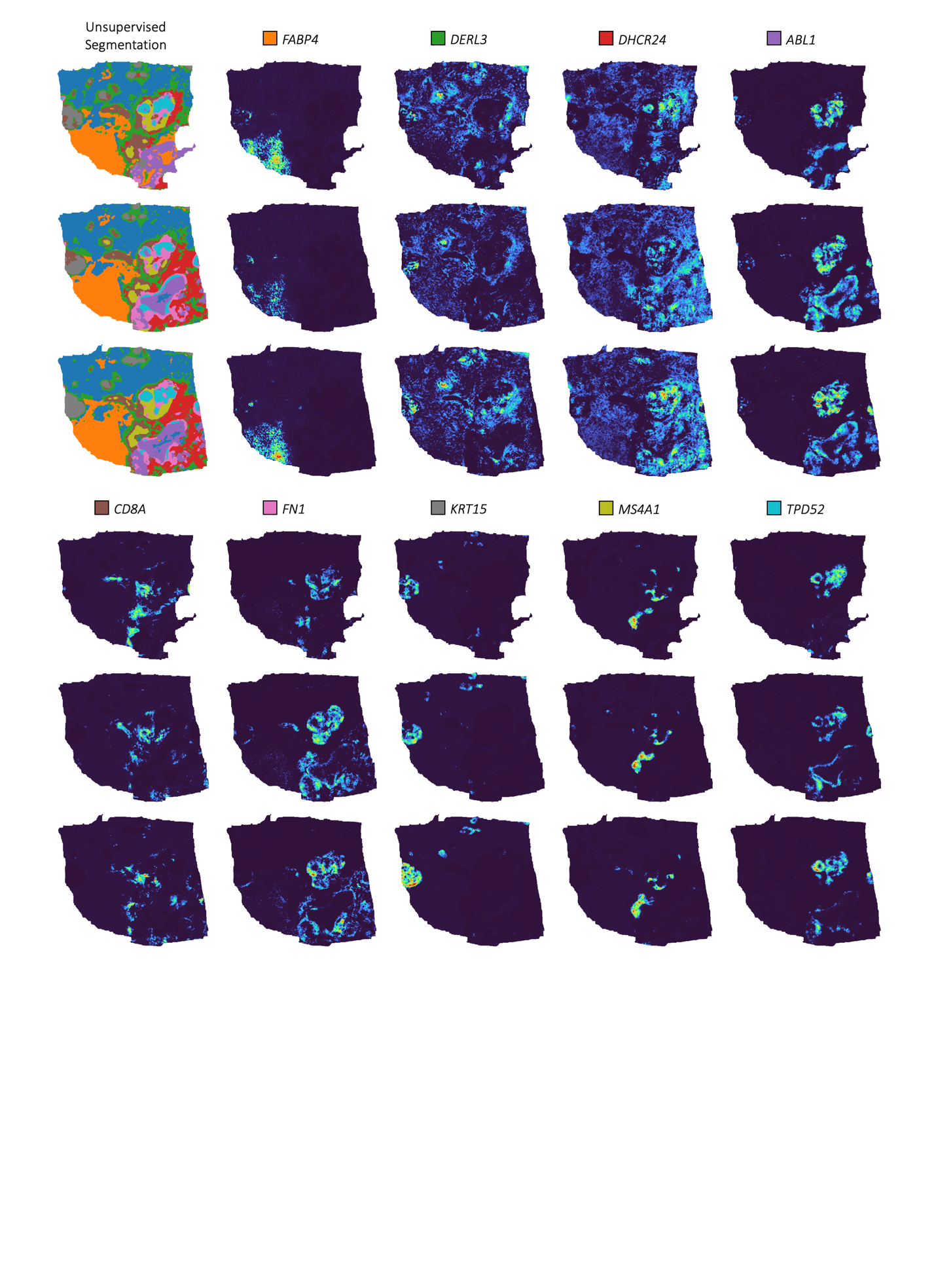
补充图 11:在 Anderson 等人 HER2ST 乳腺癌数据集中,受试者 H 的三个连续切割的组织切片中,每个自动检测到的组织簇中最过表达的基因。仅包含平均倍增变化 2.0 或以上的基因的簇。超分辨率在 128 倍分辨率增强下进行。
方法¶
iStar 算法¶
iStar 算法由三个部分组成:组织学特征提取器、超分辨率基因表达预测器和组织结构注释器。
组织学特征提取器¶
为了方便处理不同分辨率的组织学图像,我们首先将每张图像重新缩放,使一个像素的大小为 \(0.5 \times 0.5 \mu \mathrm{m}^2\)。这种重新缩放确保了 \(16 \times 16\) 像素的 patches 对应于 \(8 \times 8 \mu \mathrm{m}^2\),大约是一个单细胞的大小。为了简化随后的 patches 处理程序,我们填充重新缩放后的图像,使其高度和宽度均能被 256 整除。
接下来,我们按层次将整个图像划分为图像 patches,使得大的(高层次)patches 反映全局组织结构,而大的 patches 内的小(低层次)patches 反映组织的局部细粒度细胞结构。设 \(X \in \mathbb{R}^M \times \mathbb{R}^N \times \mathbb{R}^3\) 为高度为 \(M\),宽度为 \(N\) 的 RGB 通道组织学图像。我们首先将 \(X\) 划分为一个 \((M/256)\) 行,\((N/256)\) 列的 \(256 \times 256\) 像素图像 patches 矩形网格:
其中每个 \(X_{m_1 n_1} \in \mathbb{R}^{256} \times \mathbb{R}^{256} \times \mathbb{R}^3\)。接下来,每个 \(256 \times 256\) 像素图像 patches 进一步划分为一个 16 行,16 列的 \(16 \times 16\) 像素图像 patches 矩形网格:
其中每个 \(X_{m_1 n_1 m_2 n_2} \in \mathbb{R}^{16} \times \mathbb{R}^{16} \times \mathbb{R}^3\)
为了提取分层组织学特征,我们使用了一个 HViT 架构,该架构由一个局部视觉 transformer \((\mathrm{ViT}) f_1 f_2\) 和一个全局 ViT \(f_0\) 组成。首先,在每个 \(256 \times 256\) 像素图像 patches 内,局部 ViT 将每个 \(16 \times 16\) 像素子 patches 映射为长度为 \(C_2\) 的低层次局部特征向量,即,
然后,将 \(256 \times 256\) 像素图像 patches 中的所有 256 个低层次局部特征向量映射为长度为 \(C_1\) 的高层次局部特征向量,即,
接下来,为了对整个图像中组织学特征的长程依赖关系进行建模,全局 ViT 将整个图像中的所有高层次局部特征映射为相同维度的高层次全局特征:
在完成这个分层的组织学特征提取过程后,我们得到:
- 高层次全局特征图像 \(T=\left[\mathbf{t}_{m_1, n_1}\right]_{m_1=1, n_1=1}^{M / 256, N / 256}\),这是一个大小为 \((M / 256) \times(N / 256)\),具有 \(C_1\) 个通道的图像;
- 低层次局部特征图像 \(Z=\left[z_{m_1 n_1 m_2 n_2}\right]_{m_1=1, n_1=1, m_2=1, n_2=1}^{M / 256, N / 256,16,16}\),这是一个大小为 \((M / 16) \times(N / 16)\),具有 \(C_2\) 个通道的图像;
- 原始 RGB 图像,这是一个大小为 \(M \times N\),具有三个通道的图像。
为了对齐这些特征图像,我们使用双三次插值将每个图像调整到所需的大小 \(M^{\prime} \times N^{\prime}\) 并叠加调整后的图像通道,从而得到一个组合的组织学特征图像 \(H=\left[h_{m n}\right]_{m=1, n=1}^{M^{\prime}, N^{\prime}}\),大小为 \(M^{\prime} \times N^{\prime}\),具有 \(C_1+C_2+3\) 个通道,其中每个 \(h_{m n} \in \mathbb{R}^{C_1+C_2+3}\) 是像素 (\(m, n\)) 处的组织学特征向量。在我们的实现中,我们设定 \(C_1=192\) 和 \(C_2=384\)。对于图像大小,我们在 \((M / 16, N / 16),(M / 32, N / 32),(M / 64, N / 64)\) 和 \((M / 128, N / 128)\) 之间进行了变化。
训练组织学特征提取器¶
为了训练组织学特征提取器,我们通过自监督学习(SSL)优化视觉 transformer(ViTs)。由于迁移学习对 ViTs 的好处【41】,该模型在公开可用的组织学数据集上进行预训练。在这一步中,由于只需要组织学图像而不需要基因表达数据或图像级标签,许多公开可用的组织学数据集,如癌症基因组图谱(TCGA)、基因型 - 组织表达项目(GTEx)以及 Holscher 等人的肾活检数据【42】,都适合用于模型预训练。此外,针对 SSL 的选择,我们采用了 Chen 等人的预训练模型【2】,该模型使用 DINO 在 TCGA 数据上分层训练 ViTs。在我们的实验中,我们发现预训练模型能够很好地捕捉组织学特征,因此决定跳过微调步骤以提高计算效率。
超分辨率基因表达预测器¶
一旦提取了组织学特征图像,我们使用它们来预测超分辨率基因表达。每个超像素的组织学特征不仅包含其局部细胞特征的信息,还包含其与整个图像中其他区域的全局关系。因此,基因表达预测器不需要显式地建模空间依赖性,因为超像素之间的相关性已经在其高层次全局组织学特征(即组合组织学特征图像 \(H\) 中的 \(C_1\) 通道)之间的相似性中得到了反映,即使超像素在物理上相距较远。因此,在预测超像素的基因表达时,预测器的输入仅包括该超像素的组织学特征,不需要卷积、注意力或任何其他具有空间意识的机制,从而大大减少了计算成本。
为了训练超分辨率基因表达预测器,由于模型输出是在超像素级别而训练数据是在点级别,我们采用了一种弱监督学习框架。我们将每个点上观察到的基因表达建模为该点内超像素基因表达的总和。这种模型设计模拟了基于测序的 ST 平台的数据收集过程,这些平台将点内的所有转录本条形码化并合并成一个样本,然后送去下一代测序。为了表达损失函数,设 \(S\) 为整个图像中的点数,\(K\) 为要预测的基因数,\(g_k\) 为基因 \(k\) 的基因表达预测模型,\(y_{ks}\) 为点 \(s\) 处基因 \(k\) 的观测基因表达,\(\mathscr{M}_s\) 为点 \(s\) 的点掩膜(即点 \(s\) 覆盖的超像素的集合),\(h_{mn}\) 为超像素 \((m, n)\) 处的组织学特征向量。那么,弱监督损失函数为
点掩膜外的超像素,包括点间间隙和背景图像,在模型训练期间被排除。模型训练后,超像素 \((m, n)\) 处基因 \(k\) 的预测基因表达为 \(\hat{y}_{kmn}=g_k\left(h_{mn}\right)\),这给出了基因表达图像 \(\hat{Y}_k=\left[\hat{y}_{kmn}\right]_{m=1, n=1}^{M', N'}\)。此外,如果提供了细胞分割掩膜,可以使用预测的超像素级基因表达获得单细胞级基因表达,其中前者是后者的加权和,权重等于超像素与细胞掩膜重叠的比例【43】。在我们的实验中,我们只预测了每个数据集中前 1000 个最具可变性的基因和用户定义结构(如 TLS)的标记基因的并集,因为低可变性基因的信噪比低,会在模型训练过程中引入额外噪音。唯一的两个例外是使用 Xenium 乳腺癌数据集(313 个基因)和 Xenium 鼠脑数据集(248 个基因)的基准实验,在这些情况下,我们预测了所有基因,因为它们数量少且需要方法评估。
对于基因表达预测模型的网络架构,我们使用了一个具有 4 个隐藏层的前馈神经网络,每个隐藏层有 256 个节点。泄露整流线性单元(ReLU)【44】被用作隐藏层的激活函数。输出层是一个线性层,具有 256 个输入节点和 \(K\) 个输出节点,输出由指数线性单元(ELU)【45】激活,以确保预测的基因表达为非负。
组织结构注释器¶
获得超分辨率基因表达后,我们通过使用基因表达信息对超像素进行聚类来分割组织。首先,我们通过减少预测基因表达向量的维度来获得基因表达嵌入,其中每个超像素被视为一个样本,每个基因作为一个特征。虽然任何维度降低技术(例如,主成分分析【46】或统一流形近似和投影【47】)都可以从预测的超分辨率基因表达中获得基因表达嵌入,但我们建议将基因表达预测模型倒数第二个前馈层中的中间值作为基因表达嵌入,因为它们不仅是低维的(在我们的设置中为 256),而且与预测的基因表达向量线性相关,使用这些预计算的值不会产生任何额外的计算成本。接下来,为了在分割中促进空间连续性,我们通过高斯滤波器平滑基因表达嵌入,这种方法在精神上类似于用于细胞邻域识别的滑动窗口方法【48】。然后,我们将每个超像素的平滑基因表达嵌入向量作为样本,使用 \(k\)-means 算法【30】对所有超像素进行聚类。此过程基于其基因表达特征以无监督的方式将组织划分为功能上不同的区域。
为了对分割中的组织区域赋予生物学上有意义的解释,我们在超像素级别进行细胞类型推断,将每个超像素视为一个人工细胞,并使用其预测的基因表达和标记基因参考面板推断其细胞类型。回顾一下,模型中的基因总数为 \(K\)。设 \(T\) 为候选细胞类型的总数。对于每种细胞类型 \(t \in\{1, \ldots, T\}\),假设我们有一个标记基因索引列表 \(\mathscr{A}_t\),它是 \(\{1, \ldots, K\}\) 的子集。例如,在我们对乳腺癌数据的实验中,我们使用了 Wu 等人提供的标记基因列表【33】。对于每个标记基因 \(k \in \mathscr{A}_t\),我们将其预测的超分辨率基因表达图像 \(\hat{Y}_k=\left[\hat{y}_{kmn}\right]_{m=1, n=1}^{M', N'} \in \mathbb{R}^{M' \times N'}\) 标准化到 \([0.0,1.0]\) 范围内,并获得 \(\widetilde{Y}_k=\left[\tilde{y}_{kmn}\right]_{m=1, n=1}^{M', N'} \in \mathbb{R}^{M' \times N'}\),其中 \(\tilde{y}_{kmn}=\left(\hat{y}_{kmn}-\min \hat{Y}_k\right) / \left(\max \hat{Y}_k-\min \hat{Y}_k\right)\)。然后,对于每个超像素 \((m, n)\),我们通过平均其所有标记基因的标准化基因表达来计算细胞类型 \(t\) 的得分:
其中 \(\left|\mathscr{A}_t\right|\) 是 \(\mathscr{A}_t\) 中的基因数。为了推断超像素 \((m, n)\) 的细胞类型,设 \(t_{mn}^{\max}=\underset{1 \leq t \leq T}{\arg \max} u_{tmn}\) 为得分最高的细胞类型,\(u_{mn}^{\max}=\max_{1 \leq t \leq T} u_{tmn}\) 为该细胞类型的得分。给定一个预定的阈值 \(u*{\text {threshold}} \in[0,1]\),如果 \(u*{mn}^{\max}>u*{\text {threshold}}\),则超像素 \((m, n)\) 的细胞类型被预测为 \(t*{mn}^{\max}\);否则,该超像素的细胞类型未分类。在我们的实验中,我们设定 \(u*{\text {threshold}}=0.1\),发现它在大多数情况下是有效的。有关 \(u*{\text {threshold}}\) 对细胞类型推断影响的演示,请参见补充图 22。虽然在我们的实验中使用了这种基于得分的方法进行细胞类型推断,但任何细胞类型注释工具都可以实现这一目的。例如,当有一个注释良好的单细胞 RNA 测序参考面板时,可以使用 SingleR【49】或 ItClust【50】等方法进行细胞类型注释。最后,为了将超像素级别的预测细胞类型与通过无监督分割获得的组织簇结合起来,对每个细胞类型 - 组织簇对进行富集分析,通过检查在簇中过度表达的细胞类型来阐明每个簇内部的生物学活动。
除了上述的无监督组织注释过程,iStar 还允许使用用户定义的组织结构进行注释。在这个过程中,会生成一个分数图像,以反映整个组织中用户定义的结构的强度。用户定义结构分数的计算类似于前一段中描述的细胞类型分数的计算。给定一个用户定义的基因索引列表 \(\mathscr{A}\),它是 \(\{1, \ldots, K\}\) 的子集,针对感兴趣的结构(例如,TLS;见补充表 1),对于每个基因 \(k \in \mathscr{A}\),我们首先将其预测的超分辨率基因表达图像 \(\hat{Y}_k \in \mathbb{R}^{M' \times N'}\) 标准化到 \([0.0, 1.0]\) 范围内,并获得标准化图像 \(\widetilde{Y}_k=\left(\hat{Y}_k-\min \hat{Y}_k\right) / \left(\max \hat{Y}_k-\min \hat{Y}_k\right) \in \mathbb{R}^{M' \times N'}\)。然后,我们通过平均所有标记基因的标准化基因表达图像来计算用户定义结构的分数图像:\(U=|\mathscr{A}|^{-1} \sum_{k \in \mathscr{A}} \widetilde{Y}_k\),其中 \(|\mathscr{A}|\) 是 \(\mathscr{A}\) 中的基因数。所得的分数图像 \(U \in \mathbb{R}^{M' \times N'}\) 反映了组织中用户定义结构的活动情况。
基准数据生成¶
为了评估超分辨率基因表达预测的准确性,我们使用像素级的 Xenium 数据【27】生成了点级的伪 Visium 数据。Xenium 基因表达图像的像素大小为 \(0.2 \times 0.2 \mu \mathrm{m}^2\),我们将像素大小重新缩放为 \(0.5 \times 0.5 \mu \mathrm{m}^2\)。Xenium 中的基因表达测量结果基于 Visium 的点大小、形状和布局分箱到点上:一个具有 55 \(\mu \mathrm{m}\) 点直径和 100 \(\mu \mathrm{m}\) 中心到中心距离的圆盘形点的六边形网格。作为真实值,我们将 Xenium 的基因表达分箱到一个矩形网格的超像素中,超像素的大小根据实验设置在 \(8 \times 8 \mu \mathrm{m}^2\)、\(16 \times 16 \mu \mathrm{m}^2\)、\(32 \times 32 \mu \mathrm{m}^2\) 和 \(64 \times 64 \mu \mathrm{m}^2\) 之间变化。
超分辨率基因表达预测准确性的评估标准¶
为了评估预测的超分辨率基因表达的准确性,对于每个基因,我们将真实值和预测的基因表达都视为图像,并将图像强度标准化到 0 到 1 的范围内。然后,通过 RMSE 和 SSIM 来衡量预测准确性。计算 RMSE 时,首先将真实值和预测的基因表达图像展平为向量,然后 RMSE 等于两个向量之间的欧几里得距离。RMSE 是评估任何可向量化结果预测准确性的一个直接且快速的指标,但对于图像数据,RMSE 忽略了图像内部的空间上下文【51】。因此,除了 RMSE 外,我们还计算了 SSIM,以评估真实值和预测的基因表达图像之间空间结构的相似性。SSIM 是一种广泛用于计算机视觉和医学成像中的超分辨率任务的图像相似性度量【52,53,54】。更高的 SSIM 表示两个图像之间的相似度更高。在我们的情况下,SSIM 捕捉了超分辨率基因表达图像中的全局趋势和细粒度空间结构。我们的实验表明,iStar 在 RMSE 和 SSIM 两个指标上均优于 XFuse。
除了 RMSE 和 SSIM,皮尔逊相关系数(PCC)作为超分辨率任务的一种不常见的指标【55,56】,在一些关于 ST 的前期工作中被用作基因表达预测准确性的评估标准【28,57】。然而,这些工作研究了低分辨率下的 ST,空间单元(即超像素或点)的数量不超过 2000,空间单元的大小约为 100μm。相比之下,我们的实验在更高分辨率下评估预测准确性,超像素数量高达 106,大小小至 8μm。由于图像分辨率高且真实值中的噪声幅度大,PCC 对异常噪声超像素特别敏感,尤其是对稀疏表达的基因。在我们的实验中,与 RMSE 和 SSIM 相比,PCC 在分辨率高时难以区分优劣超分辨率预测。随着分辨率的降低,真实值中的噪声水平也降低,从而导致 PCC 测量的 iStar 和 XFuse 准确性之间的对比更加鲜明。此外,更多的空间可变基因与更高的信噪比相关,产生了更高的 PCC 和 iStar 与 XFuse 之间更大的 PCC 差异,这再次表明 PCC 对真实值中噪声水平的敏感性。总体而言,PCC 在区分高分辨率、高噪声基因表达图像的超分辨率预测准确性方面能力有限。另一方面,当分辨率和噪声水平较低时,PCC 产生的结果与 RMSE 和 SSIM 相似(见补充图 23 和 24)。
计算效率¶
计算效率是 iStar 优于 XFuse 的另一个方面。在基准实验中,iStar 的速度大约是 XFuse 的 200 倍。iStar 对典型数据集的端到端分析通常在 10 分钟内完成,而 XFuse 大约需要一天。训练和预测的详细运行时间报告在补充表 2 中。实验是在 NVIDIA GeForce RTX 2080 Ti 显卡上进行的。
数据可用性¶
我们分析了以下公开可用的 ST 数据集:
- 10x Xenium 人类乳腺癌数据(链接)
- 10x Xenium 小鼠脑数据(链接)
- Anderson 等人报告的 HER2 阳性乳腺癌 ST 数据(链接)
- 10x Visium 人类乳腺癌数据(链接)
- 10x Visium 人类结直肠癌数据(链接)
- 10x Visium 人类前列腺癌数据(链接)
- Erickson 等人报告的人类前列腺癌数据(链接)
- Meylan 等人报告的人类透明细胞肾细胞癌原发性肿瘤数据(GSE175540)
- 10x Visium 小鼠肾数据(链接)
- 10x Visium 小鼠脑冠状切面数据(链接)
- 10x Visium 小鼠脑矢状切面后部数据(链接)
- 10x Visium 小鼠脑嗅球数据(链接)
本文中分析的数据集的详细信息描述在补充表 3 中。10x Xenium 乳腺癌和小鼠脑数据中其他空间分辨率的基因表达可视化可在 这里 获取。本文提供了源数据。