Seeing data as t SNE and UMAP do
降维有助于可视化高维数据集。使用这些工具时应慎重考虑并调整参数。有时,这些方法需要进行进一步的思考。
用肉眼直接观察数据可以作为数据分析的起点。但在演讲或论文中向他人展示数据可视化需要更多的努力。为了在庞大的遗传和基因组数据中发现并呈现有意义的模式,研究人员可能会求助于一类用于降维的统计和计算工具。随着遗传学领域及其数据集的迅速增长,带有降维算法的数据可视化工具被广泛采用。然而,有些人认为这些工具会扭曲和误导数据。
主成分分析 (PCA)、t- 分布随机邻居嵌入 (t-SNE) 和统一流形近似与投影 (UMAP) 是生命科学家用于降维的工具之一。描述降维工具的一篇论文的作者指出,这些工具在理解数据集的几何和邻域结构时“既可能是福也是祸”。在某些情况下,他们指出,t-SNE 可能会产生虚假的聚类,误导研究人员。t-SNE 和 UMAP 都能很好地保留局部结构,但在保留全局结构方面存在困难。
对近期一篇《自然》论文中 UMAP 图的严厉批评重新引发了关于这些方法的长期讨论。一些研究人员解释了为何需要修订该图,并谈到了为什么人们对降维方法的热情如此高涨以及如何更好地使用这些方法。
了解你的维度¶
来自哈佛 T.H.陈公共卫生学院和 Dana-Farber 癌症研究所的生物统计学家 Rafael Irizarry 不喜欢他所看到的许多 t-SNE 和 UMAP 图表。他认为,这些图表对论文贡献甚微,并且这些工具的输出难以进行分析处理。他称 t-SNE 和 UMAP 图为“数据的艺术表现”。这些图表缺乏置信度测量和反映其视觉上整齐的数据簇中不确定性程度的指标。
Irizarry 指出,PCA、t-SNE 和 UMAP 将高维数据降至更易处理的维度。对于单细胞 RNA 测序(scRNA-seq)数据,t-SNE 和 UMAP 常用于将数据降至二维,以便在论文和幻灯片中绘图。为了突出簇,t-SNE 和 UMAP 比 PCA 更受欢迎,因为高维数据点在最终两个维度上会变得“非常接近”,从而留下分离组的空间。他表示,首先使用 PCA 是因为它可以加速 t-SNE 和 UMAP 处理高达 20,000 维数据的速度。因此,许多 scRNA-seq 分析流程首先使用 PCA 压缩维度至 30 到 100,然后再运行 t-SNE 或 UMAP。Irizarry 承认 t-SNE 和 UMAP 是“非常强大、实用的工具”,科学家们应该继续使用它们进行数据聚类。输出结果可以提示研究人员的高维数据中的离散簇。但科学家们不应仅依赖这些工具,他认为必须停止在基因组学中广泛且错误地使用这些工具。
加州大学洛杉矶分校(UCLA)的研究员 Jingyi Jessica Li 表示,对于错误使用这些工具及其难以处理的可视化结果,这是一种有效的担忧。她认为,用户不应选择参数设置来“找到我想要的结果”,以避免确认偏差。参数需要以合理和适当的方式选择。这需要牢记这些工具的功能,并考虑可能处理的数据集的维度。
Li 指出,在群体遗传学中,研究人员可能会测量 100 个个体的 100 万个 SNPs,这是一个高维数据集。相反,如果研究团队收集大组个体的数据但仅收集种族、性别和年龄数据,这就是三维数据,属于低维数据。图宾根大学的数据科学家 Dmitry Kobak 表示,高维数据可能只涉及一种数据模式,例如在许多单细胞中测量 20,000 个人类基因的表达。查看包含许多细胞中 20,000 个人类基因表达数据的表格是非常困难的。
Irizarry 解释说,可以使用线性方法 PCA 来减少数据的维度。例如,如果两个基因是相关的,并且它们的基因表达测量值相同或接近相同,PCA 将平均这两个数值并向前传递。“这是一种压缩和信噪比改进的结合。”
Irizarry 指出,PCA“不懂任何生物学”。它找到线性组合,最大化第一个主成分(PC)中的数据方差。第二个 PC 最大化未被第一个 PC 解释的方差。第三、第四和第五个 PC 也如此,算法找到最大化未被其他 PC 解释的变异的线性组合。基于 PCA 的降维将提高计算机处理数据分析的速度,并且如果操作正确,可以去除数据中的一些噪声。t-SNE 和 UMAP 是用于降维的非线性方法。这些方法的输出本身并不是结果的确认,并且在降维过程中会丢失一些数据。
加州理工学院的研究人员 Lior Pachter 和 Tara Chari 指出,鉴于基因组数据集的高维性,降维有助于过滤噪声,使计算分析可行,并实现探索性数据分析。目标是保留和提取数据中的局部或全局结构并进行生物学推断。由于这些方法可能引起扭曲,他们建议“最好限制降维”,并偏向于引导“聚焦可视化”的针对性分析。
Kobak 和他的同事 Philipp Berens 表示,一些人建议避免使用 UMAP 或 t-SNE 图表。他们不同意这种观点。Kobak 承认,这些图表可能会误导。他说,t-SNE 或 UMAP 的输出“不是分析的终点,而是起点”。
约翰霍普金斯大学布隆伯格公共卫生学院的遗传流行病学家 Genevieve Wojcik 表示,人类有将数据、趋势和经验分组的特性。使用这些降维工具时,应考虑这些分组是否与正在研究的科学问题相关。“每一个分析决策仍然是由你的假设引导的,”她说,“没有一个真理适用于每个数据集。”
学习艰难的教训¶
美国国家卫生研究院(NIH)All of Us 研究计划基因组研究人员在 2024 年 2 月发表的一篇《自然》论文中的一幅图正在修订中。截止发稿时,修订尚未完成。该论文展示了近 25 万名研究参与者的分析数据,包括临床级全基因组序列数据。它统计了超过 2.75 亿个此前未报告的遗传变异,并探讨了基因型与近 120 种疾病之间的潜在关联。正在修订的图是 All of Us 全基因组测序 PCA 数据的 UMAP 表示,显示了研究中每个个体的遗传血统比例,涉及六个血统群体。
发表后,许多研究人员在社交媒体上批评了这幅图。今年 2 月,All of Us 首席执行官 Josh Denny 发表声明指出,自我认定的种族和族裔的社会构建常常与遗传相似性混淆不清。声明中提到,“研究中试图在图 2 中同时表示遗传相似性和自我认定的种族和族裔引发了这一担忧。”在 4 月初的 NIH All of Us 研究者大会上,Denny 表示,这幅图正在修订,因为它“没有遵循我们的最佳指南。”他认为这幅图的批评是合理的,计划承认“这是一个错误。”
Denny 在此背景下提到,NIH All of Us 联合资助并联合撰写了一份先前发表的报告,题为《在遗传学和基因组学研究中使用人口描述符:一个不断发展的领域的新框架》,由国家科学院、工程院和医学院(NASEM)出版。
参与编写 NASEM 报告的 Wojcik 对这幅图的回应和 All of Us 研究人员的反应感到欣慰,她发现这些研究人员特别关注与多样性、遗传学、多组学和健康信息相关的最佳实践。自我认定的种族和族裔在群体遗传学中作为有效构建是有其作用的。但这些描述符无法捕捉遗传方面的内容,它们是社会构建。
在她看来,关于这幅图的反应和批评表明了人类遗传学领域的变化。人们对标准实践可能造成的危害的意识增强了。但这一事件也显示了改变研究方法需要多长时间。她认为 UMAP 本身没有什么本质上的坏处,但科学家需要对他们希望展示的点做出决定。在这个实例中,他们试图展示研究群体的多样性。然而,这种数据可视化的选择意味着“为了展示多样性,你需要展示分离性”,这很有问题,尤其是用算法展示六个来源群体时。它绘制了参与者基因组中最像这六个群体之一的比例。
“西班牙裔”这一类别意味着人们的起源地是讲西班牙语的地方,但“从遗传学角度看,它并没有多大意义”,她说。同样,将所有亚洲人群分成几个群体也没有意义。她指出,人们并不会落入整齐的簇中。使用聚类算法时,研究人员应检查这些簇是否推动了他们的科学问题,并在此过程中反思他们方法决策中的假设。更多关于这些决策的反思将改善群体遗传学和人类遗传学领域。
Aravinda Chakravarti 表示,他听说了这幅图的修订,但没有具体细节。Chakravarti 是纽约大学 Grossman 医学院人类遗传学和基因组学中心的主任,并与杜克大学的 Charmaine Royal 共同主持了 NASEM 报告。除了纠正生物医学中长期以来忽视非多数群体的做法外,Chakravarti 期待着讨论如何在研究中最好地代表多样性。调查人员“必须内化委员会的建议”,并将其用于做出关于方法和人口描述符的选择。
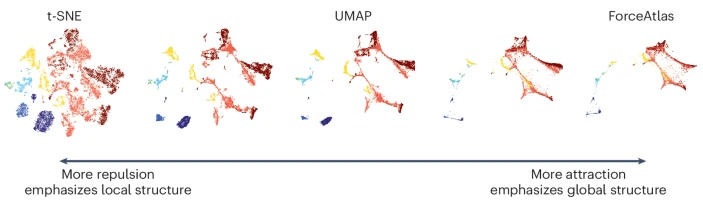
图宾根大学的研究人员为降维算法开发了数学视角,并在此对脑类器官数据进行了展示。像 t-SNE、UMAP 和 ForceAtlas2 这样的算法可以沿着一个光谱进行调节。参数设置会改变数据可视化中邻近点之间的相互作用力,从而改变算法对数据的聚类方式。出处:改编自参考文献 9, CCBY 4.0; S. Damrich, J.-N. Böhm, D. Kobak, P. Berens, 图宾根大学。
不要忘记调参¶
Berens 表示,对于降维方法,建议考虑这些方法的数学原理,牢记它们保留的数据特性,并了解“它们的优点和缺点”。这种见解有助于用户在使用这些方法时做出明智的选择。当科学家决定聚类将强调数据的哪些方面时,他们应记住数据、先验信念和所做的分析选择之间的相互作用。
Berens 指出,PCA 是一种线性变换,具有良好的数学特性。但观察许多绘制的 PC 图表是具有挑战性的。作为人类,我们更喜欢二维的探索性数据分析,这种探索可以揭示之前未发现的惊人方面,Kobak 说,但这些发现需要进一步确认。降维方法的数据显示不是生物学发现。不能把它们当作最终的结论和研究的结束。
Kobak 和 Berens 在一项已发表的研究中评估了一个大规模 scRNA-seq 数据集,他们发现 t-SNE 图生成得不够优化。但他们说,当时对如何将 t-SNE 应用于如此大数据集的了解较少。此后,研究人员在如何设置参数以更好地使用降维方法可视化大规模 scRNA-seq 数据集方面学到了更多。
Berens 和 Kobak 指出,值得关注的参数包括困惑度(perplexity)和学习率(learning rate),它们影响算法的运行方式和结果簇的形状。t-SNE 和 UMAP 都将邻居位置放得很近,但方式略有不同。UMAP 倾向于产生比 t-SNE 更紧凑的簇。另一个名为 ForceAtlas2 的算法在单细胞转录组学中也很受欢迎。即使是相同的数据,这些方法也会导致不同的簇。
t-SNE 和 UMAP 等降维方法在 k 近邻图中表示数据。博士后研究员 Sebastian Damrich 和博士生 Jan-Niklas Böhm 与 Berens 和 Kobak 一起发现,每个点都位于一种力场中。在一篇论文和其他工作中,研究团队展示了这些方法在吸引 - 排斥光谱上的连续性。改变这些力之间的平衡会改变聚类。有些调节保留局部结构更多;其他则更好地保留全局结构。Kobak 说,从某种意义上讲,没有一个固定的 t-SNE 或 UMAP。你可以在它们之间移动,几乎像是一个带有调节旋钮的算法。这一特点的好处在于,研究人员可以通过调节夸张参数,用 t-SNE 看到细微结构。UMAP 和 ForceAtlas2 可能更适合探索数据集的全局结构。研究人员还可以选择查看多个降维工具的多个图表,比较数据表示和聚类过程,有些会更多地压缩数据,有些则较少。“我们提倡查看整个光谱,”Berens 说。这种评估可以作为探索性数据分析的一部分。Kobak 说,他们可以‘转动旋钮’,观察聚类如何变化。这有点像在显微镜中交换物镜,但不同于显微镜,这些不是实际数据,而是数据的非线性变换。
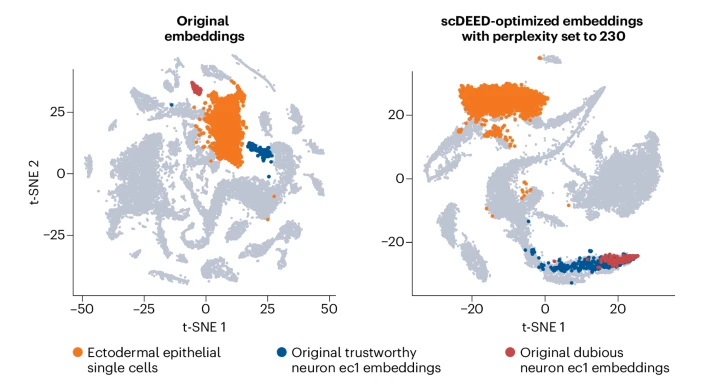
scDEED 有助于评估使用降维算法制作的数据可视化的可信度。下图展示了一个基于 scRNA-seq 的成年水螅体细胞图谱,包含超过 24,000 个细胞的聚类数据集。出处:改编自参考文献 10, CCBY 4.0; L. Xia, 香港科技大学; C. Lee 和 J. J. Li, 加州大学洛杉矶分校。
UCLA 的研究员 Li 强调了在使用降维方法时参数设置的重要性。对于 t-SNE 或 UMAP,一些研究人员可能只使用默认设置,但 Li 认为“默认设置可能并不适合你的数据”。对于 t-SNE,一个需要注意的参数是困惑度(perplexity),而对于 UMAP,则有两个所谓的超参数。根据参数设置,数据可以表示为更紧密或更松散的聚类。“你会信任哪一个?”她问道。当研究人员使用这些工具根据科学假设得出数据结论时,她说,“你需要小心”。她与她的博士生 Christy Lee 及香港科技大学的 Lucy Xia 开发了一种名为单细胞可疑嵌入检测器(scDEED)的统计方法,旨在帮助研究人员更轻松地解决这个信任问题。团队发现,经过 scDEED 优化后,t-SNE 和 UMAP 的表示看起来比之前更相似。
scDEED 对数据可视化进行质量评估,以避免团队所谓的 t-SNE 和 UMAP 等降维技术产生的“可疑嵌入”。该软件为每个数据点计算“可信度评分”,从而评估数据可视化中表示的邻近点的可信度。该工具评估在 PCA 中生成的“预嵌入空间”,这是 t-SNE 或 UMAP 降维之前的步骤。
Christy Lee 表示,运行 scDEED 可能需要一些时间,“因为我们确实需要检查不同的参数”。其运行时间取决于计算设置和数据集大小。值得花时间,因为对数据可视化有更多信任将推动科学结论,例如比较肿瘤细胞和健康细胞的基因表达。
统计学的参与¶
Irizarry 说,“我有一句话:与一个优秀的统计学家合作的回报是,你会发表更少的论文,速度也更慢。”虽然对一些人来说可能显得更慢,但负责的统计分析实际上并不总是更慢。他说,好的统计分析对好的科学至关重要。统计方法有着悠久的历史,早在遗传学研究的早期就有了,而统计学家长期以来一直参与遗传学和基因组学的合作。然而,他记得几十年前听到一些著名研究人员说过类似的话:“如果我需要统计学,那我就做错了实验。”部分由于基因组学的进步,这种情况不再发生。
Wojcik 表示,关于 UMAP 和 t-SNE 的争论有着相当的历史和负担。这些工具需要谨慎使用,但科学中的激励结构“并没有为深思熟虑、缓慢和有条理的科学设置好”。更广泛地获取统计方法和工具是一种积极的变化,追求给定科学问题时增加反思也是如此,即考虑一种方法是否是一个好的选择。“妖魔化一种方法”,如 UMAP 或 t-SNE,“并没有生产力”,她说。“这只是数学。”重要的是“人类如何使用它”。