Towards a general purpose foundation model for computational pathology
摘要¶
在计算病理学(CPath)任务中,组织图像的定量评估至关重要,这需要从整片切片图像(WSIs)中客观地表征组织病理实体。WSIs 的高分辨率和形态特征的多样性带来了显著挑战,这使得为高性能应用进行大规模数据注释变得复杂。为了应对这一挑战,当前的研究努力提出通过从自然图像数据集进行迁移学习或在公开可用的组织病理学数据集上进行自监督学习来使用预训练的图像编码器,但这些方法尚未在不同组织类型上进行大规模开发和评估。我们引入了 UNI,一种通用的自监督病理学模型,通过使用来自超过 100,000 个诊断性 H&E 染色 WSIs 的超过 1 亿幅图像(数据量超过 77TB)进行预训练,涵盖了 20 种主要组织类型。该模型在 34 项具有不同诊断难度的代表性 CPath 任务上进行了评估。除了优于先前的最先进模型外,我们还展示了 CPath 中的新型建模能力,如与分辨率无关的组织分类、使用少量样本类原型进行切片分类,以及在 OncoTree 分类系统中对多达 108 种癌症类型进行疾病亚型化归类的泛化能力。UNI 在 CPath 中推动了大规模无监督表示学习,无论是在预训练数据还是在下游评估方面均如此,使得人工智能模型能够有效地利用数据,在解决解剖病理学中一系列诊断挑战性任务和临床工作流程中实现泛化和迁移。
正文¶
病理学的临床实践包括执行大范围的任务:从肿瘤检测和亚型化到分级和分期,鉴于数千种可能的诊断,病理学家必须擅长同时解决极其多样化的问题组合。现代计算病理学(CPath)通过预测分子变化、预后评估和治疗反应预测等应用,进一步扩展了这一任务数组。由于在收集病理学家的注释、构建单一疾病的大型组织学收集以及获取罕见疾病数据方面存在挑战,从头开始训练模型具有实际限制。这些因素促使 CPath 领域依赖迁移学习技术,这在转移学习在转移学习、概括性和规模化能力上已被证明在诸如转移性检测、突变预测、前列腺癌分级和结果预测等任务中非常有效。
在一般计算机视觉领域,许多基本的自监督模型的开发和评估都基于 ImageNet 大规模视觉识别挑战和其他大型数据集。这些模型也被称为“基础模型”,因为它们在预训练大量数据后能够适应广泛的下游任务。在 CPath 领域,癌症基因组图谱(TCGA)同样作为许多自监督模型的基础,以及其他组织学数据集,许多先前的工作在学习组织学组织的有意义表征方面取得了巨大进展,用于临床病理学任务。然而,目前的预训练 CPath 模型仍受到预训练数据规模和多样性有限的约束,鉴于 TCGA 主要包括原发性癌症组织学幻灯片,并且对多样组织类型的泛化性能评估有限,许多泛癌症分析和 CPath 中流行的临床任务也基于 TCGA 的注释组织学区域(ROIs)和幻灯片。解决这些限制对于 CPath 中基础模型的更广泛发展至关重要,这些模型可以泛化并转移到具有广泛应用的真实世界临床设置中。
在这项工作中,我们在先前的努力基础上,介绍了一种通用的自监督视觉编码器用于病理学,名为 UNI,这是一个大型视觉变换器(ViT-Large 或 ViT-L),预训练于为自监督学习创建的最大的组织学幻灯片收集之一,名为‘Mass-100K’。Mass-100K 是一个预训练数据集,包含来自超过 100,426 个诊断性 H&E 染色 WSIs 的超过 1 亿个组织片段,涵盖了来自麻省总医院(MGH)和布莱根妇女医院(BWH)以及基因型 - 组织表达(GTEx)联盟的 20 种主要组织类型,为学习组织病理生物标志物的客观表征提供了丰富的信息来源。在预训练阶段,我们使用一种称为 DINOv2 的自监督学习方法,该方法已被证明能够为下游任务提供强大的即用型表征,无需进一步使用标记数据进行微调。我们展示了 UNI 在 CPath 中多样化的机器学习设置上的多功能性,包括 ROI 级分类、分割和图像检索,以及幻灯片级弱监督学习。总体而言,我们在解剖病理学的 34 个临床任务中评估了 UNI,这些任务涵盖了一系列的诊断难度,如核分割、原发性和转移性癌症检测、癌症分级和亚型化、生物标志筛查和分子亚型化、器官移植评估以及包括在 OncoTree 癌症分类系统中对 108 种癌症类型进行亚型化的几个泛癌症分类任务。除了优于先前的最先进模型如 CTransPath 和 REMEDIS 外,我们还展示了诸如与分辨率无关的组织分类和基于提示的幻灯片分类的少量类原型等能力,突显了 UNI 作为进一步开发解剖病理学中人工智能(AI)模型的基础模型的潜力。
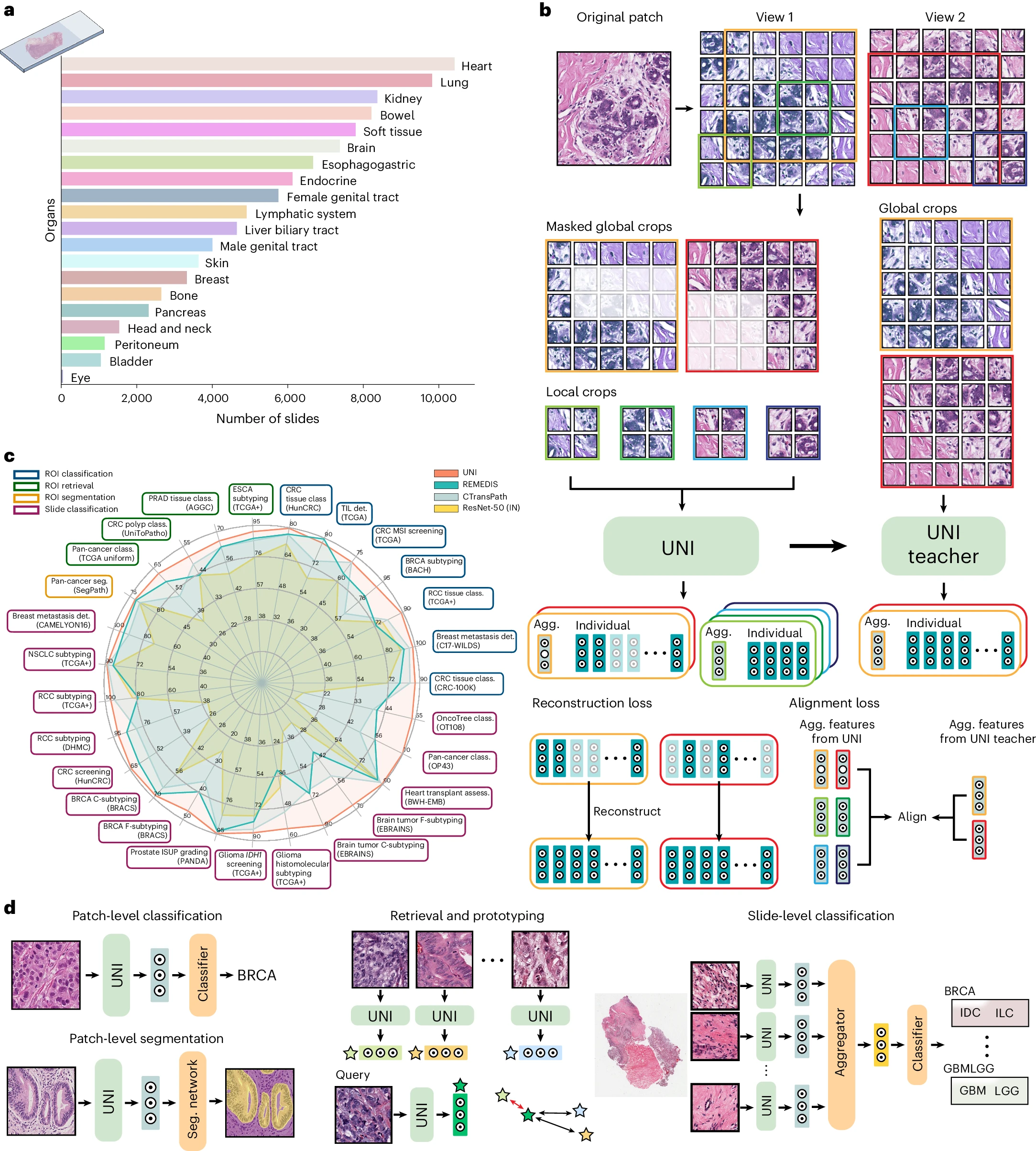
图一:UNI 是一种基于视觉变换器架构的通用自监督视觉编码器,用于解剖病理学,在解剖病理学的 34 个临床任务中实现了最先进的性能。a, Mass-100K 的幻灯片分布,这是一个大规模且多样化的预训练数据集,从超过 100,000 个诊断性 WSIs 中采样了 1 亿个组织片段,涵盖了 20 种主要器官类型。b, UNI 在 Mass-100K 上使用 DINOv2 自监督训练算法进行预训练,该算法包括一个掩码图像建模目标和一个自我蒸馏目标。c, 在解剖病理学的 34 个临床任务中,UNI 通常优于其他预训练的编码器(报告的 8 个 SegPath 任务的平均性能)。d, 评估任务包括 ROI 级分类、分割、检索和原型制作,以及幻灯片级分类任务。更多细节在方法部分给出。分类(class.);分割(seg.);检测(det.);评估(assess.)。
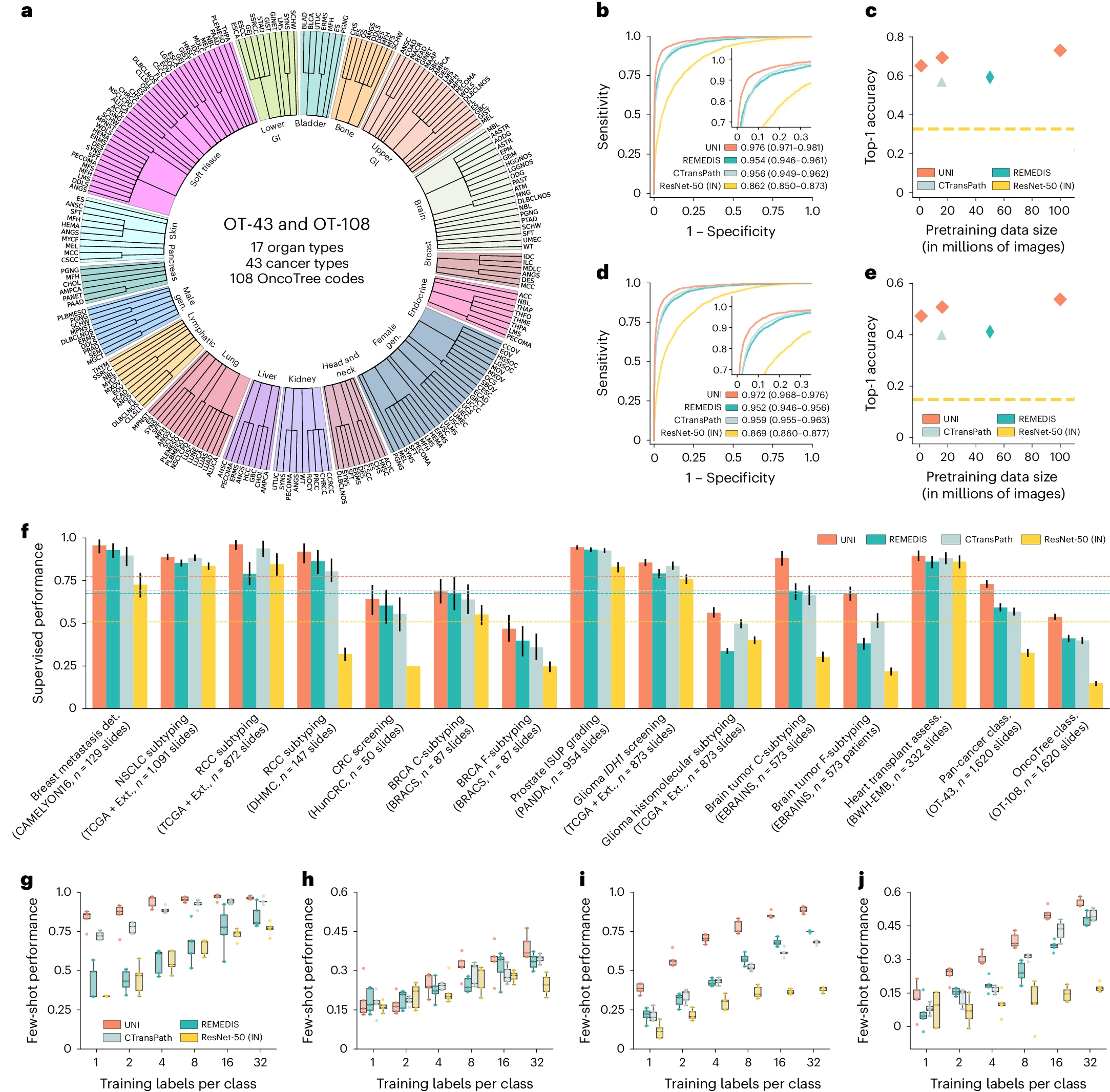
图二:a, 幻灯片级 OT-43 和 OT-108 分类任务中器官和 OncoTree 编码的分布。所有与 UNI 的比较均在 43 种癌症类型分类和分别用 OT-43 和 OT-108 进行的 108 种 OncoTree 编码分类任务中进行评估。有关数据分布的更多详细信息,请参见补充表 4。生殖器(Gen.);胃肠道(GI)。 b,d, 在 OT-43(b)和 OT-108(d)任务中,UNI 与其他预训练编码器的宏平均 AUROC 比较(每个 n=1,620 幻灯片)。c,e, 在不同预训练数据规模(Mass-1K、Mass-22K、Mass-100K)下,UNI 在 OT-43(c)和 OT-108(e)任务中的 Top-1 准确率(每个 n=1,620 幻灯片)。f, 在 15 个弱监督幻灯片级分类任务中,UNI 及其比较的监督性能。虚线表示每个模型在所有任务中的平均性能。所有数据均以平衡准确度给出,ISUP 分级除外,后者以二次加权 Cohen 的κ给出。误差条表示 95% 置信区间,中心点对应于上述指定的每个指标的计算值。所有任务的详细结果提供在补充表 12-35 中。外部测试集(Ext.)。 g–j, 以 K∈{1, 2, 4, 8, 16, 32}幻灯片/类报告的四项任务的少量样本幻灯片级性能。g, RCC 亚型(训练,TCGA;测试,CPTAC-DHMC;n=872 幻灯片)。h, BRCA 细粒度亚型(BRACS,n=87 幻灯片)。i, 脑肿瘤粗粒度亚型(EBRAINS,n=573 幻灯片)。j, ISUP 分级(PANDA,n=954 幻灯片)。框表示模型性能的四分位数值(n=5 次运行),而晶须延伸至 1.5 倍四分位距离内的数据点。所有任务的少量样本结果在扩展数据图 1 中给出。
结果¶
计算病理学中预训练缩放定律¶
基础模型的关键特性在于其在训练数据集更大时,能够在各种任务上提供改进的下游性能。尽管诸如 CAMELYON16(2016 年淋巴结癌转移挑战赛)和 TCGA 非小细胞肺癌子集(TCGA-NSCLC)等数据集常用于使用弱监督多示例学习(MIL)算法基准测试预训练编码器,但这些数据集仅源自单一器官的组织幻灯片,并且常用于预测二进制疾病状态,这并不反映真实世界解剖病理学实践中所见的更广泛的疾病实体数组。相反,我们通过构建一个大规模的、层次化的、罕见癌症分类任务来评估 UNI 在多样组织类型和疾病类别上的泛化能力,该任务遵循 OncoTree 癌症分类系统。使用 BWH 内部幻灯片,我们定义了一个包括来自 43 种癌症类型的 5,564 个 WSIs 的数据集,进一步细分为 108 个 OncoTree 编码,每个 OncoTree 编码至少有 20 个 WSIs。根据 RARECARE 项目和国家癌症研究所的监测、流行病学和最终结果(NCI-SEER)计划的定义,108 种癌症类型中有 90 种被指定为罕见癌症。该数据集构成了两个诊断难度不同的任务的基础:43 类 OncoTree 癌症类型分类(OT-43)和 108 类 OncoTree 编码分类(OT-108)。这些大型多类分类任务的目标不一定是临床实用性,而是评估基础模型的能力和与其他模型相比的特征表示的丰富性。为了评估缩放趋势,我们还在不同的数据规模上预训练 UNI,将 Mass-100K 细分为 Mass-22K(1,600 万图像,21,444 WSIs)和 Mass-1K(100 万图像,1,404 WSIs)。我们还通过使用两种不同的 ViT 架构大小来评估模型规模:ViT-Base(或 ViT-B)和 ViT-Large(或 ViT-L)。最后,我们还评估了自监督学习算法的选择,与 MoCoV3(参考文献 24)进行了比较。对于弱监督幻灯片分类,我们遵循首先从 WSI 中包含组织的片段预提取补丁级特征的常规范例,使用预训练的编码器,然后训练基于注意力的 MIL(ABMIL)算法。为了反映这些任务的标签复杂性挑战,我们报告了 top-K 准确率(K = 1、3、5)以及加权 F1 分数和接收者操作特征曲线(AUROC)下的面积性能。有关 OT-43 和 OT-108 任务、实验设置、实施细节和性能的更多详细信息,请参见方法部分、补充表 1-11 和补充表 12-18。
总体而言,我们展示了 UNI 中自监督模型的模型和数据缩放能力,UNI 在 OT-43 和 OT-108 上的缩放趋势如图 2c,e 所示。在 OT-43 和 OT-108 上,我们观察到当使用 ViT-L 从 Mass-1K 缩放到 Mass-22K 时,top-1 准确率增加了 +4.2%(P < 0.001,双侧配对排列测试),在 OT-108 上的类似表现增加了 +3.5%(P < 0.001)。从 Mass-22K 缩放到 Mass-100K,性能进一步增加:OT-43 和 OT-108 分别增加了 +3.7% 和 +3.0%(P < 0.001)。使用 ViT-B,性能趋势类似,但从 Mass-22K 到 Mass-100K 性能趋于平稳(补充表 13 和 16)。补充表 14 和 17 显示了数据多样性和预训练长度的影响,两个任务从 50,000 到 125,000 的训练迭代显示出单调改进。总体而言,这些缩放趋势与许多应用于自然图像的 ViT 模型的发现一致,随着预训练数据集的增长,更大的 ViT 变体的性能得到改善。探索其他自监督学习算法,我们还在 Mass-1K 上训练了 MoCoV3(参考文献 24)(使用 ViT-L 和 ResNet-50 主干),其性能不如 DINOv2(补充表 18)。为了随着模型和数据大小的增加而提升性能,选择算法及其超参数在开发 CPath 基础模型中也很重要。
我们将在 Mass-100K 上预训练的 ViT-L 的 UNI 与 CPath 中使用的公开可用的预训练编码器进行比较,任务包括 OT-43 和 OT-108:在 ImageNet-1K 上预训练的 ResNet-50(参考文献 84);在 TCGA 和 PAIP(病理 AI 平台)上预训练的 CTransPath(参考文献 37);以及在 TCGA 上预训练的 REMEDIS(参考文献 38)。我们观察到 UNI 在所有基线上都有很大的优势。在 OT-43 上,UNI 达到了 93.8% 的 top-5 准确率和 0.976 的 AUROC,性能超过了下一个最好的模型(REMEDIS)+6.3% 和 +0.022(两者 P < 0.001)(图 2b 和补充表 12)。在 OT-108 上,我们观察到类似的性能提升幅度,分别为 +10.8% 和 +0.020(P < 0.001),超过了 REMEDIS(图 2c 和补充表 15)。总体而言,我们发现 UNI 能够在 OT-43 和 OT-108 上对罕见癌症进行分类,与所有预训练编码器相比,性能提升幅度很大。
弱监督幻灯片级分类¶
此外,我们研究了 UNI 在 15 个幻灯片级分类任务中的能力,这些任务包括乳腺癌转移检测(CAMELYON16)、前列腺癌国际泌尿病理学会(ISUP)分级(前列腺癌分级评估,PANDA)、心脏移植评估(BWH 内部幻灯片)以及脑肿瘤亚型分析(EBRAINS;代表由 RARECARE 项目定义的 30 种罕见癌症)等。类似于 OT-43 和 OT-108 的评估,我们使用 ABMIL 算法比较了从 UNI 和其他预训练编码器预提取的特征。鉴于 CTransPath 和 REMEDIS 使用几乎所有 TCGA 幻灯片进行训练,这些模型在 TCGA 任务上的报告性能可能受到数据泄漏的影响,因此可能不公平地被夸大了。有关幻灯片任务、实验设置和性能的更多细节,请参见方法部分、补充表 19-21 和补充表 22-35。
在所有 15 个幻灯片级任务中,UNI 始终优于其他预训练编码器(平均性能提高了 ResNet-50 的 +26.4%、CTransPath 的 +8.3% 和 REMEDIS 的 +10.0%),在对罕见癌症类型或具有更高诊断复杂性的任务上观察到更大的改进(图 2f)。在前列腺 ISUP 分级(PANDA)上,UNI 达到了 0.946 的二次加权 Cohen’s κ,比下一个最好的模型(REMEDIS)高出 +0.014(P < 0.05)(补充表 29)。在层次分类任务上(这些任务也涉及罕见疾病类别),例如胶质瘤生物标志物预测(2 类 IDH1 突变预测和 5 类组织分子亚型分析,使用 TCGA 和 EBRAINS)以及脑肿瘤亚型分析(12 类粗粒度和 30 类细粒度脑肿瘤亚型分析,使用 EBRAINS),UNI 超过了下一个最好的模型(CTransPath 或 REMEDIS),分别提高了 +2.0%(P = 0.076)、+6.4%(P = 0.001)、+19.6%(P < 0.001)和 +16.1%(P < 0.001)(补充表 31-34)。与 OT-43 和 OT-108 相似,我们发现 UNI 在评估涉及仅罕见癌症类型的脑肿瘤亚型分析任务时具有最大的影响。
在现有排行榜的比较中,我们发现使用 UNI 特征的 ABMIL 超过了许多复杂的 MIL 架构。在乳腺癌转移检测(CAMELYON16)上,ABMIL 配合 UNI 超过了此任务上所有最先进的 MIL 方法(补充表 36),并且是少数在原始挑战中无时间限制情况下表现优于人类病理学家的 MIL 结果之一(AUROC 为 0.966)。在进行详细比较的任务上,如前列腺 ISUP 分级(PANDA)和细胞介导的异体移植排斥(BWH-EMB),ABMIL 配合 UNI 的表现优于 WholeSIGHT 和 CRANE 等方法(补充表 37 和 38)。尽管许多这些比较由于使用带有 ImageNet 转移的 ResNet-50(ResNet-50IN)特征而不完全相同,但我们注意到他们提出的 MIL 架构通常是为解决这些具有挑战性的任务而专门开发的。我们的比较突出了具有更好预训练编码器相对于 MIL 架构的优势。
在基于大型公共数据集训练的基础模型中,数据污染是一个关注点。尽管在自监督训练期间可能没有将标签明确泄漏到模型中,但在评估测试集上预训练的模型可能会表现出乐观的偏见性能,这在其他 CPath 研究中已观察到。我们还将 UNI 与 CTransPath 和 REMEDIS 在 TCGA 测试集上进行了比较,涉及非小细胞肺癌(NSCLC)亚型分析、肾细胞癌(RCC)亚型分析、胶质瘤 IDH1 突变预测和胶质瘤组织分子亚型分析任务,观察到在领域内与领域外性能比较时性能下降。在 NSCLC 亚型分析上,REMEDIS 在 TCGA 评估上超过 UNI(97.3% 对比 94.7%),但在 CPTAC(临床蛋白质组肿瘤分析联盟)评估上表现不佳(79.0% 对比 96.3%)(补充表 23)。在胶质瘤 IDH1 突变预测上,CTransPath 和 REMEDIS 在 TCGA 评估上超过 UNI(89.1% 和 81.9% 对比 80.8%),但在 EBRAINS 评估上表现不佳(83.6% 和 79.2% 对比 85.6%)(补充表 31 和 32)。我们强调,数据污染仅存在于模型的使用方式中,而不在模型本身,这些模型已显示在 TCGA 独立的环境中转移性能良好。鉴于许多 CPath 研究使用 TCGA 来研究多种癌症类型,UNI 在开发公共组织学数据集和基准上的病理 AI 模型比 CTransPath 和 REMEDIS 更具灵活性。
标签效率的少量样本幻灯片分类¶
我们进一步评估了 UNI 在所有幻灯片级任务中的少量样本多示例学习(MIL)性能。少量样本学习是一种评估方案,用于研究模型在给定少量示例(每类 K 个训练样本,也称为支持或样本)时对新任务(C 类)的泛化能力。对于所有预训练的编码器,我们训练了一个 ABMIL 模型,每个类的训练样本 K∈{1, 2, 4, 8, 16, 32},由于罕见疾病类别中支持大小较小,K 限制在 32。鉴于性能可能会根据每个类选择的 K 个示例而波动,我们重复进行了五次实验,每次随机采样 C×K 个训练示例。关于少量样本 MIL 实验和性能的更多详细信息,请参见方法部分和扩展数据图 1。
在所有任务中,UNI 通常都优于其他预训练编码器,并且在标签效率上具有优势,尤其是在分类罕见疾病方面(图 2g-j 和扩展数据图 1)。在比较 UNI 与其他编码器的 4 样本性能时(使用中值性能),次优的编码器需要多达八倍的训练样本每个类别才能达到 UNI 的相同 4 样本性能。在前列腺 ISUP 分级(PANDA)上,UNI 在所有少样本设置中的标签效率始终是两倍(图 2j)。在诸如细粒度脑肿瘤亚型分析(EBRAINS)等具有挑战性的罕见癌症亚型分析任务上,UNI 的 4 样本性能大幅优于其他编码器,仅与 REMEDIS 的 32 样本性能相当(图 2i)。总体而言,我们对幻灯片分类任务的全面评估展示了 UNI 作为基础模型的潜力,该模型可以用于筛查罕见和代表性不足的疾病的组织病理学工作流程。
线性分类器中的监督 ROI 分类¶
除了幻灯片级任务外,我们还在 11 个 ROI 级任务上评估了 UNI,这些任务包括结直肠组织和息肉分类(CRC-100K-NONORM98, HunCRC99, UniToPatho100)、前列腺腺癌组织分类(2022 年自动化格里森分级挑战赛(AGGC)101)、全癌种肿瘤 - 免疫淋巴细胞检测(TCGA-TILS67)、32 类全癌种组织分类(TCGA Uniform Tumor68)等。为了评估和比较,我们在每个编码器的预提取特征上进行了逻辑回归和 K 最近邻(KNN)分析,这是一种常见的做法,称为线性探针和 KNN 探针,分别用于衡量预提取特征的区分性能力和表示质量。我们使用平衡精度评估所有任务,前列腺组织分类使用加权 F1 分数评估 101。关于 ROI 任务、实验设置和性能的更多细节,请参见方法部分和补充表 39-60。
在所有 11 个 ROI 级任务中,UNI 几乎在所有任务上都优于所有基线,线性探针的平均性能增加了 ResNet-50 的 +18.8%、CTransPath 的 +7.58% 和 REMEDIS 的 +5.75%(图 3a)。在 KNN 探针上,UNI 同样优于 ResNet-50、CTransPath 和 REMEDIS,平均性能增加了 +15.6%、+8.6% 和 +9.4%。我们在具有挑战性的任务上发现更大的增益,例如前列腺组织分类(加权 F1 分数,+0.131, P < 0.001; +0.020, P < 0.001; +0.027, P < 0.001)和食管癌亚型分析(+25.3%, P < 0.001; +10.1%, P < 0.001; +5.5%, P < 0.001)与其他三种预训练编码器相比。图 3b 显示了前列腺癌分级的 UNI 预测,其中一个简单的线性分类器训练有预提取 UNI 特征可以与病理学家的注释高度一致(扩展数据图 2)。在 32 类全癌种组织分类(其中 32 类中有 19 类为罕见癌症)上,UNI 达到了最高的整体平衡精度和 AUROC,分别为 65.7% 和 0.975,优于下一个最好的模型(REMEDIS)+4.7% 和 +0.017(均 P < 0.001)。
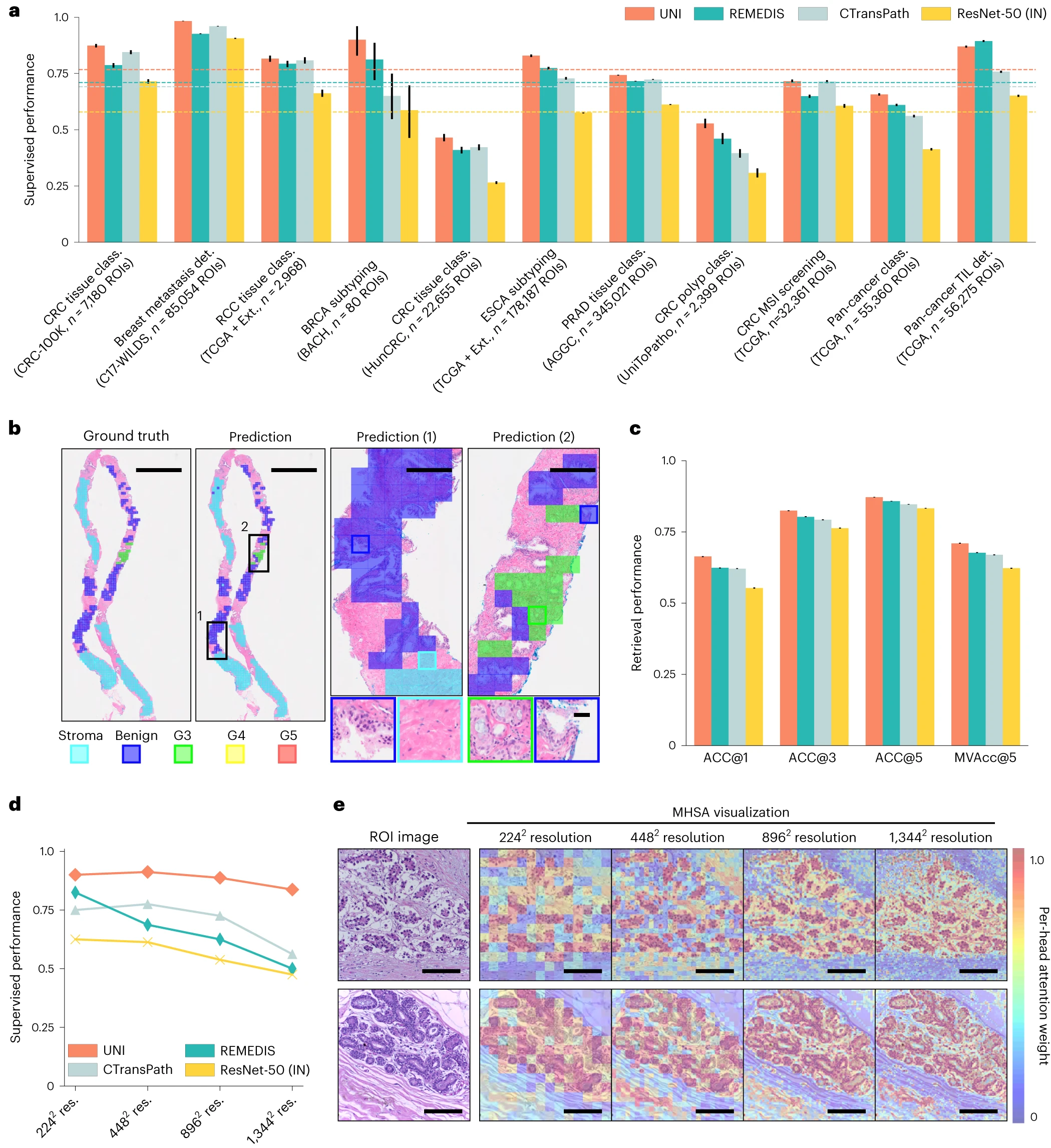
图三:a, UNI 在 11 个 ROI 级分类任务中的监督线性探针性能及其比较。除了前列腺腺癌组织分类使用加权 F1 分数外,所有结果均以平衡精度给出。虚线表示每个模型在所有任务中的平均性能。误差条代表 95% 置信区间,中心点对应于上述每个指标的计算值。所有任务的详细结果请见补充表 39-60。b, UNI 在 AGGC 的前列腺腺癌组织分类中的 ROI 分类示例。左图:在整片切片图像(WSI)上叠加的实地 ROI 级标签。右图:预测的补丁标签。为了更好的可视化,ROI 被放大,更多的比较显示在扩展数据图 2 中。c, UNI 在 AGGC 的前列腺腺癌组织分类中的 ROI 检索性能(n=345,021 个 ROI)。我们报告了 K∈{1, 3, 5}的召回率@K 以及平均召回率,误差条代表 95% 置信区间,中心点对应于每个指标的计算值。d, UNI 在 BACH 的乳腺癌亚型分析中不同图像分辨率(以像素为单位)下的监督 KNN 探针性能(n=80 个 ROI)。所有任务的检索性能请见扩展数据图 3 和补充表 63-68。 e, UNI 在 BACH 中不同图像分辨率下的多头自注意力(MHSA)热图可视化。每个彩色方块代表由 UNI 编码的 16×16 像素补丁令牌,热图颜色对应于该补丁令牌对全局 [CLS](即分类)令牌的注意力权重,位于 UNI 倒数第二层。上下图分别为:侵袭性和正常标记图像的可视化,更多的可视化和解释请见扩展数据图 4-6。刻度尺:b, 实地真相和预测,2 mm;预测 (1) 和预测 (2),200 µm;插图,30 µm;e, ROI 图像,32 µm;2242,64 像素;4482,128 像素;8962,256 像素;1,3442,384 像素。
我们还将 UNI 的性能与官方排行榜上的性能进行了比较。对于肿瘤 - 免疫淋巴细胞检测,与 ChampKit 基准中的最佳模型相比,该模型的 AUROC 为 0.974,假阴性率(FNR)为 0.246,而 UNI 的 AUROC 为 0.978,FNR 为 0.193(未进行染色标准化)(补充表 61)。对于乳腺癌转移检测(CAMELYON17-WILDS 排行榜),与迄今为止最佳的模型相比,该模型在域外验证和测试集上的准确率分别为 95.2% 和 96.5%,而 UNI 分别达到了 97.4% 和 98.3%(补充表 62)。我们注意到,这些比较中的许多都是通过从自然图像(而非病理学)进行迁移学习进行端到端的微调。虽然这些实验与 UNI 的不完全相同,但这些比较突出了 UNI 的多功能性,即使用线性分类器的开箱即用评估能与使用端到端微调的最先进技术竞争。
ROI 检索¶
除了使用 UNI 中的表示来构建特定任务的分类器外,这些表示还可以用于图像检索。检索与 KNN 类似,我们评估查询图像检索同一类别的其他图像的效果,假设在表示空间中视觉上相似的图像应该比视觉上不同的图像更接近。与 KNN 评估不同,我们考虑检索的准确性,即 Acc@K,其中 K∈{1, 3, 5},如果正确标记的图像位于检索到的前 K 个图像中,则检索成功,并且 MVAcc@5,使用前 5 个检索图像的多数票。我们在六个至少有 5 个类别的 ROI 级任务上评估组织学图像检索。关于 ROI 检索实验和性能的更多细节,请参见方法部分,扩展数据图 3 和补充表 63-68。
UNI 在所有任务上都优于其他编码器,展示了在不同设置下的卓越检索性能。在 PRAD 组织分类(AGGC)上,UNI 在 Acc@1 和 MVAcc@5 上分别比下一个最佳表现的编码器(REMEDIS)高出 +4% 和 +3.3%(两者 P<0.001)(图 2c)。在结直肠癌(CRC)组织分类(CRC-100K)上,顶级表现的编码器之间的差距相对较小(与 REMEDIS 相比分别高出 +3.1%,P<0.001 和 +0.01%,P=0.188),可能是因为不同组织类型具有非常独特的形态学,如线性探针中的相对高分类性能所示。在包含许多罕见癌症类型的更具挑战性的 32 类全癌种组织分类任务上,UNI 比第二好的表现编码器(REMEDIS)的领先幅度更大,Acc@1 和 MVAcc@5 分别高出 +4.6% 和 +4.1%(两者 P<0.001)。
对高图像分辨率的鲁棒性¶
虽然视觉识别模型通常在调整大小后的 224×224 像素(2242 像素)图像上进行评估,但图像调整大小会改变每像素微米数(mpp),并可能改变细胞异型性等形态特征的解释。我们研究了在乳腺浸润性癌(BRCA)亚型分析(乳腺癌组织学图像大挑战,BACH)(2242 像素,2.88 mpp 到 13442 像素,0.48 mpp)和结直肠息肉分类(UniToPatho)(2242 像素,3.60 mpp 到 17922 像素,0.45 mpp)中,UNI 中的特征质量在不同分辨率下如何受影响,采用线性和 KNN 探针。关于多分辨率实验和性能的更多细节,请参见方法部分,扩展数据图 4 和补充表 45, 46, 51 和 52。
在这两个任务上,我们展示了 UNI 对不同图像分辨率的鲁棒性,以及高分辨率 ROI 任务中图像调整大小引入的偏见。在扩大用于评估的图像分辨率时,我们观察到其他编码器的性能退化更严重,在 BRCA 亚型分析中,CTransPath 和 REMEDIS 的 KNN 性能分别下降了−18.8% 和−32.5%(2242 像素对比 13442 像素),而 UNI 只下降了−6.3%。在结直肠息肉分类中,虽然其他编码器没有显著性能下降(2242 像素对比 17922 像素),但 UNI 通过 KNN 探针增加了 +5.1%。图 2e 和扩展数据图 5 和 6 展示了 UNI 在评估高分辨率图像时如何突出更细粒度的视觉特征。在结直肠息肉分类中,调整到 2242 像素会模糊定位隐窝的重要细粒度细节,而这些细节在 UNI 的高分辨率下则可以被检测到。这些观察表明,UNI 可以编码在大多数图像分辨率下语义上有意义的表示,这在 CPath 任务中非常有价值,这些任务在不同的图像放大倍数下是最佳的。
ROI 细胞类型分割¶
我们在最大的公共 ROI 级分割数据集 SegPath102 上评估 UNI,该数据集用于分割肿瘤组织中的八种主要细胞类型:上皮细胞、平滑肌细胞、红细胞、内皮细胞、白细胞、淋巴细胞、浆细胞和髓细胞。所有预训练的编码器都使用 Mask2Former103 进行端到端的微调,这是一个灵活的框架,通常用于评估预训练编码器的现成性能。鉴于 SegPath 数据集将细胞类型分为独立的密集预测任务(总共八个任务),每个编码器都分别针对每种细胞类型进行单独的微调,使用骰子得分作为主要评估指标。关于分割任务和性能的更多细节,请参见方法部分和补充表 69。
尽管像 Swin 变换器(CTransPath)和卷积神经网络(CNN;ResNet-50 和 REMEDIS)这样的分层视觉主干在分割上有众所周知的优势,我们观察到 UNI 仍然在 SegPath 中的大多数细胞类型上胜过所有比较。在上皮、平滑肌和红细胞类型的个别分割任务中,UNI 分别获得了 0.827、0.690 和 0.803 的骰子得分,分别比下一个最好的表现编码器(REMEDIS)高出 +0.003(P=0.164)、+0.016(P<0.001)和 +0.008(P=0.001)。在 SegPath 的所有八种细胞类型中,UNI 以平均 0.721 的骰子得分达到了整体最佳性能,超过了 ResNet-50(0.696)、CTransPath(0.695)和 REMEDIS(0.716)。扩展数据图 7 显示了 UNI 和其他编码器对所有细胞类型的分割可视化,所有比较在匹配地面真相分割方面表现良好。总体而言,我们发现 UNI 可以在分割任务上胜过最先进的 CNN 和分层视觉模型,扩展了其在非常规设置中的多功能性。
少样本 ROI 分类及类原型¶
与幻灯片级分类类似,我们还评估了 UNI 在 ROI 级任务上的标签效率。我们使用非参数化 SimpleShot 框架评估所有预训练的编码器,这是少样本分类文献中的一个强基准,它提出将每个类的提取特征向量平均作为 K=1 最近邻(或最近质心)分类中的支持示例。这些平均特征向量也可以被视为“类原型”,一组独特的一次性示例,代表诸如类标签的语义信息(例如,肺腺癌(LUAD)与肺鳞状细胞癌(LUSC)的形态学)。在测试时,未见测试样本通过欧几里得距离被分配到最近的类原型的标签(图 4a)。对于所有预训练的编码器,我们使用 SimpleShot 评估其预提取特征,其中 K∈{1, 2, 4, 8, …, 256}训练样本每类用于大多数任务,实验在 1000 次运行中重复,每次运行随机抽样 C×K 训练样本。关于少样本 ROI 实验和性能的更多细节,请参见方法部分和扩展数据图 8。
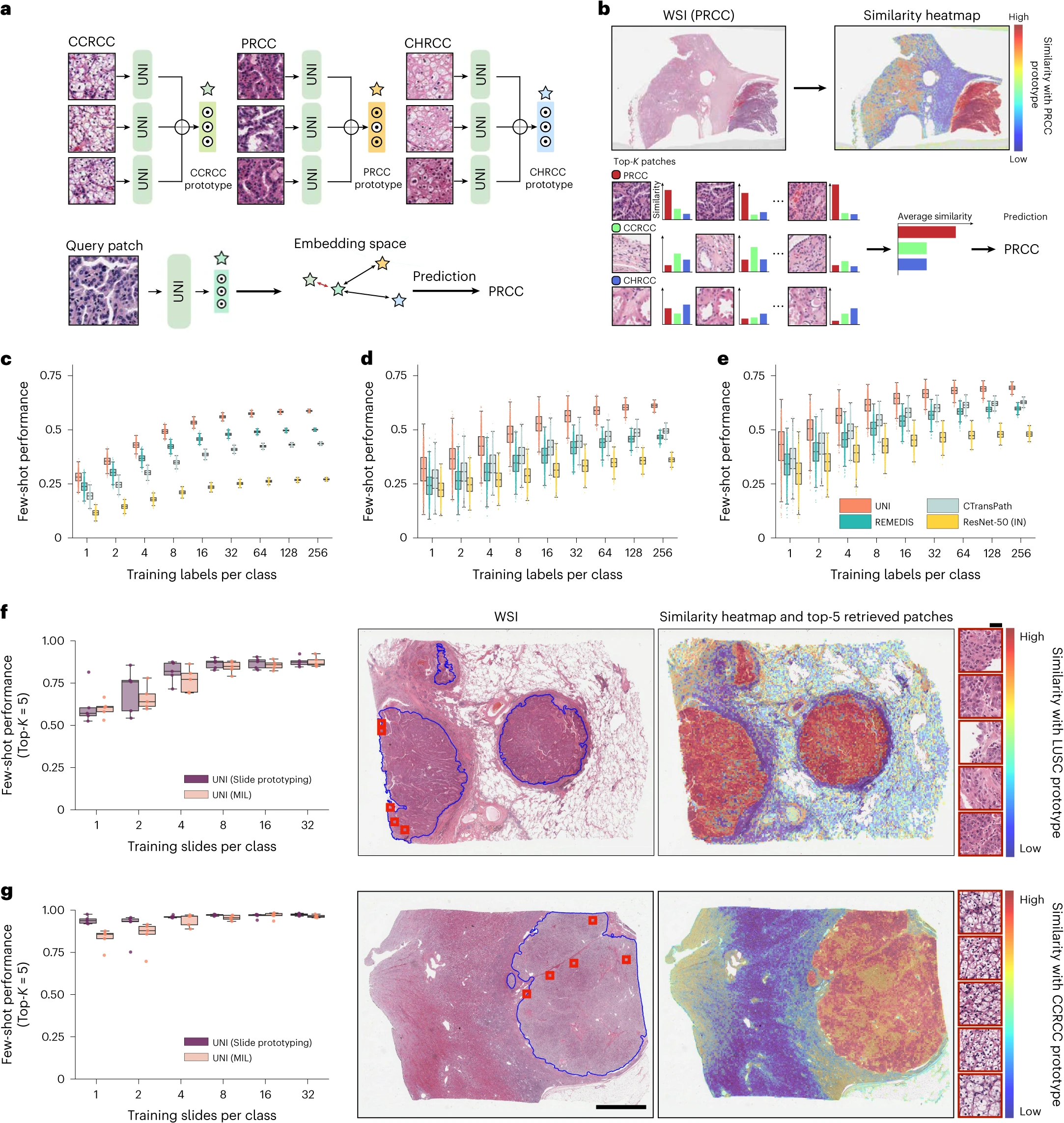
图四:a, 通过 SimpleShot 进行原型的少样本 ROI 分类。通过对同一类别的 ROI 提取的特征进行平均,构建一个类原型。对于测试 ROI,SimpleShot 将最相似的类原型(欧氏距离最小)的类别指定为预测的 ROI 标签。
b, 通过 MI-SimpleShot 进行原型的少样本幻灯片分类。使用预先计算的 ROI 级类原型集(与幻灯片共享相同的类别标签),MI-SimpleShot 使用类原型中平均相似度最高的前 K 个补丁来预测幻灯片标签。相似性热图可视化了真实类原型与 WSI 中每个补丁的相似性。
c-e, 通过 SimpleShot 在三个任务上的少样本 ROI 分类性能,箱形图显示了模型性能的四分位数(n=1000 次运行),晶须延伸到 1.5 倍四分位范围内的数据点。c, 全癌种组织分类(TCGA, n=55,360 个 ROI)。d, 结直肠息肉分类(UniToPatho, n=2,399 个 ROI)。e, 前列腺腺癌组织分类(AGGC, n=345,021 个 ROI)。所有任务的少样本 ROI 性能在扩展数据图 8 中提供。
f,g, 通过 MI-SimpleShot 进行的少样本幻灯片分类性能和相似性热图,用于 NSCLC 亚型分析(训练,TCGA;测试,CPTAC;n=1,091 张幻灯片)(f)和 RCC 亚型分析(训练,TCGA;测试,CPTAC-DHMC;n=872 张幻灯片)(g)。在这两个任务中,使用从 UNI 提取的特征,我们将 MI-SimpleShot 与 ABMIL 在相同的少样本设置下进行比较(箱形图显示了模型性能的四分位数,n=5 次运行,晶须延伸到 1.5 倍四分位范围内的数据点),并可视化相似性热图和前 5 个相似补丁(用红色边框标出),分别针对 LUSC(f)和 CCRCC(g)幻灯片。刻度尺:WSI,2 mm;前 5 个检索补丁,56 µm。更多的细节、比较和可视化在方法部分和扩展数据图 8-10 中提供。
在多种任务和评估设置中,我们发现 UNI 是一个强大的少样本学习者,并且比其他预训练的编码器具有更高的标签效率。当比较 UNI 的中位 8 样本性能与其他编码器时,UNI 在许多任务中的表现一致超过下一个最佳表现编码器的 128 样本和 256 样本性能(图 4c-e 和扩展数据图 8)。我们注意到,由于随机选择作为原型的 ROI,所有编码器在 1 样本和 2 样本性能的变异性可能很高,这可能受到 H&E 染色变异性的影响。然而,随着形成类原型的支持示例数量的增加,我们观察到少样本性能运行的变异性单调减少(在 UNI 的 256 样本性能中跨任务的标准偏差为 0.32-1.59%),这证明了在 SimpleShot 中将训练示例随机平均为类原型时的性能稳定性。尽管如此,我们观察到 UNI 的最低少样本性能有时甚至超过其他编码器在 1000 次运行中报告的最大少样本性能。在全癌种组织分类中,UNI 在 2 样本、8 样本和 32 样本评估中的最低性能分别优于 ResNet-50、CTransPath 和 REMEDIS 的最佳可能性能。这些发现表明了 UNI 的卓越标签效率和表示质量,只需平均少数几个 ROI 的提取特征就能创建有效的类原型。
基于提示的幻灯片分类使用类原型¶
虽然通过 MIL 的弱监督学习已将幻灯片级分类转变为不再需要 ROI 注释,但获取和策划组织学幻灯片集可能仍然是处理罕见和代表性不足疾病的临床任务的障碍。从观察 UNI 在检索性能和少样本能力上的表现,我们重新审视了使用类原型的少样本幻灯片分类问题。类似于文本提示,我们也将 SimpleShot 的类原型用作对 WSI 中检索到的前 K 个补丁(顶部 K 池化)进行多数投票的“提示”,我们称之为多示例 SimpleShot(MI-SimpleShot)(图 4b)。我们在与训练 ABMIL 模型相同的数据集上评估 MI-SimpleShot 在少样本幻灯片分类中的表现,使用来自全癌种组织分类任务的训练幻灯片的注释 ROI 创建原型。我们还比较了使用其他预训练编码器的 MI-SimpleShot 以及 UNI 的 MIL 基线。我们还开发了相似性热图,显示幻灯片中所有补丁与真实标签的类原型的标准化欧氏距离,用蓝色标出与幻灯片标签相匹配的组织区域的病理学家注释。关于 MI-SimpleShot 实验和性能的更多细节,请参见方法部分,扩展数据图 9 和 10 以及补充表 70 和 71。
仅使用每个类别的少数几个注释 ROI 示例作为原型,我们展示了使用 UNI 与 MI-SimpleShot 作为简单但高效的系统进行幻灯片级疾病亚型分析和检测的潜力。在 NSCLC 和 RCC 亚型分析(在 TCGA 上训练,在外部队列上测试)上,MI-SimpleShot 在使用 1、2 和 4 张训练幻灯片每类创建原型时的表现优于 ABMIL,在使用更多幻灯片时表现类似于 ABMIL(图 4f,g)。使用相似性热图,我们还观察到 UNI 检索到的与幻灯片标签相符的补丁与病理学家的注释高度一致,如图 4f,g 右侧所示,用于 LUSC 和清晰细胞肾细胞癌(CCRCC)幻灯片。我们认为 MI-SimpleShot 的有效性可以归因于不需要可训练参数(ABMIL 模型可能在少样本设置中仍会过度拟合和欠拟合)以及 UNI 特征在 ROI 检索上的强大表示质量。尽管其他预训练编码器可以用于在 MI-SimpleShot 中学习原型,但 UNI 可能对 H&E 染色的变异性不太敏感。这在 RCC 亚型分析的一次性表现的高标准偏差中得到体现(在扩展数据图 1 中的 ABMIL 和扩展数据图 9 中的 MI-SimpleShot),其中 MI-SimpleShot 仅使用一个地点学习类原型。这也在 CAMELYON17-WILDS 的乳腺转移检测的 SimpleShot 评估中得到强调,CTransPath 和 REMEDIS 在两个域外医院测试队列之间的性能差异较大(准确度差异分别为 12.3% 和 12.8% 与 5.1%),暗示 H&E 染色强度可能影响检索性能(补充表 42)。在扩展数据图 10 中,我们观察到预测标签与病理学家注释相比检索性能的不正确实例。总体而言,我们通过 MI-SimpleShot 对 UNI 的评估展示了具有强大检索能力的视觉中心基础模型如何在解剖病理学应用中发挥作用。
讨论¶
在这项研究中,我们展示了 UNI 的多功能性,这是一种通用的、自监督的模型,预训练于迄今为止在计算病理学(CPath)中最大的组织切片集合之一。我们策划了包含超过 1 亿个组织片段的预训练数据集 Mass-100K,这些片段来自 100,426 个 WSI,涵盖了 20 种主要器官类型,包括正常组织、癌变组织和其他病理。使用 DINOv2 自监督学习方法(已证明能扩展到大型数据集),我们开发并验证了在 Mass-100K 上预训练的 ViT-L,该模型在各项任务上一致超越其他组织病理学图像编码器。尽管在某些任务上 CTransPath 和 REMEDIS 可能达到相似的表现,但我们的发现表明这些编码器在检索能力、标签效率以及在域外评估中对 H&E 染色强度的潜在偏见方面存在限制。
作为一种以视觉为中心的基础模型,UNI 可能使 CPath 中的多种临床应用成为可能,我们在开发 UNI 时遇到了几个挑战,这些挑战涉及模型和数据规模如何影响迁移性能等因素。尽管许多实证研究探索这些组件以实现自然图像的良好泛化,但许多解决方案可能由于病理学图像与自然图像之间的差异而无法转化。例如,尽管 MoCoV3 在 ImageNet 上的表现低于 DINOv2 但仍具有竞争力,相同的训练配置在 Mass-1K 上开发 ViT-L 时显示出在 OT-108 上的大幅性能差距。继我们的研究之后,我们注意到最近出现了一些在更大的组织切片数据集和收藏上进行训练的其他研究。与以往和近期的工作不同,我们的研究在自监督模型的扩展定律和迁移学习能力方面为 CPath 提供了独特的见解。尽管模型和数据规模是构建以视觉为中心的自监督学习的重要组成部分,我们发现自监督学习(SSL)算法选择是最具影响力的,MoCoV3(在 Mass-1K 上的 ViT-L)的表现不仅不如其 DINOv2 的对应物,而且还不如 CTransPath 和 REMEDIS。增加模型规模(从 ViT-B 到 ViT-L)和数据规模(从 Mass-1K 到 Mass-100K)确实反映了性能的提升,但请注意,UNI 在 OT-43 和 OT-108 上的性能相对接近,并且相对于 CTransPath 和 REMEDIS 持续改进,这表明仍然可以使用较小的模型和较少的数据开发具有竞争力的预训练编码器。与 UNI 展示的许多临床应用并行,我们相信我们对上述因素的测试将指导 CPath 从业者使用私有的内部幻灯片集合开发自己的基础模型。
关于 UNI 可以应用于的广泛临床任务,与其他编码器相比,我们发现 UNI 在分类罕见和代表性不足的疾病方面表现出色,如 OT-108 基准中的 90 种罕见癌症类型、EBRAINS 数字肿瘤图谱中的 30 种罕见脑肿瘤诊断,以及从 TCGA 来源的 32 种癌症亚型中的 19 种。在这些任务和其他任务上,UNI 一致且显著地超过了下一个最佳表现的编码器(REMEDIS 或 CTransPath)。我们推测 UNI 的表现归功于预提取特征的强大表示质量,如在使用类原型的少样本 ROI 和幻灯片分类中所见。在弱监督范式中,罕见癌症类型在当前幻灯片数据集中不常见且代表性不足,使用 UNI 的 MI-SimpleShot 表明,注释每个类别的四张幻灯片可以胜过特定任务的 MIL 算法。总体而言,我们认为 UNI 和其他正在开发的以视觉为中心的基础模型可以在使需要大量数据的创新临床应用成为可能方面具有变革性。
在与公共排行榜的比较中,我们认为 UNI 也代表了从特定任务模型开发到 CPath 中通用 AI 模型的重要转变。除了本研究评估的 34 个临床任务外,与其他工作的公布结果相比,开箱即用的 UNI 具有竞争力,超过了通常经过端到端训练或使用为解决这些特定公共挑战而精心设计的训练配方的领先模型。总之,我们的发现突出了拥有更好的预训练编码器的优势,而不是开发针对狭窄临床问题的特定任务模型,我们希望这将引导 CPath 研究方向转向开发通用 AI 模型,这些模型在针对病理学中的多样化临床应用时将具有更大的性能和灵活性。按照计算机视觉中自监督模型的传统命名,诸如“基础模型”之类的标签可能会造成误导性期望。
我们的研究有几个局限性。基于 ViT-L 架构的 UNI 缺乏解决 CPath 中密集预测任务的视觉特定偏见,我们注意到在 SegPath 中细胞类型分割的性能提升并不像在其他任务中观察到的那样显著。我们预见随着更好的适用于分割任务的 ViT 架构适配方法的出现,性能将进一步提升。我们的研究还未评估在 DINOv2 中表现最佳的 ViT-Giant 架构,这是一个更大的模型,可能在 CPath 中表现良好,但需要更多的预训练计算资源。虽然我们的研究组织了迄今为止(据我们所知)评估 CPath 中预训练模型的最大的临床任务集合,但其他临床任务,如细胞病理学或血液病理学,在我们的分析中没有代表。由于我们的评估范围广泛,而且某些任务的验证集很小(或缺失),超参数是固定的,这遵循了 CPath 中其他工作的做法。进一步的超参数调整和其他训练配方可能会进一步改善结果;然而,我们的评估协议是为了排名预训练编码器主干的表示质量而实施的。在开发 UNI 的过程中,虽然 Mass-100K 是有意开发的,以避免与大多数公共组织学收藏重叠显著,但如果相同模型在许多应用中被重复使用,尤其是如果它可能对不同人群产生不同影响的话,数据污染和图像获取偏移等偏见应进一步研究。UNI 是一个用于 CPath 的单模态模型,意味着跨模态检索和视觉问答等多模态能力超出了研究范围,我们在并行工作中探索这些能力。最后,UNI 也只是一个用于 CPath 的 ROI 级模型,而病理学中的大多数临床任务是在幻灯片或患者级别进行的。未来的工作将专注于使用 UNI 作为幻灯片级自监督模型的构建模块,以及在解剖病理学中开发通用幻灯片级病理 AI。
方法¶
大规模视觉预训练¶
麻省总医院布莱根妇女医院机构审查委员会批准了本研究中使用的内部病理图像及相应报告的回顾性分析。所有内部数字数据,包括全幅切片图像(WSIs)、病理报告和电子病历在计算分析和模型开发前均已去标识化。患者没有直接参与或被招募进行本研究。针对档案病理切片的回顾性分析免除了知情同意。
在 CPath 中开发和评估自监督模型的一个重要且相对较少讨论的挑战是开发可用于公共组织学数据集评估的大规模模型的难度。对于自然图像,ImageNet-1K 是自监督学习方法的模型开发和评估生命周期的一个集成数据集。具体来说,模型首先在 ImageNet-1K 的训练集上进行预训练,然后通过微调和线性探测性能在验证集上进行评估(作为测试集处理),作为社区接受的“适配度”报告,通过其他下游任务如细粒度分类和活动视频识别进一步评估泛化性能。虽然这些现成的自监督学习方法可以轻松适应 CPath,但我们注意到,CPath 的公共数据用于预训练的数量远少于自然图像,并且在大型公共组织学幻灯片集合上的预训练也限制了它们适应公共 CPath 基准的能力。特别是,许多自监督病理学模型的开发仅限于在 TCGA 上进行预训练,TCGA 是 CPath 中最大和最多样化的公共组织学数据集之一,许多模型选择使用整个 TCGA 集合来实现自监督学习中的数据规模优势。然而,由于许多流行的 CPath 临床任务也源自 TCGA(例如,全癌种分析)等,它们适用于公共 CPath 基准的可能仅限于归纳推理,因此广泛评估领域外的泛化性能是有限的。尽管像 CAMELYON 和 PANDA 这样的数据集可用于评估 TCGA 预训练模型,但我们注意到这些数据集仅限于单一组织类型且疾病类别有限。
Mass-100K 数据集策划¶
为了克服这一限制,我们开发了 Mass-100K,这是一个大规模且多样化的预训练数据集,包含来自 MGH 和 BWH 的内部组织学幻灯片,以及来自 GTEx 联盟的外部组织学幻灯片。借鉴自然图像数据集的做法,我们还创建了三个不同大小的 Mass-100K 分区,以评估数据规模定律,这是自然语言和图像基础模型中的一个经验观察,即数据集大小的扩展也会提升模型性能。类似于 ImageNet-22K 和 ImageNet-1K,我们开发了包含 16,059,454 个组织学图像片段的 Mass-22K 数据集,这些片段来自 21,444 个诊断用甲醛固定石蜡包埋(FFPE)H&E 染色的 WSI,涵盖 20 种主要组织类型,主要是癌症组织,以及它的子集 Mass-1K(1,064,615 图像,1,404 个 WSI)。所有 Mass-22K 和 Mass-1K 中的组织学幻灯片均来自 BWH,并使用 Aperio GT450 扫描仪或 Hamamatsu S210 扫描仪扫描。为使图像数据集大小大致等同于 ImageNet-22K 和 ImageNet-1K,我们从每个 WSI 的组织学区域中抽取大约 800 个图像片段,图像分辨率为 256×256 像素,放大倍数为 20 倍。对于幻灯片预处理,我们采用了 CLAM 工具箱中的 WSI 预处理方法,该方法通过 RGB→HSV 颜色空间中饱和度通道的二值阈值分割来进行低分辨率的组织分割;中值模糊、形态闭合和过滤面积低于最小区域的轮廓以平滑组织轮廓并去除伪影;以及在每个 WSI 的分割组织区域中提取不重叠的 256×256 组织片段的坐标。Mass-22K 和 Mass-1K 中主要组织类型的分布分别在补充表 2 和 3 中给出。
对于在 Mass-1K 和 Mass-22K 上进行的小规模视觉预训练,我们使用了 iBOT,该方法具有与上文中介绍的 DINOv2 相同的损失目标。我们注意到,iBOT 和 DINOv2 是同一系列 ViT 预训练技术中的重叠方法,因为两种方法都扩展了原始的 DINO 方法(该方法为 ViT 引入了学生 - 教师知识蒸馏),iBOT 通过引入在线分词器组件用于遮蔽图像建模来扩展 DINO,而 DINOv2 则通过引入额外的修改来扩展 iBOT,从而提高了大型 ViT 架构的训练稳定性和效率。这六种修改可以概括如下:解除上述损失目标之间头部权重的绑定,而不是像 iBOT 那样绑定这些目标;使用 Sinkhorn-Knopp 居中而不是 iBOT 中执行的教师 softmax 居中;引入 KoLeo 正则化以提高令牌多样性;在预训练末期进行高分辨率微调;改进的代码实现,实现了 FlashAttention,全分片数据并行训练和高效的随机深度;以及在大规模数据集上对 ViT-Large 架构的改进预训练配方。最后,尽管 iBOT 和 DINOv2 使用相同的两个损失目标,但这些方法的训练配方是为不同的数据规模开发的:iBOT 是为 ImageNet-1K 和 ImageNet-22K 上的 ViT-Base 和 ViT-Large 模型开发的,而 DINOv2 是为 LVD-142M 上的 ViT-Large 和 ViT-Giant 模型开发的,LVD-142M 是一个包含 1.42 亿精选自然图像的数据集。为了利用 DINOv2 中在大规模数据集上对 ViT-Large 的改进训练配方,同时也使与 iBOT 训练的 ViT-Base 模型的比较公平,我们排除了 DINOv2 中修改 iBOT 损失目标的前两个修改(解绑头部权重和使用 Sinkhorn-Knopp 居中),如补充表 5 中所述。高分辨率微调也在预训练的最后 12,500 次迭代中进行(总共 125,000 次迭代)。
评估设置¶
比较和基准¶
在幻灯片和 ROI 级评估中,我们将 UNI 与 CPath 社区中常用的三种预训练编码器进行比较。对于与 ImageNet 迁移的模型进行比较,我们使用了在 ImageNet 上预训练的 ResNet-50(在第三个残差块后截断,具有 8,543,296 个参数),这是许多幻灯片级任务中常用的基准。与当前最先进的编码器相比,我们比较了 CTransPath,它是一种使用窗口大小为 14 的“微型”配置的 Swin 变换器(Swin-T/14,28,289,038 个参数),主要在 TCGA 上通过 MoCoV3 预训练,以及 REMEDIS,这是一个初始化时使用“大转移”- 中型协议在 ImageNet-22K 上预训练然后使用 SimCLR 进行的 ResNet-152×2(232,230,016 个参数)。关于数据分布,CTransPath 使用 TCGA 的 29,753 个 WSI(包括 FFPE 和冷冻组织切片)和 PAIP 的 2,457 个 WSI(跨六个解剖部位),共使用了 15,580,262 个组织片段和 32,120 个 WSI 进行预训练。REMEDIS 使用从 TCGA 的 29,018 个 WSI 随机抽样的约 5000 万个片段进行预训练。在自监督学习中,CTransPath 使用 MoCoV3 算法训练了 100 个周期,预训练期间大约看到了 1.56×10^9(或 15.6 亿)图像,而 REMEDIS 使用 SimCLR 算法训练了最多 1000 个周期,看到的图像超过 50×10^9(或 500 亿)。在这些预训练编码器的实现中,我们使用了 CLAM 提供的截断的 ResNet-50 实现,并使用了 CTransPath 和 REMEDIS 的官方模型检查点。这些模型输出的图像嵌入分别为 1024、768 和 4096。与 ResNet-50 和其他 ResNet 模型类似,在分类头之前的倒数第二个特征层是 [1×7×7×4096] 维的网格状特征图,我们应用二维(2D)自适应平均池化层输出单个 [1×4096] 维图像嵌入。对于所有用于 ROI 任务的图像和从幻灯片任务中提取的片段,跨所有模型,所有特征提取操作都在放大 20 倍的 224×224 图像上进行。我们注意到,CTransPath 使用的 Swin-T/14 架构有限制,它只能接受长度可被 224 整除的图像尺寸。我们还注意到,尽管 CTransPath 是在 10 倍放大下预训练的,但它在 20 倍放大下表现出了最先进的性能。所有预训练的编码器都使用 ImageNet 的平均值和标准差参数进行图像标准化(包括 UNI)。为了与通用病理任务的迁移学习进行比较,我们还在 TCGA 的 32 类全癌种组织分类任务上端到端训练了一个 ViT-L/16 架构(用 ImageNet-22K 迁移初始化)。在几个基准测试任务中,我们注意到这种削减研究的性能比 UNI 差,即使是在域内任务如 TCGA 中的全癌种肿瘤 - 免疫淋巴细胞检测中也是如此(补充表 72)。
最后,我们注意到虽然许多幻灯片和 ROI 任务是使用 TCGA 的注释数据创建的,但 CTransPath 和 REMEDIS 也使用了 TCGA 中几乎所有的幻灯片进行训练,这可能导致信息泄露,使这些模型在 TCGA 基准测试中的性能被夸大。在可能的情况下,我们报告了所有任务在 TCGA 之外的外部队列上的评估。鉴于官方的训练 - 验证 - 测试划分可能都是使用 TCGA 开发的,这对所有任务来说可能并不可行。
弱监督幻灯片分类¶
弱监督幻灯片分类任务的训练和评估遵循传统的两阶段 MIL 范式,包括从 WSI 的分割组织区域的非重叠组织片段中预提取 ROI 级特征作为实例,以及学习一个可训练的排列不变池化算子,将片段级(或实例)特征聚合为单一幻灯片级(或包)特征。对于幻灯片预处理,我们使用了在数据策划部分描述的相同 WSI 预处理流程,该流程使用 CLAM 工具箱,使用预训练编码器在打补丁的坐标上进行额外的片段特征提取。图像缩小至 224×224 像素并使用 ImageNet 的平均值和标准差参数进行标准化。作为质量控制,我们执行了以下额外步骤:首先,对于组织掩模分割不足或过度的幻灯片,我们调整了 CLAM 中的分割参数(阈值和降采样级别)以仅分割组织区域;其次,我们移除了非 H&E 和非 FFPE 的幻灯片;第三,对于 WSI 金字塔格式中没有相当于 20 倍放大的降采样级别的幻灯片,我们将组织打成非重叠的 512×512 像素组织片段,放大 40 倍,然后在特征提取期间将这些图像缩小至 224×224 像素。所有预训练编码器使用的预提取特征都使用了相同的 WSI 坐标集进行特征提取。
在弱监督学习中比较预训练编码器的预提取特征时,我们在所有任务中使用了 ABMIL 算法,这是幻灯片分类任务中的一个典型弱监督基线。我们使用具有两层门控的 ABMIL 架构,所有输入嵌入都映射到第一个全连接层的 512 维嵌入维度,然后是中间层的 384 维隐藏维度。为了正则化,我们在输入嵌入上使用 P=0.10 的 dropout,在每个中间层之后使用 P=0.25 的 dropout。除了第一个全连接层依赖于预提取特征的嵌入维度外,所有比较都使用相同的 ABMIL 模型配置。我们使用 AdamW 优化器训练所有 ABMIL 模型,使用余弦学习率调度器,学习率为 1×10^-4,交叉熵损失,最多 20 个周期。我们还在验证损失上执行了早停,如果有验证折叠的话。对于所有幻灯片分类任务,我们按病例和标签分层将幻灯片数据集划分为训练 - 验证 - 测试折叠,或使用官方折叠(如果有的话)。鉴于 CTransPath 和 REMEDIS 是使用 TCGA 中的所有幻灯片进行预训练的,我们考虑了可能进行额外外部评估的 TCGA 幻灯片任务(例如,由于 CPTAC 中有 LUAD 和 LUSC 幻灯片,包括了 NSCLC 亚型分析,而 BRCA 亚型分析则被排除)。对于胶质母细胞瘤 IDH1 突变预测和组织分子亚型分析,训练 - 验证 - 测试折叠还额外进行了站点分层,以减轻潜在的批次效应。
评估设置¶
线性探测与 K- 最近邻探测在 ROI 分类中的应用¶
对于 ROI 级别的分类任务,我们遵循先前的工作,分别使用逻辑回归(线性探测)和 K- 最近邻探测(KNN 探测)来评估预提取特征嵌入在下游任务中的判别性迁移性能和表示质量。在线性探测中,根据自监督学习社区推荐的做法,我们将ℓ2 正则化系数λ固定为 \( \lambda = \frac{1}{M} \),其中 M 是嵌入维度,C 是类别数,并使用 L-BFGS 求解器,最大迭代次数设为 1000。KNN 探测是自监督社区提倡的一种额外的评估技术,用于测量预提取特征的表示质量。与线性探测不同,KNN 探测是非参数的(除了选择 K 值外),它仅基于未见测试样例与已标记训练样例的特征相似度进行分类(例如,在表示空间中相似的样例也应该在视觉上相似并共享相同的类标签)。我们使用 Scikit-Learn 中的 KNN 实现,设置 K=20 并使用欧几里得距离作为距离度量,这是观察到的其他自监督作品中此评估设置的稳定性。对于所有 ROI 任务,我们大致按病例和标签将数据集分层为训练 - 测试折叠,或使用官方折叠(如果可用)。
对于所有任务,我们将图像大小调整为 224×224 像素(如果可用,调整为 448×448 像素)并使用 ImageNet 平均值和标准差参数进行归一化。此外,我们注意到许多 ROI 数据集包含高分辨率图像,将图像大小调整为固定的 224×224 像素或 448×448 像素也会改变图像放大倍数和每像素微米数(mpp)。例如,在 UniToPatho 的 CRC 息肉分类任务中,将原始图像分辨率为 1812×1812 像素、0.45 mpp 的 ROI 调整为 224×224 像素将改变放大倍数为 3.6 mpp。对于 CRC 息肉分类以及 BACH 中的 BRCA 亚型分析,我们使用调整后的图像分辨率{2242 像素, 4482 像素, 8962 像素, 17922 像素}和{2242 像素, 4482 像素, 8962 像素, 13442 像素}进行评估,由于 CTransPath 的限制选择了 224 的倍数。为了从高分辨率图像中预提取特征,对于如 UNI 中的普通 ViT-Large 架构和 CTransPath 中的分层 Swin transformer-T 架构的 ViT,这些架构的前向传递没有修改,而且进行了位置嵌入的插值,使其与 ROI 中的补丁标记具有相同的序列长度。例如,在我们 UNI 中 ViT-Large 架构的补丁嵌入层中,16×16 的补丁标记大小下,一个 224×224 像素的图像将通过 2D 卷积层(核大小和步长为 16,三个来自 RGB 输入图像的输入通道和作为特征嵌入长度的超参数设置的 D 维输出通道)转换成 [14×14×D]- 维的 2D 补丁嵌入网格,然后进行展平和转置(现在是 [196×D]- 维的补丁嵌入序列),这可以用于变换器注意力(称为“打补丁”)。对于 CRC 息肉分类中 1792×1792 像素的图像,使用相同的补丁嵌入层打补丁将产生 [112×112×D]→[12,544×D]- 维的补丁嵌入序列。尽管在变换器注意力的前向传递中输入此序列在计算上昂贵,但通过像 FlashAttention 或 MemEffAttention 这样的内存高效实现仍然是可行的。对于位置嵌入的插值,我们使用了 DINO 中提供的实现。对于多头自注意力(MHSA)的可视化,我们使用 HIPT 代码库提供的笔记本实现来可视化最后一个注意力层的权重,我们注意到这仅适用于普通的 VIT 架构。
ROI 检索¶
为了评估不同编码器产生的嵌入对基于内容的组织病理学图像检索的质量,我们使用 ROI 级别分类数据集,目标是检索与给定查询图像相似的图像(即,具有相同类标签的图像)。在每个基准测试中,我们首先使用预训练的编码器将所有图像嵌入到低维特征表示中。我们将测试集中的每个图像视为一个查询。每个查询图像与作为候选库(键)的 ROI 级别分类训练集中的每个图像进行比较。注意,这些实验中不进行监督学习,类标签仅用于评估目的(即,评估检索图像是否与查询图像共享相同的类标签)。我们首先通过从每个嵌入中减去其欧几里得质心并对每个键进行ℓ2 规范化到单位长度来居中键数据库。对于每个新查询,我们应用相同的移位和规范化步骤,然后通过ℓ2 距离度量将其与数据库中的每个键进行比较,其中较低的距离被解释为较高的相似性。检索到的图像按其相似性得分排序,其对应的类标签用于使用 Acc@K(K∈1, 3, 5)和 MVAcc@5 评估给定检索的成功情况,这些在评估度量中有描述。
ROI 级别细胞类型分割¶
对于 ROI 级别细胞类型分割任务的训练和评估,我们遵循先前的工作,使用 Mask2Former,这是一个通用框架,常用于评估预训练视觉编码器的现成性能。对于非分层的 ViT 架构,我们还使用 ViT-Adapter 框架与 Mask2Former 头部结合使用。对于 ViT-Adapter 和 Mask2Former,我们使用了与 ADE20k 语义分割相同的超参数。具体来说,我们使用 AdamW 优化器,配合阶梯学习率计划。初始学习率设置为 0.0001,应用了 0.05 的权重衰减。为了特别调整主干的学习率,我们应用了 0.1 的学习率乘数。此外,我们在总训练步数的 0.9 和 0.95 分数处将学习率降低 10 倍。对于所有主干,我们对整个模型进行了 50 个周期的微调,批量大小为 16。每 5 个周期评估一次模型在验证集上的性能,并保存基于验证性能的最优模型以供测试。为了增强数据,我们使用了大规模抖动(LSJ)增强,随机比例从 0.5 到 2.0 中抽样,然后固定尺寸裁剪为 896×896 像素以适应 CTransPath 的尺寸限制。在推理时,我们将图像尺寸调整为 224 的最接近倍数。
少样本 ROI 分类和原型学习¶
对于少样本分类,我们遵循先前的工作,使用 SimpleShot 框架评估自监督模型的原型表示的少样本学习性能。原型学习是少样本学习社区中的一个长期任务,也以多种相关形式在 CPath 中提出。与基于元学习的传统少样本学习者不同,SimpleShot 及相关工作表明,结合特定变换和简单分类器的强大特征表示可以达到少样本任务的最先进性能。SimpleShot 类似于最近邻分类,其中训练集(在少样本学习文献中称为“支持”)从 C 类(“方式”)中抽取 K 个样本(“次数”),用于预测测试集中的未见图像(“查询”)。SimpleShot 使用基于 ProtoNet 的最近质心方法,其中每个类的平均特征向量(质心)用作通过距离相似性标记查询集的原型“一次性”示例。如前所述,这些平均特征向量也可以被视为“类原型”,一组一次性代表性示例,独特地代表诸如类标签的语义信息(例如,LUAD 与 LUSC 的形态学)。鉴于 SimpleShot 是少样本学习社区中一个简单且出人意料的强大基线,并且在评估自监督模型时已被普及,我们采用这一基线来评估 UNI 及其在少样本 ROI 分类任务中的比较。我们遵循 SimpleShot 中的建议,在计算类原型前对支持集进行居中(减去在支持集上计算的平均值)和ℓ2 规范化,查询集也进行相同的变换(也使用支持集的平均值进行居中),然后进行最近质心分类。
传统的自然图像分类任务中的少样本学习者通过从训练集中抽取 10,000 个 C 方式,K 次的情节,并将每类的 15 张查询图像作为测试集进行评估。为了与线性和 KNN 探测的度量标准进行等效比较,我们改为抽取 1,000 个 C 方式,K 次的情节,但在每个情节中使用测试集中的所有图像。由于 ROI 任务中可用的训练样例数量相对于幻灯片任务较多,我们改变了每个类的标记样例数量,K∈{1, 2, 4, 8, 16, 32, …256}或给定类的最大标记样例数量。为了与使用所有训练样例的线性和 KNN 探测进行比较,我们还通过对每个类的所有训练样例取平均来评估 SimpleShot,我们将这称为“1-NN”,在补充表 40-60 中标注。
评估幻灯片分类的多示例 SimpleShot(MI-SimpleShot)¶
为了评估作为幻灯片分类任务中类原型的提取表示的质量,我们将 SimpleShot 中的类原型作为“提示”(类似于零样本分类中使用的文本提示),我们称之为 MI-SimpleShot。正如正文中所描述的,我们使用两个幻灯片级数据集(NSCLC 和 RCC 亚型数据集),这些数据集具有与支持集匹配的 ROI 训练示例。简而言之,我们使用 TCGA 统一肿瘤数据集中标注的 LUAD 和 LUSC ROI 进行 NSCLC 亚型分类,以及 TCGA 统一肿瘤数据集中标注的 CCRCC、乳头状肾细胞癌(PRCC)和嫌色性肾细胞癌(CHRCC)ROI 进行 RCC 亚型分类。TCGA 统一肿瘤数据集(在方法部分进一步描述)包含 32 种癌症类型的 271,170 个 256×256 像素 ROI,大约 0.5 mpp,从 8,736 个 H&E FFPE 诊断组织病理学 WSI 中标注和提取。我们注意到,TCGA-LUAD、-LUSC、-CCRCC、-PRCC 和 -CHRCC 队列中每张幻灯片的标注 ROI 数量从 10 到 70 个不等。对于每个类别,我们首先使用预训练的编码器将支持集中的 ROI 嵌入到低维特征表示中,然后对该类中的所有 ROI 特征进行平均池化。平均池化后的特征表示被视为类原型,用作通过归一化欧氏距离相似度为查询集中每张幻灯片的顶部 K 个 ROI 标记的提示。然后通过顶部 K 个 ROI 预测的多数投票进行幻灯片级别的预测。对于每个基准,我们评估 MI-SimpleShot 的顶部 5 个平均池化和顶部 50 个平均池化,并在{1, 2, 4, 8, 16, 32}张训练幻灯片每类的情况下进行评估,类似于我们使用与训练 ABMIL 模型相同的五个折叠进行的少样本幻灯片分类评估,原型从同一训练幻灯片中的标注 ROI 创建。我们注意到在考虑每个类原型的顶部 5 和顶部 50 个补丁的平均分数时性能变化不大。为了与使用所有带 ROI 注释的训练幻灯片的性能进行比较,我们还通过对每个类的所有训练 ROI 特征表示进行平均来评估 MI-SimpleShot,详细结果在补充表 70 和 71 中报告。为了创建相似性热图,我们可视化了与地面真实类原型相比幻灯片中所有补丁的归一化欧氏距离。
评估指标¶
我们报告分类任务的平衡精度、加权 F1 分数和 AUROC。平衡精度是通过取每个类别的召回率的非加权平均来计算的,这考虑了评估集中的类别不平衡。加权 F1 分数是通过对每个类别的 F1 分数(精确率和召回率的调和平均数)进行加权平均计算的,权重由其各自的支持集大小决定。AUROC 是接收者操作特征曲线下的面积,该曲线在变化分类阈值时绘制真阳性率对假阳性率。此外,我们计算二次加权 Cohen’s κ(两组标签之间的注释者协议,例如,真实情况和预测),我们为 ISUP 分级(PANDA)执行此操作,并为 OT-43 和 OT-108 计算顶部 K 精度,其中 K∈{1, 3, 5}(对于给定的测试样本,如果真实标签位于预测的顶部 K 标签中,则该样本被正确得分)。对于检索,我们考虑 Acc@K,其中 K∈{1, 3, 5},这代表检索与查询具有相同类标签的图像的标准顶部 K 精度得分。具体来说,如果在检索到的顶部 K 个图像中至少有一个具有与查询相同的类标签,则认为检索成功。我们还报告 MVAcc@5,与 Acc@5 相比,这更严格地要求顶部 5 个检索到的图像的多数投票与查询在同一类别中,以便将检索视为成功。对于分割,我们报告 Dice 得分(与 F1 得分定义相同)、精确度和召回率,对所有图像和类别进行宏观平均。
统计分析¶
对于所有半监督和全监督实验,我们使用 1000 个自举重复样本进行非参数自举,估计模型性能的 95% 置信区间。对于统计显著性,我们使用 1000 次置换的双侧配对置换测试来评估两个模型性能的观察差异。对于所有少样本设置,我们报告使用箱形图显示模型性能的四分位数值(n=5 次运行),并将须线延伸到四分位距的 1.5 倍数据点。对于 ROI 级别的少样本分类,对于每个 C 方式,K 次设置,我们随机从 C 类中抽取 K 个训练样例,并在整个测试集上评估 1000 次重复实验(称为“情节”或“运行”)。对于幻灯片级别的少样本分类,我们遵循上述相同的设置,但由于罕见疾病类别中支持大小较小,运行次数限制为 5 次。
任务、数据集和与排行榜的比较¶
在这一部分中,我们概述了数据预处理、每个类别的样本数量、训练 - 验证 - 测试折叠等细节(这可能涉及多个任务)。我们还尽可能地在现有排行榜和其他研究的基准中添加我们结果的上下文和比较,并注意由于超参数、划分和预提取特征的差异(许多现有的基准可能不使用针对病理学的预训练编码器),比较可能并不总是等价的。在与排行榜和比较中,我们采用公共评估中使用的度量标准,在表标题中进一步阐述。
基于内部 BWH 数据的 OncoTree 癌症分类(43 种癌症类型,108 个 OncoTree 代码)¶
正如正文中所述,OncoTree 癌症分类是 CPath 中的一个大规模分层分类任务,遵循 OncoTree(OT)癌症分类系统。这项任务旨在评估预训练模型在分类多种疾病类别和组织类型方面的泛化能力。使用内部 BWH 幻灯片,我们定义了一个包含 43 种癌症类型的 5564 个 WSI 的数据集,这些癌症类型进一步细分为 108 个 OncoTree 代码,每个 OncoTree 代码至少有 20 个 WSI。由于 OT-108 中几个 OncoTree 代码的支持大小较小,所有 ABMIL 模型都使用训练 - 测试折叠进行训练,没有提前停止。对于训练和评估,我们大致将数据集按标签分层为 71:29 的训练 - 测试折叠(比例为 3944:1620 张幻灯片),对 OT-43 和 OT-108 使用相同的折叠,测试集中每个 OncoTree 代码使用 15 张幻灯片,训练集中每个 OncoTree 代码至少使用 5 张幻灯片。粗细粒度任务的分层分类在补充表 4 中报告。除了膀胱移行细胞癌(BLCA)、浸润性导管癌(IDC)、浸润性小叶癌(ILC)、结肠腺癌(COAD)、直肠腺癌(READ)、子宫内膜样癌(UEC)、胃腺癌(STAD)、头颈部鳞状细胞癌(HNSC)、未特指的弥漫大 B 细胞淋巴瘤(DLBCLNOS)、黑色素瘤(MEL)、LUAD、LUSC、胰腺腺癌(PAAD)、PRAD、皮肤鳞状细胞癌(CSCC)、小细胞肺癌(SCLC)、胃食管交界处腺癌(GEJ)和慢性淋巴细胞性白血病/小淋巴细胞性淋巴瘤(CLLSLL)等癌症类型外,此任务中的癌症类型是由 RARECARE 项目和国家癌症研究所的监测、流行病学和最终结果(NCI-SEER)计划指定的罕见癌症。我们注意到,OT-43 和 OT-108 的训练折叠中的幻灯片包含在 OP-1K 和 OP-22K 预训练中,测试集从这些预训练来源中保留(遵循 ImageNet 的做法)。
由于在所有预训练模型(包括中间检查点)中反复提取所有非重叠组织补丁的特征存在存储限制,我们为每个 WSI 抽取了 200 个代表性补丁进行特征提取。为了选择这些补丁,我们首先提取了 ResNet-50IN 特征,然后进行了聚类,这在 WSISA、DeepAttnMISL 和其他工作中曾经使用过。我们注意到这些工作受到视觉词袋(vBOW)的启发,已经适应病理学,通过应用于深度特征的聚类将高分辨率 ROI 和 WSI 公式化为较小但代表性的组织补丁集合,其下游应用包括 MIL 和检索。对于所有预训练编码器,我们从同一采样集合的补丁中提取特征。尽管采取了额外的计算步骤来导出这些采样补丁,我们注意到这不属于归纳推理,因为测试集的所有 WSI 样本从未对任何学习组件(聚类按 WSI 拟合,' 样本 ' 在幻灯片级别而不是补丁级别定义)可见。为了验证这种方法的性能与使用 WSI 的所有组织补丁的特征相当,我们比较了使用采样特征与 UNI、CTransPath、REMEDIS 和 ResNet-50IN 的全部特征的性能,我们也在补充表 12 和 15 中报告。我们不仅观察到使用采样特征时性能略有下降(顶部 1 准确率最大下降 0.9%,AUROC 下降 0.007),而且对于许多模型观察到性能提高。对于 REMEDIS,我们观察到当使用全部特征时,ABMIL 模型的性能崩溃,OT-43 和 OT-108 的顶部 1 准确率分别为 4.0% 和 11.8%(与使用采样特征的 59.3% 和 41.2% 相比)。我们假设这些性能提高是由于 OT-43 和 OT-108 的困难性质,补丁采样减少了 ABMIL 的输入数据复杂性(例如,不是在 10000+ 补丁的包中找到诊断相关特征,而只考虑 200 个代表性补丁)。
乳腺转移检测基于 CAMELYON16(2 类)¶
乳腺转移检测任务源自 2016 年的癌症转移淋巴结挑战(CAMELYON16),包含拉德堡大学医学中心和乌特勒支大学医学中心的 400 张 H&E 染色的 FFPE 组织病理学 WSI,用于转移检测。我们从测试集中移除了一张标记错误的幻灯片,结果为 399 张幻灯片(239 张正常,160 张转移)。我们使用官方的训练 - 测试折叠进行训练和评估,并将训练集分层为 90:10 的训练 - 验证,结果为 61:7:32 的训练 - 验证 - 测试折叠(243:27:129 张幻灯片)。除了内部比较外,我们还将我们的结果与挑战时的排行榜进行比较,提供最近同行评审文献中报告的最佳性能模型的时间线,并在补充表 36 中增加与最先进方法的比较。我们注意到,与 UNI 的比较可能不等同,许多提出的方法使用了 ResNet-50IN 特征,并且还使用了更复杂的 MIL 架构。
基于 TCGA 和 CPTAC 的 NSCLC 亚型分类(LUAD 与 LUSC,2 类)¶
NSCLC 亚型分类任务包括来自 TCGA 和 CPTAC 的 NSCLC H&E 染色的 FFPE 诊断组织病理学 WSI,用于分类两个亚型:原发性 LUAD 和 LUSC 病例。为了质量控制,在 TCGA 中,我们排除了具有缺失或错误元数据的幻灯片,共计 1041 张幻灯片(529 张 LUAD 和 512 张 LUSC)。在 CPTAC 中,我们排除了冷冻组织、非肿瘤组织的幻灯片或未标记为有可接受的肿瘤段的幻灯片,共计 1091 张幻灯片(578 张 LUAD 和 513 张 LUSC)。我们对 TCGA-NSCLC 队列进行标签分层,按 80:10:10 的训练 - 验证 - 测试折叠(848:97:98 张幻灯片),并使用保留的 CPTAC 队列进行外部评估。
基于 DHMC 的 RCC 亚型分类(CCRCC 与 PRCC 与 CHRCC 与 ROCY 与良性,5 类)¶
RCC 亚型分类任务包括 563 张来自达特茅斯 - 希区柯克医疗中心(DHMC)的 RCC H&E 染色的 FFPE 诊断组织病理学 WSI(485 张切除术和 78 张活检),用于分类五个亚型:原发性 CCRCC(344 张幻灯片),PRCC(101 张幻灯片)和 CHRCC(23 张幻灯片),肾上皮瘤(ROCY,66 张幻灯片)和良性病例(29 张幻灯片)。对于这两项任务的训练和评估,我们使用了修改的训练 - 验证 - 测试折叠配置,比例为 70:4:26(393:23:147 张幻灯片),由于训练折叠中缺少 CHRCC 病例,我们将 8 个 CHRCC 病例从测试折叠移至训练折叠。
基于 TCGA、DHMC 和 CPTAC 的 RCC 亚型分类(CCRCC 与 PRCC 与 CHRCC,3 类)¶
RCC 亚型分类任务包括 1794 张来自 TCGA、DHMC 和 CPTAC 的 RCC H&E 染色的
FFPE 诊断组织病理学 WSI,用于分类三个亚型:原发性 CCRCC、PRCC 和 CHRCC。为了质量控制,在 TCGA 中,我们排除了缺少低分辨率下采样的幻灯片,共计 922 张幻灯片(519 张 CCRCC,294 张 PRCC 和 109 张 CHRCC)。在 DHMC 集合中,我们过滤掉之前描述的 DHMC-Kidney 队列中的肾上皮瘤,共计 468 张幻灯片(344 张 CCRCC,101 张 PRCC 和 23 张 CHRCC)。在 CPTAC 中,我们排除了冷冻组织、非肿瘤组织的幻灯片或未标记为有可接受的肿瘤段的幻灯片,共计 404 张幻灯片(404 张 CCRCC)。对于训练和评估,我们对 TCGA-NSCLC 队列进行标签分层,按 80:10:10 的训练 - 验证 - 测试折叠(736:89:97 张幻灯片),并在保留的 DHMC 和 CPTAC 队列上进行外部评估。由于 CPTAC 仅包括 CCRCC 病例,我们将 DHMC 和 CPTAC 合并为一个评估队列。
基于 HunCRC 的 CRC 筛查(4 类)¶
CRC 筛查任务包括 200 张来自匈牙利结直肠癌筛查(HunCRC)项目的 Semmelweis University 的 H&E 染色的 FFPE 诊断组织病理学 WSI,这是结直肠活检的数据集。在此数据集中,我们定义了一个 4 类的粗分类亚型任务,类别为阴性(10 张幻灯片)、非肿瘤性病变(38 张幻灯片)、CRC(46 张幻灯片)和腺瘤(106 张幻灯片),其中地面真实标签由研究的病理学家设置。对于这项任务的训练和评估,我们将 HunCRC 幻灯片数据集按标签分层为 50:25:25 的训练 - 验证 - 测试折叠(158:21:21 张幻灯片)。
基于 BRACS 的 BRCA 粗分类和细分类亚型(3 类和 7 类)¶
BRCA 粗分类和细分类亚型任务包括来自 IRCCS Fondazione Pascale、国家研究委员会(CNR)高性能计算和网络研究所(ICAR)和 IBM 研究 - 苏黎世的 547 张乳腺癌 H&E 幻灯片。在此数据集中,我们定义了一个 3 类的粗分类亚型任务,使用“良性肿瘤”、“非典型肿瘤”和“恶性肿瘤”的标签。此外,我们定义了一个 7 类的细分类亚型任务,将良性肿瘤亚型为“正常”、“病理性良性”和“常见导管增生”,将非典型肿瘤亚型为“平坦上皮异型”和“非典型导管增生”,将恶性肿瘤亚型为“原位导管癌”和“浸润性癌”。粗分类和细分类任务的分层分类在补充表 19 中报告。对于这两项任务的训练和评估,我们使用官方训练 - 验证 - 测试折叠,比例为 72:12:16(395:65:87 张幻灯片),两种粗分类和细分类任务使用相同的折叠。
基于 TCGA 和 EBRAINS 的胶质瘤 IDH1 突变预测和组织分子¶
亚型分类(2 类和 5 类)胶质瘤 IDH1 突变预测和组织分子亚型分类任务包括来自 TCGA 和 EBRAINS 数字肿瘤图谱的 1996 张胶质瘤、星形细胞瘤和少突细胞瘤的 H&E 染色的 FFPE 诊断组织病理学 WSI,这些病例具有分子状态。我们首先定义了一个 5 类的胶质瘤组织分子亚型分类任务,其标签为:IDH1 突变星形细胞瘤(257 张幻灯片),IDH1 突变胶质母细胞瘤(93 张幻灯片),IDH1 突变且 1p/19q 缺失的少突细胞瘤(408 张幻灯片),IDH1 野生型胶质母细胞瘤(1094 张幻灯片)和 IDH1 野生型星形细胞瘤(144 张幻灯片)。此外,我们定义了一个更简单的 2 类任务,仅预测 IDH1 状态:IDH1 野生型(1238 张幻灯片)和 IDH1 突变(756 张幻灯片)。所有这些脑肿瘤任务中的肿瘤都被 RARECARE 项目和 NCI-SEER 计划指定为罕见癌症。粗分类和细分类任务的分层分类在补充表 21 中报告。对于这两项任务的训练和评估,我们大致将 TCGA-GBMLGG(TCGA 胶质母细胞瘤低级别胶质瘤)数据集分层为 47:22:31 的训练 - 验证 - 测试折叠(525:243:355 张幻灯片),并使用保留的 EBRAINS 队列进行外部评估(873 张幻灯片),两种粗分类和细分类任务使用相同的折叠。
基于 EBRAINS 的脑肿瘤粗分类和细分类亚型(12 类和 30 类)¶
脑肿瘤粗分类和细分类亚型任务包括来自维也纳大学的 EBRAINS 数字肿瘤图谱的 2319 张脑肿瘤 H&E 染色的 FFPE 诊断组织病理学 WSI。原始数据集大小为 3114 张幻灯片,我们定义了一个 30 类的细分类脑肿瘤亚型任务,仅限于具有至少 30 张幻灯片的诊断标签:IDH1 野生型胶质母细胞瘤(474 张幻灯片),毛细胞性星形细胞瘤(173 张幻灯片),硬膜下脑膜瘤(104 张幻灯片),垂体腺瘤(99 张幻灯片),IDH1 突变且 1p/19q 缺失的间变性少突细胞瘤(91 张幻灯片),神经胶质瘤(88 张幻灯片),血管母细胞瘤(88 张幻灯片),粘液性胶质瘤(85 张幻灯片),IDH1 突变且 1p/19q 缺失的少突细胞瘤(85 张幻灯片),非典型脑膜瘤(83 张幻灯片),神经鞘瘤(81 张幻灯片),IDH1 突变的弥散性星形细胞瘤(70 张幻灯片),过渡性脑膜瘤(68 张幻灯片),中枢神经系统(CNS)的弥散大 B 细胞淋巴瘤(59 张幻灯片),胶质肉瘤(59 张幻灯片),纤维性脑膜瘤(57 张幻灯片),间变性室管膜瘤(50 张幻灯片),IDH1 野生型间变性星形细胞瘤(47 张幻灯片),转移性肿瘤(47 张幻灯片),DH1 突变间变性星形细胞瘤(47 张幻灯片),室管膜瘤(46 张幻灯片),间变性脑膜瘤(46 张幻灯片),分泌性脑膜瘤(41 张幻灯片),脂肪瘤(38 张幻灯片),血管周细胞瘤(34 张幻灯片),IDH1 突变胶质母细胞瘤(34 张幻灯片),非 WNT/非 SHH 髓母细胞瘤(32 张幻灯片),朗格汉斯细胞组织细胞病(32 张幻灯片),血管瘤性脑膜瘤(31 张幻灯片)和血管瘤(30 张幻灯片)。从同一 2319 张幻灯片的细分类任务中,我们还定义了一个 12 类的粗分类脑肿瘤亚型任务,将上述标签分为以下类别:成人型弥散性胶质瘤(837 张幻灯片),脑膜瘤(430 张幻灯片),涉及中枢神经系统的间质性、非脑膜性肿瘤(190 张幻灯片),垂体区域肿瘤(184 张幻灯片),局限性星形胶质瘤(173 张幻灯片),室管膜肿瘤(96 张幻灯片),涉及中枢神经系统的血淋巴肿瘤(91 张幻灯片),神经胶质和神经瘤(88 张幻灯片),颅神经和脊神经肿瘤(81 张幻灯片),儿童型弥散性低级别胶质瘤(70 张幻灯片),转移性肿瘤(47 张幻灯片)和胚胎性肿瘤(32 张幻灯片)。这些脑肿瘤任务中的肿瘤都被 RARECARE 项目和 NCI-SEER 计划指定为罕见癌症。粗分类和细分类任务的分层分类在补充表 20 中报告。对于这两项任务的训练和评估,我们大致将数据集分层为 50:25:25 的训练 - 验证 - 测试折叠(1151:595:573 张幻灯片),两种粗分类和细分类任务使用相同的折叠。
前列腺 ISUP 分级基于 PANDA (6 类)¶
这个 ISUP 分级任务来源于 PANDA 挑战赛,涵盖了来自拉德堡大学医学中心和卡罗林斯卡学院的 10,616 例前列腺癌核心针吸活检样本。每个幻灯片被分配一个 ISUP 分数,定义前列腺癌等级(6 类分级任务)。为了质量控制,我们按照之前的工作排除了错误标注或标签嘈杂的幻灯片,最终得到 9,555 张幻灯片(2,603 G0,2,399 G1,1,209 G2,1,118 G3,1,124 G4,1,102 G5)。在训练和评估中,我们将 PANDA 按照标签分层划分为 80:10:10 的训练 - 验证 - 测试折叠(7,647:954:954 幻灯片)。除了内部比较外,我们还使用公共 MIL 基线的相同分割重新评估我们的结果,并采用 WholeSIGHT 的评估策略分别评估 Karolinska 和 Radboud 队列。补充表 30 报告了 UNI 与公共分割的内部比较的性能,补充表 37 报告了我们与公共 MIL 基线的结果。我们也注意到由于使用了 ResNet-50IN 特征,与公共 MIL 性能的比较可能不等价,但请注意这些基线也采用了更复杂的 MIL 架构。
内部 BWH 数据的心内膜评估(2 类)¶
BWH-EMB 数据集包含了 BWH 收集的 1,688 例内部心内膜活检(EMBs)的 5,021 张 H&E FFPE 组织病理学 WSI,用于细胞介导的移植排斥反应(ACR)(2,444 ACR,2,577 其他)。在训练和评估中,我们按病例和标签分层将数据集分为训练 - 验证 - 测试折叠(3,547:484:900 幻灯片,1,192:164:332 病人),评估在病人层面进行。除了内部比较外,我们还将我们的结果与 CRANE 的结果进行比较(使用相同的分割)。我们也注意到由于 CRANE 使用了 ResNet-50IN 特征,与 UNI 的比较可能不等价,但请注意这个基线也使用了多任务学习来评估 EMB。
基于 CRC-100K 的 CRC 组织分类(9 类)¶
CRC 组织分类任务基于 CRC-100K 数据集,该数据集包含来自国家肿瘤病中心(NCT)生物库和曼海姆大学医学中心(UMM)病理档案库的 136 例结直肠腺癌样本的 107,180 个 224×224 像素的 ROI,已标注以下 9 类:脂肪(11,745 个 ROI),背景(11,413 个 ROI),碎片(11,851 个 ROI),淋巴细胞(12,191 个 ROI),黏液(9,931 个 ROI),平滑肌(14,128 个 ROI),正常结肠粘膜(9,504 个 ROI),与癌相关的间质(10,867 个 ROI)和结直肠腺癌上皮(15,550 个 ROI)。在训练和评估中,我们使用官方按病例分层的训练 - 测试折叠(100,000:7,180 个 ROI),训练折叠由 NCT 生物库和 UMM 病理档案库的 100,000 个 ROI(86 个 WSI)组成,称为“NCT-CRC-HE-100K”,测试折叠由 NCT 生物库的 7,180 个 ROI(50 个 WSI)组成,称为“CRC-VAL-HE-7K”。此外,我们使用的是未经染色归一化的 NCT-CRC-HE-100K 版本。我们在 0.5 mpp 的 224×224 像素 ROI 上评估此数据集。
基于 CAMELYON17-WILDS 的乳腺转移检测(2 类)¶
乳腺转移检测任务基于 CAMELYON17 数据集的补丁版本(称为 PatchCAMELYON 或‘PCAM’),由 WILDS 创建的折叠用于测试模型在分布偏移下的鲁棒性。该数据集包含 417,894 个 96×96 像素的组织病理学 ROI,大约在 0.92-1.00 mpp,从 CAMELYON17 挑战赛获得的乳腺癌转移淋巴结切片的 WSI 中提取。ROI 标签指示补丁是否包含肿瘤。在训练和评估中,我们使用 WILDS 提供的官方训练 - 验证 - 测试折叠。训练集包含来自三家医院的 302,436 个补丁,模型在两个外部分布(OD)数据集上进行评估,这两个数据集包含来自另外两家医院的 34,904 个补丁(ValOD)和 80,554 个补丁(TestOD)。我们将所有图像双线性上采样至 224×224 像素以与 CTransPath 进行等效比较。除了内部比较外,我们还将我们的结果与 WILDS 基准的公共排行榜进行比较,我们在补充表 62 中报告了这些结果。域内验证折叠未与训练集合并,也未用于超参数调整。我们注意到,与我们的评估相比,许多方法是端到端的微调,采用从自然图像(而非病理学)转移学习的方法,因此比较可能不等价。
基于 HunCRC 的 CRC 组织分类(9 类)¶
CRC 组织分类任务基于 HunCRC 数据集,该数据集包含来自同一 200 个 H&E FFPE 诊断组织病理学 WSI 的 101,398 个 512×512 像素的 ROI,这些 WSI 也在幻灯片级任务中描述过。ROI 被标记为以下九类:腺癌(4,315 个 ROI),高级别异型增生(2,281 个 ROI),低级别异型增生(55,787 个 ROI),炎症(763 个 ROI),肿瘤坏死(365 个 ROI),可疑侵袭(570 个 ROI),切缘(534 个 ROI),技术性伪影(3,470 个 ROI),正常(31,323 个 ROI)。在训练和评估中,我们按病例和标签大致分层将数据集分为训练 - 测试折叠(151:49 例,76,753:22,655 个 ROI),用于线性探针,KNN 和 SimpleShot 评估。我们在 0.55 mpp 的 448×448 像素 ROI 上评估此数据集。
基于 BACH 的 BRCA 亚型分类(4 类)¶
BRCA 亚型分类任务基于 Breast Carcinoma Subtyping (BACH) 数据集,该数据集包含 400 个 2,048×1,536 像素的 ROI,这些 ROI 在 0.42 mpp,从 ICIAR 2018 乳腺癌组织学图像分析挑战赛(BACH)的 H&E FFPE 诊断组织病理学 WSI 中提取。ROI 被标记为以下四类:正常(100 个 ROI),良性(100 个 ROI),原位癌(100 个 ROI)和浸润性癌(100 个 ROI)。在训练和评估中,我们按标签分层将数据集分为训练 - 测试折叠(320:80 个 ROI),用于线性探针,KNN 和 SimpleShot 评估。此外,我们在以下中心裁剪和调整大小的图像分辨率上评估此数据集:224×224 像素在 2.88 mpp,448×448 像素在 1.44 mpp,896×896 像素在 0.72 mpp 和 1,344×1,344 像素在 0.48 mpp。
基于 TCGA 和 HEL 的 CCRCC 组织分类(3 类)¶
CCRCC 组织分类任务包括 52,713 个 256×256 像素和 300×300 像素的 ROI,这些 ROI 在大约 0.25 mpp,从 TCGA(502 个样本)和赫尔辛基大学医院(HEL)(64 个样本)的 H&E FFPE 诊断组织病理学 WSI 中提取。ROI 被标记为以下六类:癌症(13,057 个 ROI),正常(8,652 个 ROI),间质(5,460 个 ROI),红细胞(996 个 ROI),空背景(16,026 个 ROI)和其他纹理(8,522 个 ROI)。对于这项任务,我们只考虑癌症、正常和间质标签,因为在数据来源分层和“其他”类别的歧义时标签不平衡。我们使用 TCGA 的 21,095 个 ROI 和 HEL 的 6,074 个 ROI 分别作为训练和测试队列(训练 - 测试折叠比为 21,095:6,074),用于线性探针,KNN 和 SimpleShot 评估。我们在大约 0.29 mpp 的 224×224 像素 ROI 上评估此数据集。
基于 AGGC 的 PRAD 组织分类(5 类)¶
PRAD 组织分类任务基于 2022 年自动化格里森分级挑战赛(AGGC),该挑战赛由新加坡国立大学医院和科技研究局(A*STAR)举办 101。它包括来自前列腺切除术(105 个训练,45 个测试)和活检(37 个训练,16 个测试)的 203 个 WSI,使用 Akoya Biosciences 扫描仪在 0.5 mpp 的×20 放大倍数下数字化。每张幻灯片包括部分像素级注释,标出不同的格里森模式和间质区域。从原始 WSI 和注释中,我们建立了一个包括 1,125,640 个不重叠的 256×256 像素 ROI 的数据集(训练 - 测试折叠比为 780,619:345,021),用于线性探针,KNN 和 SimpleShot 评估。具有多个格里森模式的 ROI 被分配给最具侵略性的等级。我们在大约 0.57 mpp 的 224×224 像素 ROI 上评估此数据集。
基于 UKK、WNS、TCGA 和 CHA 的 ESCA 组织分类(11 类)¶
ESCA(食道癌)组织分类任务包括来自科隆大学医院(UKK,22 张幻灯片),维也纳新城地区医院(WNS,62 张幻灯片),TCGA(22 张幻灯片)和柏林查理特大学医院(CHA,214 张幻灯片)的 320 张 H&E FFPE 诊断组织病理学 WSI 的 367,229 个 256×256 像素的 ROI,分辨率为 0.78 mpp。ROI 被标记为以下 11 类:外膜(71,131 个 ROI),粘膜固有层(2,173 个 ROI),粘膜肌层(2,951 个 ROI),固有肌层(83,358 个 ROI),退行性组织(56,490 个 ROI),胃粘膜(44,416 个 ROI),食道粘膜(18,561 个 ROI),粘膜下层(22,117 个 ROI),粘膜下腺体(1,516 个 ROI),肿瘤(63,863 个 ROI)和溃疡(753 个 ROI)。在训练和评估中,我们将 UKK、WNS 和 TCGA 合并为一个训练队列(189,142 个 ROI),并将 CHA 作为测试队列(178,187 个 ROI),训练 - 测试折叠比为 51:49,然后用于线性探针,KNN 和 SimpleShot 评估。我们在大约 0.89 mpp 的 224×224 像素 ROI 上评估此数据集。
基于 UniToPatho 的大肠息肉分类(6 类)¶
大肠息肉分类任务基于 UniToPatho 数据集,该数据集包含来自都灵大学的 292 个 H&E FFPE 诊断组织病理学 WSI 中提取的 9,536 个 1,812×1,812 像素的 ROI,分辨率为 0.44 mpp。ROI 被标记为以下六类:正常(950 个 ROI)、增生性息肉(545 个 ROI)、管状腺瘤伴高级别非典型增生(454 个 ROI)、管状腺瘤伴低级别非典型增生(3,618 个 ROI)、管状 - 绒毛状腺瘤伴高级别非典型增生(916 个 ROI)和管状 - 绒毛状腺瘤伴低级别非典型增生(2,186 个 ROI)。在训练和评估中,我们使用官方的训练 - 测试折叠(6,270:2,399 个 ROI)。我们在以下调整大小的图像分辨率上评估此数据集:224×224 像素分辨率为 3.60 mpp,448×448 像素分辨率为 1.80 mpp,896×896 像素分辨率为 0.90 mpp,以及 1,792×1,792 像素分辨率为 0.45 mpp。
基于 TCGA CRC-MSI 的大肠微卫星不稳定性(MSI)预测(2 类)¶
大肠微卫星不稳定性(MSI)预测任务基于 TCGA CRC-MSI 数据集,该数据集包含 51,918 个 512×512 像素的 ROI,大约 0.5 mpp,从标记和提取自 TCGA 的大肠腺癌样本的 H&E FFPE 诊断组织病理学 WSI 中获得,并且已使用 Macenko 标准化预处理。ROI 按照样本的患者级标签被标记为以下两类:微卫星不稳定(15,002 个 ROI)和微卫星稳定(36,916 个 ROI)。在训练和评估中,我们使用官方的训练 - 测试折叠(19,557:32,361 个 ROI)进行线性探针,KNN 和 SimpleShot 评估。我们在 448×448 像素分辨率为 0.57 mpp 的 ROI 上评估此数据集。
基于 TCGA 统一肿瘤的泛癌组织分类(32 类)¶
泛癌组织分类任务基于 TCGA 统一肿瘤数据集,该数据集包含 271,170 个 256×256 像素的 ROI,大约 0.5 mpp,从 TCGA 中的 8,736 个 H&E FFPE 诊断组织病理学 WSI 中标记和提取的 32 种癌症类型。图像被标记为以下 32 类:肾上腺皮质癌(ACC)(4,980 个 ROI)、膀胱尿路上皮癌(BLCA)(9,990 个 ROI)、脑低级别胶质瘤(LGG)(23,530 个 ROI)、乳腺癌(BRCA)(23,690 个 ROI)、宫颈鳞状细胞癌和宫颈腺癌(CESC)(6,270 个 ROI)、胆管癌(CHOL)(900 个 ROI)、结直肠腺癌(COAD)(8,150 个 ROI)、食管癌(ESCA)(3,380 个 ROI)、多形性胶质母细胞瘤(GBM)(23,740 个 ROI)、头颈癌(HNSC)(11,790 个 ROI)、肾嫌色细胞瘤(KICH)(2,460 个 ROI)、肾透明细胞癌(KIRC)(11,650 个 ROI)、肾乳头状细胞癌(KIRP)(6,790 个 ROI)、肝细胞癌(LIHC)(8,370 个 ROI)、肺腺癌(LUAD)(16,460 个 ROI)、肺鳞状细胞癌(LUSC)(16,560 个 ROI)、淋巴瘤弥漫大 B 细胞淋巴瘤(DLBC)(840 个 ROI)、间皮瘤(MESO)(2,090 个 ROI)、卵巢浆液性囊腺癌(OV)(2,520 个 ROI)、胰腺癌(PAAD)(4,090 个 ROI)、嗜铬细胞瘤和副神经节瘤(PCPG)(1,350 个 ROI)、前列腺癌(PRAD)(9,810 个 ROI)、直肠腺癌(READ)(1,880 个 ROI)、肉瘤(SARC)(13,480 个 ROI)、皮肤黑色素瘤(SKCM)(10,060 个 ROI)、胃腺癌(STAD)(9,670 个 ROI)、睾丸生殖细胞肿瘤(TGCT)(6,010 个 ROI)、胸腺瘤(THYM)(3,600 个 ROI)、甲状腺癌(THCA)(11,360 个 ROI)、子宫肉瘤(UCS)(2,120 个 ROI)和子宫体内膜癌(UCEC)(12,480 个 ROI)。除了 BLCA、BRCA、COAD、HNSC、LUAD、LUSC、PAAD、PRAD、READ、SKCM、STAD、THCA 和 UCEC 之外,此任务中的所有其他癌症类型都被 RARECARE 项目和 NCI-SEER 计划指定为罕见癌症。在训练和评估中,我们对数据集进行病例分层和大致标签分层,分为训练 - 测试折叠(216,350:55,360 个 ROI),用于线性探针,KNN 和 SimpleShot 评估。我们在大约 0.57 mpp 的 224×224 像素的 ROI 上评估此数据集。为了减轻 TCGA 中特定站点的 H&E 染色变异可能带来的偏见,我们使用 Macenko 标准化方法对所有 ROI 进行了标准化。
肿瘤免疫淋巴细胞(TIL)检测基于 TCGA-TILs(2 类)¶
肿瘤免疫淋巴细胞(TIL)检测任务基于 TCGA-TILs 数据集,该数据集包括 304,097 个 100×100 像素的组织病理学 ROI,分辨率大约为 0.5 mpp,从 TCGA 中的 H&E FFPE 诊断组织病理学 WSI 中提取。这些 ROI 根据图像中是否存在至少两个 TILs 被标记为以下两类:TIL 阳性(54,910 个 ROI)和 TIL 阴性(249,187 个 ROI)。在训练和评估中,我们使用官方的训练 - 验证 - 测试折叠(209,221:38,601:56,275 个 ROI),并将训练和验证折叠合并为一个训练折叠。我们将所有图像双线性上采样到 224×224 像素,分辨率为 0.20 mpp,以便与 CTransPath 进行等价比较。为了减轻 TCGA 中特定站点的 H&E 染色变异可能带来的偏见,我们使用 Macenko 标准化方法对所有 ROI 进行了标准化。除了内部比较外,我们还将我们的结果与 ChampKit 排行榜进行了比较,结果报告在补充表 61 中。我们注意到,与公共结果的比较可能与我们的评估不同,因为许多方法是使用从自然图像(而非病理学)的迁移学习进行端到端微调的。
泛癌细胞类型分割基于 SegPath(8 个细胞类型作为单独任务)¶
细胞类型分割任务来源于 SegPath 数据集,该数据集包括 158,687 个 984×984 像素的 ROI,分辨率为 0.22 mpp,从东京大学医院的癌症组织中的 H&E FFPE 诊断组织病理学 WSI 中提取。免疫荧光和 DAPI 核染色用于 ROI,并用作以下分类的图像掩模:内皮细胞(10,647 个 ROI)、上皮细胞(26,509 个 ROI)、白细胞(24,805 个 ROI)、淋巴细胞(12,273 个 ROI)、髓样细胞(14,135 个 ROI)、浆细胞(13,231 个 ROI)、红细胞(25,909 个 ROI)和平滑肌(31,178 个 ROI)。每种细胞类型在数据集中形成一个独立的组织分割任务,包括组织/细胞区域和非组织/细胞区域两类。在训练和评估中,我们使用官方的训练 - 验证 - 测试分割,大约按 80:10:10 的比例进行。此外,我们使用此数据集的公共评估来比较我们的结果,也在补充表 69 中报告。我们注意到,在官方数据集中没有公开单个模型的性能,因此我们插值了每种细胞类型的最佳性能模型的性能界限。
计算硬件和软件¶
我们使用 Python(v3.8.13)和 PyTorch(v2.0.0, CUDA 11.7)(https://pytorch.org)进行所有实验和分析(除非另有说明),这些实验可以使用下面概述的开源库复现。为了训练 UNI 使用 DINOv2,我们修改了由 Hugging Face 维护的开源 timm 库(v0.9.2)(https://huggingface.co)中的视觉变压器实现作为编码器主干,并使用原始的 DINOv2 自监督学习算法(https://github.com/facebookresearch/dinov2)进行预训练,这使用了 4×8 个 80GB NVIDIA A100 GPU(图形处理单元)节点,配置为多 GPU、多节点训练,使用分布式数据并行(DDP)。所有其他用于下游实验的计算都在单个 24GB NVIDIA 3090 GPU 上进行。所有 WSI 处理由 OpenSlide(v4.3.1),openslide-python(v1.2.0)和 CLAM(https://github.com/mahmoodlab/CLAM)支持。我们使用 Scikit-learn(v1.2.1)的实现进行最近邻分类,并使用 logistic 回归实现和 SimpleShot 实现,这些都由 LGSSL 代码库提供(https://github.com/mbanani/lgssl)。其他视觉预训练编码器的实现可以在以下链接找到:带有 ImageNet Transfer 的 ResNet-50(https://github.com/mahmoodlab/CLAM),CTransPath(https://github.com/Xiyue-Wang/TransPath),和 REMEDIS(https://github.com/google-research/medical-ai-research-foundations)。我们注意到,使用 REMEDIS 需要完成数据使用协议,可在 PhysioNet 网站(https://physionet.org/content/medical-ai-research-foundation)访问和提交。对于多头注意力可视化,我们使用了 HIPT 代码库(https://github.com/mahmoodlab/HIPT)提供的可视化工具。对于训练弱监督 ABMIL 模型,我们适应了 CLAM 代码库的训练脚手架代码(https://github.com/mahmoodlab/CLAM)。对于训练语义分割,我们使用了原始的 Mask2Former 实现(https://github.com/facebookresearch/Mask2Former),这是基于 detectron2(参考文献 174)(v0.6),并需要一些较老的包进行兼容:Python(v3.8)和 PyTorch(v1.9.0, CUDA 11.1)。为了在 UNI 中添加 ViT-Adapter,我们适应了其原始实现(https://github.com/czczup/ViT-Adapter)在 detectron2 中使用 Mask2Former 进行训练。Pillow(v9.3.0)和 OpenCV-python 用于执行基本图像处理任务。Matplotlib(v3.7.1)和 Seaborn(v0.12.2)用于创建图表和图形。使用其他杂项 Python 库的详细信息在报告摘要中详细说明。
报告摘要¶
关于研究设计的更多信息可在本文链接的 Nature Portfolio 报告摘要中找到。